解决细菌和酵母的基因组后,合成生物学家地方重写这些更为复杂的有机体,包括人类。
在典型的实验室条件下,应变JF1细菌大肠杆菌看起来像任何其他发黄,飞溅的殖民地琥珀琼脂板。但是洗澡的殖民地波长的红色、绿色或蓝色光和色素细胞生长介质中的化学物质转换成模式相匹配的彩色光,他们接触,产生一个温和的和模糊的形象,让人想起1970年代的宝丽来。

克里斯托弗•沃伊特的实验室在剑桥的麻省理工学院创造了复杂的基因电路驱动这一转变,2017年5月报道称,他的团队使用了系统重建一个五彩缤纷的几何图解蜥蜴的荷兰画家m·c·埃舍尔1。锻炼只是为了好玩,他说,一种方法来证明合成生物学的最新进展。但是这并不容易:电路包含18个基因和32监管元素,分布在4个小的DNA环状分子称为质粒,和46198个碱基对的DNA。它响应分别为红色、绿色和蓝色的光。“当你把这一切加起来,那是一个相当复杂的项目,“沃伊特说。
这并不是唯一的一个。合成生物学是充斥着类似的项目或更大的复杂性。改进DNA合成和编辑技术带来了降低成本和巨大的精度,帮助生物学家从头构建或重新设计等微生物的基因组大肠杆菌和啤酒酵母(酿酒酵母)。合成生物学的研究人员现在有严重的讨论再造的基因组复杂的有机体,包括人类,尽管大量的障碍阻碍。例如,大型的DNA片段的操纵提供了技术挑战,尽管DNA合成的成本不断下降,成本仍然是禁止当数十亿基地必须重写。
”结果在过去两年已经增加了我的乐观,我们可以做一些很深刻的工程在动物身上,“彼得•卡尔说麻省理工学院林肯实验室合成生物学家在列克星敦,马萨诸塞州。
在2015年的一份信2对《华尔街日报》生物技术的发展趋势卡尔问道:“有合成生物学的音障,还是光速?“问题是修辞,因为显然存在——增长率不变的限制,例如,不能无限快。但什么是声障合成生物学进化,他说。设计上的尖端几年前现在实际的可行性。
研究人员曾试图产生几个碱基的合成DNA正在构建整个基因组megabases的规模。2016年3月,测序和合成生物学先驱克雷格·文特尔和他的同事宣布他们修剪和重写的细菌的基因组支原体mycoides从1 megabase 531碱基来创建一个最小的基因组3——最小的一组基因,是生命所必需的。
2016年8月,由乔治教堂,波士顿哈佛医学院的遗传学家,马萨诸塞州和Nili Ostrov,他的实验室的一位博士后研究员,报道说,他们已经产生了一种细菌,被称为“rE.coli-57”,七个密码子的三胞胎核苷酸编码特定的氨基酸——被剥离,取而代之的是同义的替代品4在这一过程称为基因重新编码。和2017年3月,领导的一个团队帕梅拉银,生化学家在哈佛大学生物工程学院在波士顿,马萨诸塞州,描述其最初试图重新编码LT2菌株的基因组的细菌鼠伤寒沙门氏菌5,取代约200个碱基的基因组DNA,并消除特定白氨酸密码子,希望阻止基因致病微生物之间的转移。
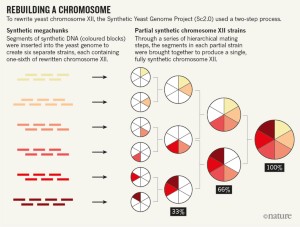
最显著,2017年3月,杰夫Boeke)领导的一个国际财团在纽约大学兰贡医学中心和乔尔·巴德位于巴尔的摩的约翰霍普金斯大学、马里兰、报道16条染色体的端到端改写为5酿酒酵母6——一个里程碑在国际项目称为合成酵母基因组计划(Sc2.0)。Sc2.0旨在优化和合成的完整基因组酿酒酵母对工业和纯粹的研究应用。Boeke说,例如,通过删除所有不编码蛋白质的DNA序列(内含子),团队可以评估所需的细胞机制的生物角色处理这些遗传元素。
酵母人工染色体设计用于简化和稳定了Sc2.0删除重复序列和基因内区,通过移动序列编码的关键部分蛋白质转译机械专用的染色体,通过消除密码子标签,这信号停止翻译,并取代另一种终止密码子,TAA,促进蛋白质工程。一个定制的软件包称为BioStudio启用团队管理所需的基因记账完成如此巨大的任务。

Sc2.0被大量的物流,爱丁堡大学的Patrick演变中Cai说,英国是项目的国际协调。然而实际的酵母染色体相当常规的编辑过程中,需要几个盘子酵母的孵化器,莱斯利·米切尔说博士后Boeke的实验室领导的小组合成染色体VI。
Sc2.0团队使用的策略称为换入重写染色体一块一块的(见“重建染色体”)。研究人员首先将短的DNA单链分子称为寡核苷酸组装成大约750基地的构建块,然后成“块”的10个碱基或更少,反过来,是30 - 60个碱基的组合成“megachunks”。每个megachunk包含两种标记基因,使酵母携带这些基因的选择——在这种情况下,URA3,使酵母生长在没有尿嘧啶,和LEU2亮氨酸缺失时,使经济增长。megachunks然后开槽成现有的染色体通过同源重组,一个自然的过程,一段DNA被替换为另一个重写DNA从一端到另一端。因为每个后续部分集成,它取代了前一段的标志基因,尿嘧啶之间交换酵母细胞的营养需求和亮氨酸。后定量PCR分析,确保每个megachunk已经完全合并,生成的酵母菌株检测的能力,形成殖民地相对严格的条件下;其增长放缓或死亡与野生型相比表明问题需要修复。“我们的想法是:整合megachunk,测试健身”,并重复,米切尔解释道。
文思枯竭
单个megachunk可以集成和测试在大约两周,米切尔说,假设没有问题。体能测试和“调试”(纠错)“需要更长的时间比实际构建在这一点上”,Boeke说。
每个染色体完成了到目前为止只有少数著名的“错误”,他说。一些源于基因组注释中的错误,而另一些是由于密码子替换,例如,改变RNA二级结构。
在大多数情况下,酵母与拳卷。然而,故障,出现暗示称为基因组的大规模的工程项目面临的挑战Project-write (GP-write),旨在重写更复杂的真核生物的基因组。除了明显的问题规模——在30亿个碱基对,人类基因组比的大两个数量级酿酒酵母——复杂的生物体的基因组会少注释。当文特尔的团队首先试图建立一个最小基因组m . mycoides,它应用一个理性的设计,使用基因数据编译出版重要基因的列表——这种方法不起作用。“我们缺乏基本的生物学知识,即使最简单的细菌的基因组,是巨大的,”文特尔说。成功不是来自一个自上而下的方法,削弱了基因组到达一组核心的473个基因。但约三分之一没有已知的函数。“我发现一种令人兴奋的结果,“银说。
还有进一步的挑战。Sc2.0和其他genome-rewriting项目往往避开监管区域的基因,但在真核生物等复杂的生物体,这些通常远离他们的基因影响,并且可能尚未完全映射。研究人员可能因此不知道哪段重写,并独自离开。还不清楚这种大规模基因组的变化如何影响染色质结构和基因表达。
在实践层面,chromosome-sized DNA分子无法轻易操纵没有被打破,并没有有效的方法将它们传递到大多数真核细胞。即使科学家可以提供DNA,他们可能无法将其集成到基因组,因为大多数这些细胞无法执行同源重组酵母一样容易,和他们的经济增长放缓拖出每个实验步骤。
也有合成DNA的成本需要考虑。银的团队接到美国国防高级研究计划局资助她的工作美国沙门氏菌感染,它允许谈判DNA合成的优惠价格。但每个基站的价格0.10美元,她说,它将花费超过100万美元来完成自己的项目;相比之下,人类基因组将花费数百倍。
它会变得容易随着时间的推移建立庞大的基因组。
然而教会说,这只是一个时间问题技术赶上了野心。“我的猜测是,它要容易随着时间的推移建立大型基因组。”
精密重写
基因组重写到目前为止在很大程度上,仍在大自然的配方。但最终,生物学家希望赋予新的功能。
几个项目,包括rE.coli和美国沙门氏菌感染研究,都在关注基因重新编码,密码子的基因释放其他用途。杰森的下巴,合成生物学家在剑桥大学医学研究委员会分子生物学实验室,英国已经做了大量工作来操纵遗传密码。他说,这样的重新编码可以促进蛋白质工程,更不用说设计、测试和合成标准以外建造的新型化学聚合物单体氨基酸。其他可能的应用包括biocontainment(防止释放有机体外部实验室)和遗传隔离(保护生物体免受病毒感染)。
2005年,与教堂做博士后,艾萨克,现在生物在耶鲁大学在纽黑文,康涅狄格州,开始追求的想法重新编码大肠杆菌基因组,精力关注取代了终止密码子标签。
因为大肠杆菌仅包含321标签密码子,艾萨克斯能够完成这项任务通过修改现有的基因组而不是从头合成一个7。使用一种称为法师的策略(多路自动基因组工程),使多个DNA序列编辑,艾萨克斯和他的同事们第一次分裂大肠杆菌基因组到32码段和改变了标记在每一个同义密码子TAA。接下来,他们加入了32修改段划分为一个单分子利用细菌之间的基因交换的自然过程。完成重新编码过程中,团队删除一个基因编码一种蛋白质被称为RF1,识别密码子标签。(相关的蛋白质,RF2,认识到密码子TAA)。生存的修改大肠杆菌后删除此否则至关重要的基因显示,他们重新编码过程工作。
许多研究人员,现有技术提供所有他们需要黑客基因组。eGenesis位于剑桥,马萨诸塞州,是使用基因编辑工具CRISPR把猪变成移植器官的来源。Luhan杨公司的共同创始人,解释说,这个想法是为了削减使用CRISPR去除猪基因组序列编码蛋白质,会引发免疫反应的人。新基因编码的蛋白质,有助于使猪组织符合人类也可以了。“我们认为许多修改可能就足够了,”她说。
但更大规模项目的方法是不同的。Ostrov和教堂的rE.coli项目,例如,移除标签停止密码,两个密码子丝氨酸,精氨酸和亮氨酸大肠杆菌创建一个57-codon应变4。工作需要62214变化,团队使用自下而上的DNA合成,而不是自上而下的编辑。很多必要的基因改造,Ostrov解释说,“我们不妨让基因组从头开始。”
这些genome-hacking研究已经建立了一个chromosome-sized DNA分子在一个连续的延伸。大多数商业供应商合成DNA依赖于一个几十年的合成方法,不适合生产分子超过大约200个核苷酸。教堂的团队,大多数组织追求基因组合成、组装DNA需要层级。这购买现成的基因片段长2 - 4个碱基,组装成50-kilobase-long块酵母利用同源重组和转移这些段划分为完成大肠杆菌。该小组随后删除相应的区域大肠杆菌基因组,为健身和测试结果的细菌菌株。
根据Ostrov,重新编码过程很顺利,尽管有一些错误。例如,改变一个特定基因的编码序列无意中削弱了一个重叠基因的启动子,减少压力的健身。
“基因组中有特殊的信息,”解释了下巴,它只能解密后的实验。
电路城
其他研究人员正在开发基因电路基因组灌输新功能。
一般来说,这些电路,如沃伊特的拍照的细菌菌株,建立从简单的设计使用称为转录因子的蛋白质作为正面或负面的输入和输出信号。Wilson Wong在马萨诸塞州波士顿大学的生物医学工程师,建立他的设计而不是使用酶称为可反转或删除片段的DNA——一个设计策略称为刀片(布尔逻辑和算术通过DNA切除)。
Wong说,叶片释放人员连接电路的难度,这就需要一个电路的输出强度与未来的预期的输入。
在一个演示中,黄和他的团队创造了一个布尔逻辑查表8——基因电路,长约10个碱基,能够变成任何的16个逻辑门取决于6可存在。
黄的团队设计,电路使用铅笔和纸。但最终,合成生物学家希望建立他们的设计在网上。沃伊特与道格拉斯Densmore,波士顿大学的一个电气工程师开发了一个工具叫大提琴(cellocad.org),要使这成为可能。研究人员指定基因电路的设计在一个叫Verilog的编程语言,和大提琴产生所需的DNA序列,使它们的工作9。
尽管他们看起来简单,微生物的基因组展示不可思议的微妙的基因控制的能力,沃伊特说。“我们几乎嘲笑所存在于自然。“但是通过结合基因组编辑、基因组合成和巧妙设计工具,研究人员正在慢慢的再平衡尺度。
引用
Fernandez-Rodriguez, j . et al。化学性质。医学杂志。13,706 - 708 (2017)。
Zakeri, b &卡尔,p。生物科技趋势》。3357-58 (2015)。
和记黄埔,c . a . III et al。科学351年aad6253 (2016)。
Ostrov: et al。科学353年,819 - 822 (2016)。
刘,y . h . et al。核酸Res。45,6971 - 6980 (2017)。
理查森,s m . et al。科学355年,1040 - 1044 (2017)。
艾萨克斯,f . j . et al。科学333年,348 - 353 (2011)。
Weinber, b . h . et al。生物科技自然》。35,453 - 462 (2017)。
尼尔森,a . a . k . et al。科学352年aac7341 (2016)。
作者信息
作者指出
杰弗里·m·Perkel自然是技术编辑
- 杰弗里·m·Perkel
权利和权限
关于这篇文章
引用这篇文章
Perkel, j .如何破解的基因组。自然547年,477 - 479 (2017)。https://doi.org/10.1038/547477a
发表:
发行日期:
DOI:https://doi.org/10.1038/547477a
