摘要
核孔复合物是所有真核细胞的基本组成部分,介导核质交换。确定它们110兆达尔顿的结构是一项艰巨的挑战原位结构生物学方法。在大约30个核孔蛋白(Nups)中,15个是有结构的并形成Y和内环复合物。这两个主要的支架模块在多个副本中组装成一个八倍旋转对称结构,融合内外核膜,形成一个直径约60纳米的中心通道1.支架上装饰有运输通道Nups,通常含有苯丙氨酸重复序列,并介导与货物复合物的相互作用。虽然Y综合体的部分建筑布局已经阐明,但尚不清楚它究竟是如何寡聚的原位.在这里,我们将冷冻电子断层扫描与质谱、生化分析、摄动实验和结构建模相结合,以生成迄今为止最全面的人类核孔复合体建筑模型。我们的数据表明,以前未知的蛋白质界面横跨Y复合体和内环复合体成员。我们发现传输通道Nup358(也称为Ranbp2)在y -络合物的寡聚过程中发挥了以前没有预料到的作用。我们的发现模糊了支架和转运通道Nups之间的既定界限。我们得出结论,与包被囊泡类似,同一结构构件的几个副本-尽管成分相同-参与不同的局部相互作用和构象集。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
核孔组合的定量图揭示了两种不同的机制
自然开放获取2023年1月04日
SMLM数据定量分析的最大似然模型拟合
自然方法开放获取2022年12月15日
渗透转变规定了蛋白质大小特异性的障碍,被动运输通过核孔复合体
自然通讯开放获取2022年9月1日
访问选项
订阅期刊
获得1年的完整期刊访问权限
199.00美元
每期仅需3.90美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。
参考文献
霍尔兹,A.,德布勒,E. W. &布洛贝尔,G.核孔复合物的结构。为基础。学生物化学启。80, 613-643 (2011)
Vollmer, B. & Antonin, W. Nup93/Nic96复合物蛋白的不同作用-核孔复合物的结构支架与额外的细胞功能。医学杂志。化学。395, 515-528 (2014)
Hurt, E. & Beck, M.在整合结构分析时代理解核孔复杂结构和动力学。咕咕叫。当今。细胞生物。34, 31-38 (2015)
Bui, K. H.等。人核孔复合支架的综合结构分析。细胞155, 1233-1243 (2013)
坎普曼,M. & Blobel, G.核孔复合物膜涂层模块的三维结构和灵活性。自然结构。摩尔。杂志。16, 782-788 (2009)
Thierbach, K.等人。保守的Nup84复合物的蛋白质界面毛壳菌属thermophilum交联质谱和电子显微镜显示。结构21, 1672-1682 (2013)
Fornerod, M., van Baal, S., Valentine, V., Shapiro, D. N. & Grosveld, G.编码CAN/ nup214相互作用蛋白的基因的染色体定位-人类CRM1定位于2p16,而Nup88定位于17p13,并与SF2p32连接。基因组学42, 538-540 (1997)
Werner, A., Flotho, A. & Melchior, F. RanBP2/RanGAP1*SUMO1/Ubc9复合物是一种多亚基SUMO E3连接酶。摩尔。细胞46, 287-298 (2012)
Ori, A.等人。细胞类型特定的核孔:分子机器环境依赖的化学计量学的一个例子。摩尔。系统。医学杂志。9, 648 (2013)
Stuwe, T.等人。核孔。核孔复合涂层的结构。科学347, 1148-1152 (2015)
Bilokapic, S. & Schwartz, T. U. Nup37和ELY5/ELYS招募到核孔复合体的分子基础。国家科学院学报美国109, 15241-15246 (2012)
Seo, H. S.等。对Nup120的结构和功能分析表明,Nup84配合物为环状结构。国家科学院学报美国106, 14281-14286 (2009)
纳吉,V.等人。三聚体核孔蛋白复合物的结构揭示了交替的寡聚态。国家科学院学报美国106, 17693-17698 (2009)
阿尔伯,F.等。核孔复合体的分子结构。自然450, 695-701 (2007)
Boehmer, T., Jeudy, S., Berke, i.c. & Schwartz, T. U. Nup107/Nup133相互作用的结构和功能研究及其对核孔复合体结构的影响。摩尔。细胞30., 721-731 (2008)
格莱维,J. S.等。核孔Nup107-160亚复合物的细胞周期依赖性磷酸化。国家科学院学报美国104, 3811-3816 (2007)
劳雷尔,等人。在有丝分裂进入过程中,Nup98被多个激酶磷酸化是NPC分解的关键。细胞144, 539-550 (2011)
Shi Y.等。通过交联的结构表征揭示了核孔复合体中与共原子相关的七聚体模块的详细结构。摩尔。细胞。蛋白质组学13, 2927-2943 (2014)
Culjkovic-Kraljacic, B, Baguet, A, Volpon, L., Amri, A. & Borden, K. L.致癌基因eIF4E重组核孔复合物以促进mRNA输出和致癌转化。细胞的代表。2, 207-215 (2012)
金,d。i。等人。用邻近依赖生物素化方法探测核孔复合体结构。国家科学院学报美国111, e2453-e2461 (2014)
弗莱明,D.等。对酵母核孔蛋白Nup188的分析显示其具有与核啡蛋白相似的s型保守结构。j . Struct。医学杂志。177, 99-105 (2012)
Theerthagiri, G., Eisenhardt, N., Schwarz, H. & Antonin, W.核oporin Nup188控制膜蛋白通过核孔复合体。J.细胞生物学。189, 1129-1142 (2010)
达梅林,M. & Silver, p.a.。原位核孔复合体蛋白质间空间关系的分析。Biophys。J。83, 3626-3636 (2002)
盖克,等人。Nup82配合物在核孔支架中的组装和功能的结构基础。J.细胞生物学。208, 283-297 (2015)
泽普,我等人。40S核糖体亚单位前体的不同细胞质成熟步骤需要hRio2。J.细胞生物学。185, 1167-1180 (2009)
Ori, A., Andres-Pons, A. & Beck, M.使用靶向蛋白质组学来确定大分子组装的化学计量学。方法细胞生物学。122, 117-146 (2014)
在洗涤剂和脂类存在的稀溶液中定量回收蛋白质的一种方法。分析的物化学。138, 141-143 (1984)
自动电子显微镜断层扫描使用样本运动的稳健预测。j . Struct。医学杂志。152, 36-51 (2005)
李,X.等。电子计数和束流诱导运动校正使近原子分辨率的单粒子冷冻电子显微镜成为可能。自然方法10, 584-590 (2013)
Schur, F. K, Hagen, W. J., de Marco, A. & Briggs, J. A.在8.5 Å分辨率下使用冷冻电子层析成像和亚层析成像平均测定蛋白质结构。j . Struct。医学杂志。184, 394-400 (2013)
Movassagh, T., Bui, K. H., Sakakibara, H., Oiwa, K. & Ishikawa, T.核苷酸诱导的鞭毛动力蛋白臂的全局构象变化原位分析。自然结构。摩尔。杂志。17, 761-767 (2010)
Kosinski, J., Barbato, A. & Tramontano, A. MODexplorer:用于探索蛋白质序列,结构和功能关系的集成工具。生物信息学29, 953-954 (2013)
Guex, N. & Peitsch, M. C. SWISS-MODEL和Swiss-PdbViewer:比较蛋白质建模的环境。电泳18, 2714-2723 (1997)
Kurowski, m.a.和Bujnicki, j.m. GeneSilico蛋白质结构预测元服务器。核酸测定。31, 3305-3307 (2003)
埃斯瓦尔等人。使用modeler进行比较蛋白质结构建模。蛋白质科学进展第2章(2007)
彼得森,E. F.等人。UCSF chimera -用于探索性研究和分析的可视化系统。j .第一版。化学。25, 1605-1612 (2004)
Whittle, J. R. & Schwartz, T. U.建筑核oporins Nup157/170和Nup133在结构上是相关的,起源于第二个祖先元素。生物。化学。284, 28442-28452 (2009)
Suhre, K. & Sanejouand, Y. H. ElNemo:用于蛋白质运动分析和分子替换模板生成的普通模式web服务器。核酸测定。32, w610-w614 (2004)
德弗里斯,S. J.等。HADDOCK与HADDOCK: CAPRI目标上HADDOCK2.0的新特性和性能。蛋白质69, 726-733 (2007)
Minguez, P.等人。PTMcode v2:蛋白质内部和之间翻译后修饰的功能关联的资源。核酸测定。43, d494-d502 (2015)
Kucukelbir, A., Sigworth, F. J. & Tagare, H. D.对低温电磁密度图的局部分辨率进行量化。自然方法11, 63-65 (2014)
瓦尔特,t.c.等人。核孔复合体的细胞质细丝对于选择性输入核蛋白是可有可无的。J.细胞生物学。158, 63-77 (2002)
Drin, G.等人。用于膜曲率传感的一般两类α-螺旋基序。自然结构。摩尔。杂志。14, 138-146 (2007)
确认
我们非常感谢W. Baumeister和J. Plitzko访问马克斯·普朗克生物化学研究所的电子显微镜设备。我们感谢F. Schur, K. Beck, J. Briggs, C. Sachse, E. Hurt和F. Melchior的批判性建议,以及A. Neal对手稿的批判性阅读。我们感谢EMBL的机械车间,电子显微镜和蛋白质组学核心设施,统计数据分析中心的支持,并感谢J. Krijgsveld, J. Kirkpatrick和B. Klaus。K.H.B.由瑞士国家科学基金会、欧洲分子生物学组织和玛丽·居里行动提供博士后奖学金。A.O.得到了亚历山大·冯·洪堡基金会和玛丽·居里行动的博士后奖学金的支持。A.L.D.由史蒂文斯理工学院罗伯特·克鲁克斯·斯坦利奖学金和国家老龄化研究所(NIA)资助1R21AG047433-01。玛丽·居里共同基金行动下的EMBL跨学科博士后项目支持。J.S.G.获得了史蒂文斯理工学院的点火资助计划和NIA资助1R21AG047433-01。M.B.感谢EMBL和欧洲研究理事会(309271-NPCAtlas)的资助。
作者信息
作者及隶属关系
贡献
a.v.a., J.K.和W.A.设计并进行实验,分析数据并撰写手稿。a.v.a., J.K.和P.K.担任模特。B.V。l ., A.O。A.L.D, M.-T.M, K.B, A.A.-P W.H。,N.B. and S.M. designed and performed experiments, and analysed data. L.P. analysed data and performed modelling. J.S.G., E.A.L. and P.B. designed experiments and oversaw the project. K.H.B. designed and performed experiments, analysed data, oversaw the project and wrote the manuscript. M.B. designed experiments, analysed data, oversaw the project and wrote the manuscript.
相应的作者
道德声明
相互竞争的利益
作者声明没有相互竞争的经济利益。
扩展的数据图形和表格
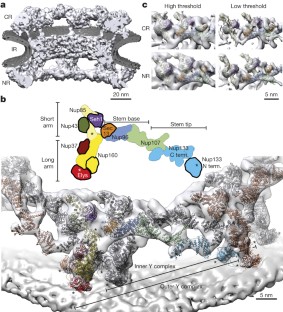
扩展数据图1人体NPC层析图。
一个使用直接电子探测器(本研究)获得的人鼻咽癌层析结构的核浆轴、CR、IR和NR的正切片,与使用类似实验流程的传统探测器获得的结构进行比较(电子显微镜数据库登录号EMD-2444)。在这两种情况下,CR、IR和NR是独立对齐的。箭头表示在IR中被解析的跨膜域。b, CR、IR和NR区域的傅里叶壳层相关曲线。c,直方图对应于中所示的彩色编码局部分辨率图d这是用ResMap计算的41.d,在两个不同的等值面阈值处显示NR环的单个节段(在所有其他图中,节段与它们的前和/或后不对称单元一起显示)。用两个相邻非对称单元的Nup43 β-螺旋桨外区域(水平箭头)和Nup133中区域(垂直箭头)的冗余密度来指明方向。边缘分辨率的降低是由于在对齐和平均期间使用的掩模的边界,覆盖了大约1.5倍的非对称单元。
扩展数据图2 b因子校正。
将滤波后的x射线结构与用不同b因子校正的层析图进行比较,以选择合适的b因子。一个, Nup107-Nup96-Sec13的x射线结构(上),滤波到23 Å(下),与b因子校正的外顶点各区域(2000 - 9000 Å)相比2.b, b因子6000 - 8000 Å2最接近x射线结构的特征。层析成像图的三个区域在b因子为6000 Å处与各自的x射线结构叠加2相比之下,8000 Å2.在这些良好分辨的区域,额外的特征,如更详细的β-螺旋桨形状或Nup107手指结构域(参见图2a, b而且扩展数据图4e)的含量明显为8000 Å2.由于局部分辨率的偏差(扩展数据图1c, d)更保守的b因子为6000 Å2被选来校正平均值。星号表示可以明确定位但在方向上有一些不确定的结构,即Nup85羧基末端结构域(CTD)。c,将Nup107-Nup96-Sec13的x射线结构系统拟合到层析图中,如图扩展数据图7a而是在不同的b因子上。调整P数值按顺序显示;插图中用红色显示了四个真正的正面击球。后者始终被认为是最热门的,除非b因子为9000 Å2使用。
图3 Y复合体混合模型与顶点区域x射线结构的比较10.
左侧所示的Y复合体(NR)的混合模型是独立于右侧所示顶点的坐标x射线结构生成的。根据空间约束和互补信息,将Y复合体成员的结构拟合到层析图中补充表1详情)。外部和内部的Y复合体/顶点分别显示在顶部和底部。一个人体Y复合物的分子量约为1个MDa,其中大部分是结构化的。星号表示可以明确定位但在方向上有一些不确定性的结构。这些是Elys和Nup133以及Nup85-CTD的β-螺旋桨。
内部和外部的Y配合物具有不同的构象,并参与局部特定的相互作用集。
一个,从上面可以看到内、外Y配合物的排列。x射线结构被过滤到2.3 nm分辨率,并用蛋白质着色。指出了Y配合物之间五个界面的位置。b, Nup160的内拷贝和外拷贝相对于膜具有相同的法向量,由于与中轴的直径不同,它们彼此略有倾斜。cNup107 C端和n端结构域之间以及Nup133 C端结构域内的铰链在内部和外部Y配合物中具有不同的构象。在内杆的情况下,与x射线结构中揭示的结构相比,Nup133的中间区域略微向内弯曲,这可以通过引入铰链来解释,正如先前基于Nup133结构的预测37.d,显示五个界面细节(I-V)的放大视图。右边的面板表示观察点。(I)外顶点的Sec13与内顶点的Nup107 n端域交界4,10;(II)在两种Y配合物的情况下,Nup133的n端β-螺旋桨与后部不对称单元的Nup160界面,形成有利于环形成的头尾接触12,14;(III)因此,只有Nup133内拷贝的β-螺旋桨被夹在两个Nup160蛋白之间。在这种情况下,n端α-螺旋结构域与后对称单元的外层Nup160延伸成一个更大的界面;(IV)内Nup133的C端分支出内杆,与外杆上的对应端形成接触。这个相对较小的界面让人联想到在Nup133-Nup107结构中观察到的晶体接触(蛋白质数据库登录号3I4R)37;外顶点的(V) nup85和43与内杆的Nup107的c端畴形成接口。eNup107手指结构域(红色)是高等真核生物所特有的。当它的内拷贝(如图所示)与外顶点的Nup43和Nup85连接时,它的外拷贝与连接两个Y复合体的密度(图2a, b).指域中的磷酸化位点被描绘为暗红色球体。星号表示可以明确定位但在方向上有一些不确定性的结构,即Nup85-CTD。f,从中心通道看CR的顶点区域。磷酸化位点表示为e.连接两个Y复合体的密度是分段的,如图2 b并且与Sec13、Nup96和Nup107的几个磷酸化位点密切接触。Seh1和Nup85的磷酸化位点直接靠近Nup214复合体区域。
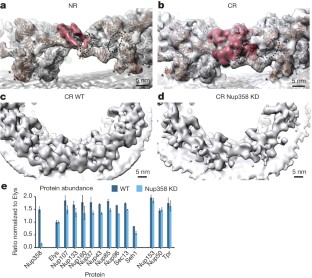
扩展数据图5内、外Y复合体连接。
一个, CR中两个Y配合物的Nup96/107区域有四个钳形密度(红色部分)。只有内部的钳形连接两个Y配合物,而外部的钳形突起在CR的外部外围形成一个更复杂的子结构(图2 b).b和在图2c, d但对于NR。c火山图可视化了Nup358敲除(处理)中HeLa细胞的鸟枪蛋白质组学数据,与对照条件进行了比较。d, NLS-MBP-GFP(即与gfp标记的麦芽糖结合蛋白结合的核定位序列)在未处理细胞和Nup358敲除条件下的核转运测定。如先前观察到的,在没有Nup358的情况下,进口具有经典核定位信号的货物42,但不能排除效率较低。e,击倒条件的亚层析图分类显示,约5%的不对称单元在CR中含有外层Y配合物,这与的击倒效率非常一致∼95%。分类是在非对称单位的水平上进行的。转移到npc水平,这表明在基因沉默条件下观察到的920个npc中,663个CR中没有外部Y复合物,183个有一个,49个有两个,14个有三个,4个有四个,6个有五个,0个有六个,1个有七个,0个有八个。f在击倒条件下,CR中观察到的外层Y配合物的邻接性远高于预期。在NPC水平上分析CR中含有外部Y配合物的5%的不对称单元,以确定其相邻的不对称单元在CR中是否也含有外部Y配合物。观察到的邻接频率以深蓝色显示。分别在CR中观察到的外层Y配合物的总数和具有相邻配合物的数量表示为(n/米).观察到的频率大大高于理论频率(亮蓝色所示),如果Y配合物与随机亚基结合,则理论频率将被预期。这一观察表明,在CR中Y配合物的组装/维护可能通过相邻Y配合物的头尾接触而产生。每个CR有0个、1个和8个外部Y复合物的npc没有显示出来,因为它们不包含任何相邻的Y复合物或没有被观察到。
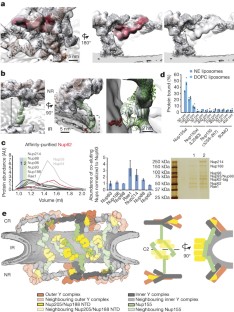
扩展数据图7所选NPC组分与电子显微镜图的系统拟合。
每个面板显示20个得分最高的拟合(左),一个图P所有解的值(右)和插图中的顶部解。用于拟合的模型显示为带状表示。拟合和数据点根据相似的组进行着色P值范围。这群人最适合PValues被标上红色(高置信度匹配),次优组(中等置信度匹配)被标上蓝色,其余的都被标上青色。为了清晰起见,膜密度已被去除。一个Sec13-Nup96-Nup107子复合体,其基础真相是已知的,因为它是顶点的一部分。b- - - - - -d,与一个而Nup155的n端结构域分别为Nup205/188- ntd的开放构象(模板Nup205)和Nup205/188- ntd的封闭构象(模板Nup188)。解id,解的单位。
扩展数据图8共洗脱分析检测弱核孔蛋白相互作用
为了检测支架核孔蛋白的弱相互作用,我们结合快速亲和分离凝胶过滤和定量靶向蛋白质组学来测量绝对蛋白质丰度。HEK293细胞表达各种亲和标记的Nups(与图3 c没有诺可达唑阻滞)被用温和的条件和超声溶解蛋白。亲和分离物进行凝胶过滤,所有组分都使用靶向质谱分析,如前所述24,26,以测量高分子量和低分子量部分的蛋白质丰度。高分子质量分数表明了大分子物种潜在的外向相互作用。低分子量部分将突出超声处理后出现的较小碎片。一个,在亲和标记的Nup85的情况下,最上面的面板显示了215 nm的凝胶过滤实验吸收曲线。中间的面板显示了所有分数中y复合体成员的任意蛋白质丰度单位(红色为Nup85,黑色为所有其他y复合体成员)。高分子部分(蓝色条)和低分子部分(绿色条)的蛋白质丰度(归一化到亲和标记蛋白质)在底部面板中以柱状图的形式显示。低分子峰对应于小臂蛋白(Nup85, Seh1和Nup43),高分子峰对应于完整的Y复合体。b对Nup205、Nup93和Nup155进行了同样的处理。在高分子部分(蓝色条形图)和低分子部分(绿色条形图)显示了七个最丰富的共洗脱Nups。以Nup85(上)为例,除了y复合体成员外,显示了7个最丰富的蛋白质。y -络合物成员与Nup205和Nup93的弱相互作用明显。在Nup155的情况下,检测到与CR和NR元件之间的弱相互作用,以及在IR与CR/NR连接的密度中,定位于Nup155 c端结构域附近的Sec13。Tpr被排除在分析之外,因为它存在于所有馏分中。基于单个参考肽的蛋白质丰度用星号标记。c,同一个(上图),但从诺可达唑阻滞细胞纯化的Nup188亲和性。d,同图3 c但从诺可达唑阻滞细胞纯化的Nup188亲和力。高分子量部分的共洗脱物与亲和纯化Nup62中观察到的共洗脱物相似(图3 c).检测到Nup188、Nup98、Nup93、Nup62等化学计量量,Nup214、Nup88和ra1富集。因此,Nup188-Nup93异源二聚体与CR中已确定的组分Nups结合,这与系统拟合方法一致。e,同c但是对应于图3 c.f,同b(第二组为Nup205),但为诺可达唑阻滞细胞。在Nup205的情况下,与未处理的细胞相比,诺可达唑被捕的共纯化物种相似。
补充信息
补充信息
该文件包含补充表1和其他参考文献。(PDF 1548kb)
y配合物的结构分析原位
NR的一段显示等值面渲染。高分辨率结构显示叠加;颜色代码如图1和3e所示。(mp429488 kb)
权利和权限
关于本文
引用本文
冯·阿彭,A.科辛斯基,J.斯帕克斯,L.et al。原位人体核孔复合物的结构分析。自然526, 140-143(2015)。https://doi.org/10.1038/nature15381
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature15381
这篇文章被引用
核孔组合的定量图揭示了两种不同的机制
自然(2023)
SMLM数据定量分析的最大似然模型拟合
自然方法(2023)
回复:评估细胞定量结构生物学的3D MINFLUX数据
自然方法(2023)
细胞定量结构生物学的三维MINFLUX数据评估
自然方法(2023)
非洲爪蟾核孔复合物核环的低温电镜结构
细胞研究(2022)
