摘要gydF4y2Ba
长期的气候变化和周期性的极端环境威胁着粮食和燃料安全gydF4y2Ba1gydF4y2Ba全球农作物产量gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba。虽然分子育种和适应性育种策略可以缓冲气候胁迫的影响,提高作物的抗逆性gydF4y2Ba5gydF4y2Ba但是,这些方法需要对构成生产力和适应性的基因有足够的了解gydF4y2Ba6gydF4y2Ba-仅限于少数经过充分研究的模型系统的知识。本文介绍了多倍体生物能源作物柳枝稷(gydF4y2Ba黍virgatumgydF4y2Ba).对732个重测序基因型的生物量和存活率进行了分析,这些基因型生长在跨越1800公里纬度的10个常见花园中,共同揭示了气候适应的广泛基因组证据。气候-基因-生物量关联丰富,但在深度分化的基因库中差异较大。此外,我们发现基因流通过来自预先适应的北方基因库的等位基因的渗入,加速了冰川后北方栖息地殖民化过程中的气候适应。柳枝稷的多倍体特性也通过基因功能的分化增强了适应潜力,因为在非显性亚基因组上有更高水平的遗传多样性。除了研究气候适应模式外,这里开发的基因组资源和基因性状关联为育种者提供了提高柳枝稷产量的必要工具,以实现生物能源的可持续生产。gydF4y2Ba
主要gydF4y2Ba
柳枝稷(gydF4y2BaP。gydF4y2BavirgatumgydF4y2Ba)是一种很有前途的生物燃料作物,也是北美高草草原的重要组成部分。从历史上看,高草草原是地球上最大的温带生物群落之一,它们仍然是大气碳的重要汇gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba。然而,大多数现存的天然柳枝稷种群都局限于“遗迹”遗址,这些遗址代表着对未来高草草原的保护和繁殖至关重要但正在减少的遗传资源。gydF4y2Ba
生物质生产是柳枝稷作为饲料和生物能源作物的主要育种目标gydF4y2Ba9gydF4y2Ba是种子生产和进化适应性的有力代表gydF4y2Ba10gydF4y2Ba。自从美国能源部将柳枝稷命名为典型的草本生物燃料原料以来,生物质产量试验已经证明了柳枝稷生物能源生产的经济可行性,并且已经培育出的品种大大超过玉米和其他纤维素原料gydF4y2Ba11gydF4y2Ba。然而,单个品种往往只在一个狭窄的气候生态位上生产。因此,为了实现收益最大化,柳枝稷育种和生物技术应重点发展气候基因型匹配gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba通过鉴定种群生物量积累和气候适应的基因组基础。这将提振未来的收益率gydF4y2Ba14gydF4y2Ba并将柳枝稷作为一种经济上和环境上可持续的生物能源产品。gydF4y2Ba
四倍体柳枝稷基因组gydF4y2Ba
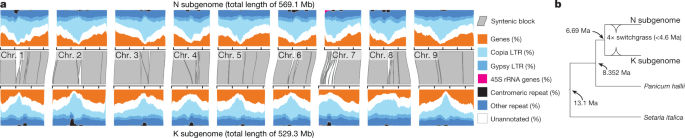
尽管大量的数量遗传变异是气候相关的胁迫耐受性和生物量生产的基础gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba然而,先前柳枝稷基因组序列的碎片化和不完整性阻碍了候选基因的发现和其他分子育种工作。AP13柳枝稷基因型基因组大(单倍体基因组大小= 1129.9兆碱基(Mb)),重复(56.9%重复)(图2)。gydF4y2Ba1gydF4y2Ba扩展数据图gydF4y2Ba1gydF4y2Ba)和多倍体。与其他一些异种杂交物种,如玉米(以自交系B73参考基因组为代表)相比,AP13是近交的。其基因组在自然异交群体范围内保持了相当水平的杂合性(扩展数据图)。gydF4y2Ba1gydF4y2Ba).尽管存在这种复杂性,我们的深度PacBio长读测序加上深度短读抛光和细菌人工染色体(BAC)克隆验证产生了高度连续的“v5”AP13基因组组装(扩展数据图)。gydF4y2Ba1gydF4y2Ba;数据可从Phytozome网站获得gydF4y2Bahttps://phytozome-next.jgi.doe.govgydF4y2Ba).我们修剪了得到的大组合(NgydF4y2Ba50gydF4y2Ba= 5.5 Mb)为单个代表性单倍型,然后使用两个高密度遗传图谱的共识定位和排序为染色体假分子(补充数据gydF4y2Ba1gydF4y2Ba).染色体通过遗传距离分配到亚基因组gydF4y2Ba黍rudgeiigydF4y2Ba17gydF4y2Ba的K亚基因组的姊妹分类群gydF4y2Bap . virgatumgydF4y2Ba),并通过从头开始重复聚类。最终版本仅包含0.4%的漏洞,与2016年的v4版本相比减少了75倍。gydF4y2Bahttps://phytozome-next.jgi.doe.gov/info/Pvirgatum_v4_1gydF4y2Ba).重要的是,基因组组装与三个遗传信息来源是共线性的,尽管它是独立于这三个来源组装的:一个近二倍体亲戚(gydF4y2Ba黍halliigydF4y2Ba),伪f的标记顺序gydF4y2Ba2gydF4y2Ba替代亚基因组的遗传图谱和基因顺序(图2)。gydF4y2Ba1gydF4y2Ba扩展数据图gydF4y2Ba1gydF4y2Ba、补充数据gydF4y2Ba2gydF4y2Ba).这些共线性表明我们已经为每个亚基因组开发了一个单倍体组装和注释。gydF4y2Ba
一个gydF4y2Ba,灰色多边形(表示gydF4y2BangydF4y2Ba= 53个synsynblock)显示亚基因组之间几乎完全共线性。基因丰富的染色体臂和高度重复的中心粒是草基因组的典型特征。LTR,长端重复。gydF4y2BabgydF4y2Ba根据校准的时间尺度系统发育树估计了<4.6 Ma的亚基因组分化gydF4y2Ba黍gydF4y2Ba- - - - - -gydF4y2BaSetariagydF4y2Ba节点在13.1 Ma。gydF4y2Ba
至关重要的是,我们能够区分两个亚基因组之间的基因和重复序列。基因注释来自Illumina RNA测序(gydF4y2BangydF4y2Ba库gydF4y2Ba= 88,gydF4y2BangydF4y2Ba条件gydF4y2Ba= 18, bbb30亿reads)和PacBio Iso-Seq (gydF4y2BangydF4y2Ba条件gydF4y2Ba= 9, > 450万次读取,补充数据gydF4y2Ba3.gydF4y2Ba) -包含80,278个初级转录本和49,664个替代转录本,并且与基因组组装一样完整(BUSCO = 99.4%)(扩展数据图。gydF4y2Ba1gydF4y2Ba).我们利用这些注释建立了多个序列比对和时间尺度的系统发育树,将亚基因组祖先物种的分化追溯到大约670万年前(Ma)。反转录转座子亚基因组特异性增殖的长端重复序列分析确定了柳枝稷形成多倍体事件的上界≤4.6 Ma(图2)。gydF4y2Ba1 bgydF4y2Ba),这表明四倍体柳枝稷出现于上新世或更新世早期的冰期-间冰期旋回。gydF4y2Ba
气候适应推动生物质产量gydF4y2Ba
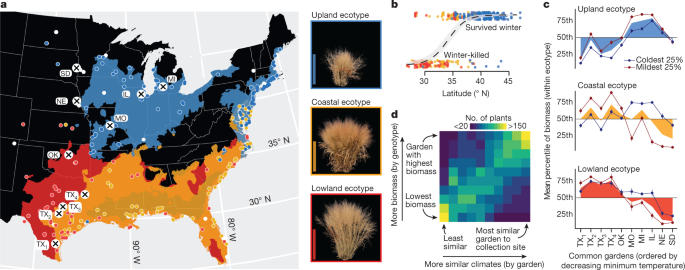
虽然有两个生殖隔离gydF4y2Ba18gydF4y2Ba柳枝稷的细胞型有四倍体(4x)和八倍体(8x),四倍体占多数gydF4y2Ba19gydF4y2Ba而且比八倍体分布的地理范围更广gydF4y2Ba20.gydF4y2Ba。为了研究气候适应、胁迫耐受性和生物量生产的遗传基础,我们建立了732个纯四倍体基因型的多样性面板(补充数据)gydF4y2Ba4gydF4y2Ba).我们无性繁殖和移植到10个普通花园,这些花园横跨1862公里的纬度,从德克萨斯州南部到南达科他州(美国)。gydF4y2BangydF4y2Ba植物gydF4y2Ba= 5,521)。gydF4y2Ba2gydF4y2Ba),并通过深度(中位数= 59×)覆盖2 × 150-bp对端无pcr的Illumina文库对每个基因型进行重测序。重要的是,重测序覆盖范围没有偏向于任何一个亚基因组(似然比检验)gydF4y2BaχgydF4y2Ba2gydF4y2Ba= 1.32,自由度= 1,gydF4y2BaPgydF4y2Ba= 0.25)。我们的重测序获得了3380万个单核苷酸多态性(snp)(次要等位基因频率≥0.5%)与基因组对应。我们还重新组装了这些深度重测序文库的252个基因型子集,并将其称为存在-缺失和结构变异(例如,100-1,500-bp的插入和缺失)。为了将性状和分子变异与气候联系起来,我们提取了46个气候变量gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba从每个基因型的地理参考收集位置,并将这些数据聚类为七个组,这些组解释了多样性面板上的大部分气候变化(扩展数据图)。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba、普通花园的地理分布(gydF4y2BangydF4y2Ba= 10)及植物收集地点(gydF4y2BangydF4y2Ba= 700个地理参考基因型),以及各生态型的空间分布模型。在地图右侧,每种生态型的代表性图像都附有生态型的彩色图例(这些图像是在2019年生长季节结束时拍摄的,背景用ImageJ (gydF4y2Bahttps://imagej.nih.gov/ijgydF4y2Ba)).白色轮廓点(按生态类型着色,如果没有指定生态类型则为白色)表示多样性小组的地理参考采集地点。标有黑色十字的白色圆圈表示10个实验花园的位置。使用rnaturalearthdata R软件包访问公开可用的文化和自然地理信息系统(GIS)层gydF4y2Ba51gydF4y2Ba。比例尺,1米。gydF4y2BabgydF4y2Ba横跨大地,生存(gydF4y2BangydF4y2Ba基因型gydF4y2Ba= 367)及冬杀(gydF4y2BangydF4y2Ba= 184)在北方花园(gydF4y2BangydF4y2Ba= 3)是地理结构的:采集地点起源的纬度预测了生存。逻辑回归预测(±s.e.)伴随着沿纬度预测器的二进制生存率。gydF4y2BacgydF4y2Ba,将估算的生存校正生物量转换为每个生态型的百分位数(0 =最低生物量,100 =最高生物量),并绘制总体平均百分位数(彩色多边形,gydF4y2BangydF4y2Ba= 447)gydF4y2BangydF4y2Ba高地gydF4y2Ba= 211,gydF4y2BangydF4y2Ba沿海gydF4y2Ba= 144,gydF4y2BangydF4y2Ba低地gydF4y2Ba= 92)和花园。30年最冷最低温度(蓝线和点)站点中25%基因型的生物量百分位数(平均值±s.e.m.) (gydF4y2BangydF4y2Ba高地gydF4y2Ba= 52岁gydF4y2BangydF4y2Ba沿海gydF4y2Ba= 35,gydF4y2BangydF4y2Ba低地gydF4y2Ba= 22)及最轻微的25%(红线及点)(gydF4y2BangydF4y2Ba高地gydF4y2Ba= 53岁gydF4y2BangydF4y2Ba沿海gydF4y2Ba= 36,gydF4y2BangydF4y2Ba低地gydF4y2Ba= 23)证明原产地气候影响生态型和整个花园的生物量。gydF4y2BadgydF4y2Ba、气候相似度等级热图(gydF4y2BaxgydF4y2Ba轴)和估算生物量(gydF4y2BaygydF4y2Ba轴)表明,571个基因型中的大多数在气候与其源栖息地相似的普通花园中获得最高的生物量。gydF4y2Ba
柳枝稷的气候相关适应先前被假设为强调北部高地和南部低地生态型之间的差异,并以不同的叶片和整个植物形态为例gydF4y2Ba13gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba。从形态学数据中进行的计算机分类,加上我们的多样性小组中专家的生态类型分配(补充数据)gydF4y2Ba5gydF4y2Ba)、露陆(gydF4y2BangydF4y2Ba= 268),低地(gydF4y2BangydF4y2Ba= 99)和第三种沿海生态型(gydF4y2BangydF4y2Ba= 184)。沿海生态型与低地生态型具有广泛的同域性,但表现出高地叶片特征和低地植物构型(图2)。gydF4y2Ba2gydF4y2Ba扩展数据图gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
我们观察到强有力的证据表明,适应性进化促进了生态型分化。尽管北部高地植物的冬季死亡死亡率很少见(2.4%),但在2018-2019年冬季,在4个最北部的花园中,近一半的沿海(42.1%)和低地(42.8%)基因型死亡。gydF4y2Ba2 bgydF4y2Ba).在西北三个平原地区,冬季死亡尤其严重,可能是由于2019年1月下旬至3月初的一段严寒时期(扩展数据图)。gydF4y2Ba2gydF4y2Ba).来自北部30%人群的基因型总数为218× (Fisher检验优势比= 218.17;gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba)在北部4个地点比最南部30%的基因型更有可能在2018-2019年冬季存活。gydF4y2Ba
在我们共同的花园中,纬度梯度也是生物量变化的主要轴。在7组相关气候变量中,生物量变化的最强预测因子始终与温度相关(扩展数据图)。gydF4y2Ba2gydF4y2Ba).我们观察到极端30年最低温度的强烈信号,作为冬季易被杀死的低地和沿海生态型生物量的预测因子。gydF4y2Ba2摄氏度gydF4y2Ba).对于这两种生态型,从历史极端最低温度较低的地点收集的基因型优于从北部花园中气候较温和的地点收集的基因型。然而,在耐冬杀旱地生态型中,没有观察到依赖于原产地气候的权衡。比2018-2019年更寒冷的冬季可能会在高地基因型中引入差异存活,并产生类似于在两种更南方的生态型中观察到的权衡。这些结果进一步支持了我们的观察,即对低温的敏感性既是自然选择的代理人,也是北方范围扩张的限制因素。gydF4y2Ba
此外,每种基因型的生物量产量通常在与其采集地点最相似的气候条件下达到最大(图2)。gydF4y2Ba二维gydF4y2Ba).因此,局部适应不仅表现在生存和抗逆性上,还表现在与每种基因型进化相似的气候条件下更高的生物量积累上。gydF4y2Ba
基因池间的生态型趋同gydF4y2Ba
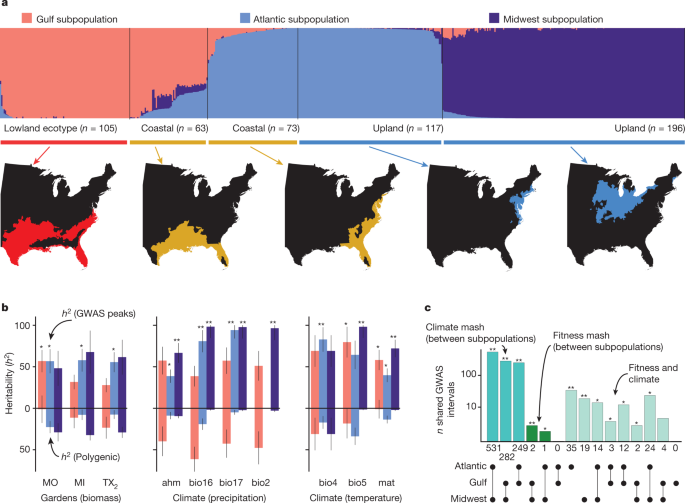
了解柳枝稷基因库的结构和多样性对预测分子育种的未来收益和了解气候适应的遗传基础至关重要gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba。先前对柳枝稷的群体遗传研究认为,群体遗传结构与用于定义生态型的形态聚类之间存在很强的对应关系gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba。对我们的3380万个全基因组SNP数据库的分析显示,我们的多样性面板被强烈地细分为三个主要的遗传亚群,总的来说,地理上是不同的(我们称之为中西部,大西洋和海湾)gydF4y2BaFgydF4y2Ba圣gydF4y2Ba= 0.27)。gydF4y2Ba3gydF4y2Ba).存在-缺失和结构变异的聚类在很大程度上概括了基于snp的亚种群结构(扩展数据图)。gydF4y2Ba3.gydF4y2Ba),提供了亚种群分化的一致证据,其中可能包括几个分子尺度上的大效应突变。gydF4y2Ba
一个gydF4y2Ba,三个基因库(以亚种群着色)和三个生态型(如下所示)之间的混合比例,使用血统同一性矩阵的特征向量分解计算。每个生态型的相应地理分布在条形图下面(用图5中的生态型分布的颜色表示)。gydF4y2Ba1gydF4y2Ba).通过rnaturalearthdata R包访问公开可用的文化和物理GIS层gydF4y2Ba51gydF4y2Ba。gydF4y2BabgydF4y2Ba, snp遗传力事后检验(平均gydF4y2BahgydF4y2Ba2gydF4y2Ba±s.e.m)归因于多基因背景(黑色水平线下方)和显著的多变量适应性收缩(黑色水平线上方)表示了三个主要地点(生物量)和与降水和温度相关的气候变量的GWAS hits(黑色水平线上方),并以亚种群颜色表示(如下)gydF4y2Ba一个gydF4y2Ba).扩展数据图gydF4y2Ba2摄氏度gydF4y2Ba提供气候变量(ahm、bio2、bio4、bio5、bio16、bio17和mat)的描述。GWAS命中相对于双侧多基因遗传的遗传率具有统计学意义gydF4y2BaZgydF4y2Ba分数gydF4y2BaPgydF4y2Ba值;**gydF4y2BaPgydF4y2Ba< 0.001, *gydF4y2BaPgydF4y2Ba< 0.05。gydF4y2BacgydF4y2Ba在大西洋和中西部亚种群中,气候相关的多变量适应收缩(mash)间隔存在较大且显著的重叠,适应度与气候冲击之间存在较小但显著的重叠。双侧费雪检验gydF4y2BaPgydF4y2Ba价值重要性,如下gydF4y2BabgydF4y2Ba。gydF4y2Ba
种群遗传结构与形态生态型差异不一致,在遗传亚群内分化强烈。在大西洋(37%)和中西部(63%)的基因库中均存在旱地生态型性状的植物。同样,54%和46%的沿海生态型加入分别分配给大西洋和海湾亚种群(图2)。gydF4y2Ba3gydF4y2Ba).所有具有低地形态的植物都聚集在海湾亚群中。然而,这些海湾低地植物在北部冬季存活和死亡的个体比例大致相等。gydF4y2Ba2摄氏度gydF4y2Ba).因此,重要的育种遗传多样性存在于遗传亚群中,这种模式通过在几个柳枝稷育种群体中实现生物量和冬季存活率的遗传收益得到了验证gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba。gydF4y2Ba
尽管亚种群之间存在生态型趋同,但coalescent模拟将亚种群的分化时间定在更新世中期(约358,000代(0.7-1.4 Ma,假设世代时间为2-4年))(扩展数据图。gydF4y2Ba3.gydF4y2Ba).因此,现存的柳枝稷基因库在多倍体柳枝稷近一半的进化史中一直处于分化状态。与亚种群之间的深度序列差异相反,我们观察到大西洋亚种群中陆地生态型和沿海生态型之间的分子遗传差异很小(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba= 0.03),或在海湾亚群的低地和沿海生态型之间(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba= 0.03)(扩展数据图gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
混合在海湾和大西洋亚群之间似乎是常见的;比较这两个亚群的沿海生态型特征的植物在分子上更为相似(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba= 0.19)比非沿岸的墨西哥湾和大西洋植物(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba= 0.24)。相比之下,中西部和大西洋亚群中具有高地形态的植物并不比这些亚群中的其他植物更相似(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba两者均= 0.30)。这种高地形态在两个高度分化的遗传亚群中的趋同可能是高地生态型独立遗传起源的结果,也可能是罕见的,但在进化上很重要gydF4y2Ba31gydF4y2Ba掺合料的事件。我们在下面评估这些假设。gydF4y2Ba
提高产量的遗传目标gydF4y2Ba
为了检测多样性面板内气候适应和适应度的遗传基础,我们进行了多变量适应收缩gydF4y2Ba32gydF4y2Ba遗传亚群内部和跨遗传亚群的全基因组关联图谱(GWAS)结果。多变量自适应收缩在单变量测试之间共享GWAS峰值效应大小和方向,以提高检测显著共享结果的能力。确定了适应度GWAS(映射了三个最大的常见花园(MI, MO和TX)的冬季存活率和生物量)的多变量自适应收缩结果gydF4y2Ba2gydF4y2Ba)和气候GWAS(检测SNP变异与起源气候(7个代表性气候变量)之间的关联)。为了在亚种群(具有不同分离snp)之间进行直接比较,我们总结了12239个显著的多变量自适应收缩(log)的连锁不平衡块“峰”gydF4y2Ba10gydF4y2Ba-变换后的贝叶斯因子> 2)gydF4y2Ba33gydF4y2Ba分成10,090个20 kb的区域(20 kb表示连杆不平衡衰减变平的拐点)(扩展数据图。gydF4y2Ba3.gydF4y2Ba)表示气候(gydF4y2BangydF4y2Ba地区gydF4y2Ba= 9,856)及体能(gydF4y2BangydF4y2Ba地区gydF4y2Ba= 332) gwas (gydF4y2Ba补充数据6gydF4y2Ba).候选基因的加权列表-包括假定的SNP效应,存在-缺失或结构变异,基因共表达和GWAS峰的物理接近性-可以在补充数据中找到gydF4y2Ba7gydF4y2Ba。gydF4y2Ba
GWAS峰解释了基因库之间和基因库内部的大部分可遗传表型和气候变异(snp遗传力)。gydF4y2Ba3 bgydF4y2Ba).适合度的单核苷酸遗传率(gydF4y2BahgydF4y2Ba2gydF4y2Ba= 51.5±15.4%(平均值±s.e.m.))和与气候相关的峰值(gydF4y2BahgydF4y2Ba2gydF4y2Ba= 70.5±14.0%)共同解释了比多基因背景(适应度= 19.5±9.1%,气候= 18.2±9.5%)多三倍的变异(扩展数据表)gydF4y2Ba1gydF4y2Ba).这些气候和生物量关联的高遗传力表明,所有变异中一小部分的亲缘关系高于预测的整体亲缘关系,为育种者提供了遗传多样性,以在当地环境中进行柳枝稷改良。gydF4y2Ba
与适应性和原始气候相关的基因座可能与当地适应有关gydF4y2Ba34gydF4y2Ba,是培育本地适应品种的有力目标。总的来说,我们观察到与气候和适应性相关的20 kb区域的重叠几乎是偶然预期的2倍(Fisher检验优势比= 1.92,gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−6gydF4y2Ba).这种重叠在两个北部亚群(中西部,赔率= 11.5×gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba;大西洋,赔率= 17.8倍和gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba)(图。gydF4y2Ba3 cgydF4y2Ba),在那里,我们预计会看到选择对寒冷冬季生存的最大影响。gydF4y2Ba
许多气候和适应度重叠的区域仅在单个遗传亚群中多态,这突出了柳枝稷气候适应的几个可能独立的遗传途径。然而,9.5%(940)的20 kb气候区间在多个遗传亚群中存在多态性。鉴于海湾和大西洋亚种群之间混合的大量证据(图2)。gydF4y2Ba3gydF4y2Ba),我们预计当代基因流动将是共享多态性的主要贡献者。与此假设相反,所有多亚群GWAS区间的大多数(511个区域)在两个遗传差异最大的基因库(大西洋和中西部)之间共享。考虑到这些亚种群之间的深度分化时间,罕见或古老的基因流动gydF4y2Ba35gydF4y2Ba可能创造了这些共享的适应性多态区域。gydF4y2Ba
通过渗入的进化趋同gydF4y2Ba
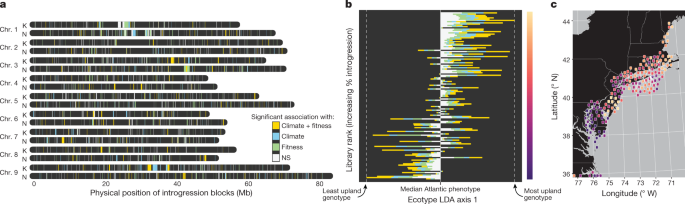
为了明确说明基因渗入如何影响气候snp关联的分布,我们使用隐马尔可夫模型研究了基因组中物理上连续的混合区域gydF4y2Ba36gydF4y2Ba。亚群间的基因渗入占我们重测序基因组内容的2.98%(图2)。gydF4y2Ba4gydF4y2Ba),但在亚群中包含共享GWAS间隔的可能性比偶然预期的高1.5倍(Fisher检验比值比= 1.55,gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−8gydF4y2Ba),这表明适应性基因渗入至少是亚种群间共享的部分遗传变异的基础。gydF4y2Ba
一个gydF4y2Ba, bbbb10基因型中所有高频基因的位置;gydF4y2BangydF4y2Ba= 1,640)从中西部向大西洋亚群的渗透在两个气候冗余分析中以显著性着色(蓝色,gydF4y2BangydF4y2Ba= 234),生物量和存活率(绿色,gydF4y2BangydF4y2Ba= 329)或“气候适应性重叠”,这在两者中都很重要(黄金,gydF4y2BangydF4y2Ba= 245)。NS,不显著。gydF4y2BabgydF4y2Ba在135个基因型中,基因渗入与较高的高地表型密切相关。对于每个基因型,沿生态型之间的第一个区别轴的位置(扩展数据图)。gydF4y2Ba2gydF4y2Ba)相对于大西洋生态型的中位数值进行缩放,然后根据每个显著性bin中渗入序列的比例进行绘图和着色gydF4y2Ba一个gydF4y2Ba。gydF4y2BacgydF4y2Ba,渗透从gydF4y2BabgydF4y2Ba被转换成紫橘色刻度(右gydF4y2BabgydF4y2Ba),并在美国北大西洋沿岸绘制了每个图书馆的收藏地点的地理参考位置。通过rnaturalearthdata R包访问公开可用的文化和物理GIS层gydF4y2Ba51gydF4y2Ba。gydF4y2Ba
特别令人感兴趣的是从中西部到大西洋亚群的一系列渗透,可追溯到距今约8700代(17 - 34000年前),这与末次冰期极大期(约22 ka)后的北部范围扩张相吻合。具有较高中西部基因渗入水平的大西洋基因型表现出更多的高原性状(图2)。gydF4y2Ba4 bgydF4y2Ba),并且在大西洋亚种群的其他亚热带和温带范围的北缘被过度代表(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).与其他系统中基因组渗入的适应性作用一致gydF4y2Ba31gydF4y2Ba,gydF4y2Ba37gydF4y2Ba这些发现表明,假定的北方适应性等位基因从中西部进入大西洋亚群,可能促进了美国东北沿海地区较冷栖息地的柳枝稷在冰川后的殖民。为了验证这一假设,我们进行了冗余分析,将渗入块的存在与气候、地理和表型因素联系起来。总体而言,大西洋亚群中的中西部基因渗入与气候(解释方差百分比= 46.5%)的相关性是地理(11.5%)的四倍多。虽然从中西部到大西洋亚群的532和651个渗透分别与原始气候或生物量有关,但254个渗透在两个分析中都是异常值,这代表了每组之间独立性预期的近7倍富集(优势比= 6.99)。gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba).这些结果强化了中西部基因渗入塑造了北大西洋基因型的气候生态位和表型分布的假设,并支持了越来越多的证据表明适应性基因渗入可以促进范围扩大和生态型进化gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba。gydF4y2Ba
显性亚基因组遗传力降低gydF4y2Ba
多倍体在开花植物谱系中是常见的,可以增加可供选择的遗传多样性gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba这可能导致适应性进化或分选,从而改变生态位特征gydF4y2Ba42gydF4y2Ba。这一过程可以解释多倍体在极地纬度和高海拔地区普遍存在的原因,这些地区在冰川循环期间曾被冰盖覆盖gydF4y2Ba43gydF4y2Ba。gydF4y2Ba
在多倍体形成过程中复制的基因可以亚功能化(将祖先基因的功能划分到旁系基因中)、新功能化(为旁系基因进化出新的基因功能)或简单地丢失gydF4y2Ba44gydF4y2Ba。在多倍体物种形成之后,一个亚基因组通常保留更多的基因,并且平均表现出比另一个亚基因组更高的表达水平,这种现象被称为亚基因组优势gydF4y2Ba45gydF4y2Ba。和其他多倍体一样gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba在柳枝稷中,亚基因组显性和亚功能化表现明显。与N亚基因组相比,K亚基因组具有更高的基因密度(77.4个基因/ Mb比68.0个基因/ Mb)gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba),更多的上调基因(5445对4477,二项gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba)和较低的突变积累率(K亚基因组中有5255个基因具有同义突变率(gydF4y2BaKgydF4y2Ba年代gydF4y2Ba)大于N亚基因组,而N亚基因组中有6,751个基因gydF4y2BaKgydF4y2Ba年代gydF4y2Ba大于二项的K亚基因组gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−15gydF4y2Ba).综合起来,我们所有的11个亚基因组统计(扩展数据图)。gydF4y2Ba4gydF4y2Ba)指出了K亚基因组更强的进化约束和偏向,这表明适应进化的潜力可能在亚基因组之间被不同地划分。gydF4y2Ba
考虑到进化倾向于保留K亚基因组,我们预计会看到更强的气候适应信号gydF4y2Ba44gydF4y2BaK亚基因组上的snp对生物量和存活率的影响。相反,在10个常见花园中,75.9%的生物量snp遗传力可归因于N亚基因组,而只有24.1%可归因于K亚基因组(扩展数据图)。gydF4y2Ba4gydF4y2Ba).此外,在N亚基因组上发现了54.3%的中西部向大西洋亚种群的渗透,这是一个显著的富集(二项检验)gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba−7gydF4y2Ba),即使校正了7.5%的N亚基因组扩增(二项检验)gydF4y2BaPgydF4y2Ba= 0.0012)。归因于N亚基因组的大量渗入和可遗传的生物量变化可能与亚基因组对K亚基因组的进化倾向不一致。对于这一违反直觉的发现,一种可能的解释是,N亚基因组上放松的进化约束(减少净化选择)可能允许通过定向或多样化选择积累适应性遗传变异。因此,N亚基因组积累了可遗传的变异gydF4y2Ba49gydF4y2Ba未来的育种制度可以以塑造天然柳枝稷种群和提高生物燃料产量为目标。gydF4y2Ba
讨论gydF4y2Ba
随着气候和自然环境的变化,对驯化物种的遗传改良期望和野生种群的适应潜力进行限定变得越来越重要gydF4y2Ba50gydF4y2Ba。事实上,植物基因组可以让我们窥见农作物和野生植物种群的过去和未来。对冰期-间冰期循环的适应为当前和未来的环境变化提供了一个有指导意义的模拟,我们在这里探讨了过去、现在和未来柳枝稷气候适应和产量提高的基因组机制。gydF4y2Ba
然而,植物基因组的复杂性也阻碍了遗传资源的开发,不利于快速有效的分子育种。我们的方法和成功测序柳枝稷复杂的基因组将促进生态和农业基因组学在几乎任何系统。例如,我们的研究结果表明,对北方气候的适应是由古代分化亚群之间的基因渗入促进的,这进一步支持了不同基因组之间的混合可以增强对新环境的适应的假设gydF4y2Ba37gydF4y2Ba。这种适应性渗入和可遗传的亚基因组特异性遗传变异gydF4y2Ba49gydF4y2Ba可能提供最小阻力的遗传路径,允许在环境变化期间在新栖息地定居。专性异交和多倍体——在选择基因组研究系统时经常有意识地避免的性状——是柳枝稷在自然界适应的主要驱动因素,也是遗传变异的来源,可用于选择,以通过不断变化的未来提高生物燃料产量。gydF4y2Ba
方法gydF4y2Ba
未采用统计学方法预先确定样本量。实验是完全随机的,研究人员在进行实验或测序时不知道基因型标识符。gydF4y2Ba
植物采集、繁殖、培养和表型分析gydF4y2Ba
为了形成多样性面板,从2010年到2018年收集了天然和普通园林来源的种子、根茎和无性系繁殖体。从种子长成的植物遵循标准的生长程序gydF4y2Ba16gydF4y2Ba。简而言之,将10-15颗种子播种在9厘米的方形花盆中,其中含有ProMix BX盆栽土壤(Premier Tech Horticulture)和Turface MVP煅烧粘土(Turface Athletics)的混合物,并在4°C下春化7天。然后将花盆放置在光照的温室中,白昼长度为14小时,昼夜温度为30°C/22°C。幼苗在3叶期减薄至每罐1株,并让其生长至5分蘖期。根茎繁殖体和5分蘖苗被转移到5加仑的盆中,盆中含有细磨松皮地膜(Lone Star mulch)和缓释肥料(Osmocote 14-14-14, ScottsMiracleGro)。所有单株于2016 - 2018年通过无性系分裂在奥斯汀进行繁殖,每一株的繁殖目标为10个无性系。Cleary 3336F系统杀菌剂(Cleary Chemicals)在必要时施用于植物以控制真菌病原体。植物被放置在1加仑的花盆中进行最后的繁殖。gydF4y2Ba
2018年5月15日至7月10日在田间进行了种植,并遵循了先前公布的方法gydF4y2Ba16gydF4y2Ba。简而言之,植物用卡车运送到每个地点,每个地点都覆盖着一层德威特除草布。植物被放置在杂草布上的洞中,这些洞被切割成蜂窝状,每个植物都有四个最近的邻居,彼此之间的距离为1.56米。为了防止边缘效应,在每个边缘位置都种植低地黑井品种。植物移植后用手浇水。所有植株的地上部分在2018-2019年冬季被保留,并在2019年春季分蘖出苗之前被移除。在2019年的季节结束时,植物被绑成一束,用镰刀割草机收割。gydF4y2Ba
我们生成了2019年生长季节的两个适应度指标:原木转化生物量(kg)和冬季存活率(kg)。gydF4y2Ba补充数据8gydF4y2Ba).生物量数据是在2019年10月和11月收获期间从所有活个体中获得的。将估计质量<750 g的植物放在纸袋中,在60°C下干燥整株,直到没有额外的水分流失,然后称重总干生物量。在田间用±5克分辨率的悬挂标尺对估计质量为b> 750 g的植物进行湿生物量称重。为了确定这些植物的生物量,从每棵植物中提取约500克整株分蘖,称重,如上所述干燥并重新称重。然后将整个植物样品的湿生物量乘以子样品中的水分百分比,以近似总干生物量。在2018-2019年冬季,植物被认为经历了冬季死亡,因为到2019年6月1日,植物冠没有出现新的生长。2019年7月或9月,从实验中切除了死亡的植物冠,代之以布莱克威尔(Blackwell)品种的植物。gydF4y2Ba
基因组组装和抛光gydF4y2Ba
我们在美国能源部联合基因组研究所和哈德逊alpha生物技术研究所使用全基因组霰弹枪测序策略和标准测序方案对Alamo柳枝稷AP13基因型进行了测序。基因组由4,520,785 PacBio reads组装和抛光(121.66×原始序列覆盖范围来自59个P6C4 2.0和2.1化学细胞,电影时间为10小时,p-read产量为91.76 Gb)(扩展数据图)。gydF4y2Ba1gydF4y2Ba),使用MECAT组装器gydF4y2Ba52gydF4y2Ba和ARROW抛光机gydF4y2Ba53gydF4y2Ba。最后用一个400 bp插入2 × 150 bp Illumina HiSeq片段文库(177.1×)进行基因组抛光和错误校正。读取>95%的简单序列重复,并在调整适配器和质量后读取<50 bp (gydF4y2Ba问gydF4y2Ba< 20,5 -bp窗口平均)被去除。最终的reads集包括1,259,053,614个reads,总共覆盖了168倍的高质量Illumina碱基。这产生了6600个支架(6600个contigs)的初始二倍体组装,其中contigs NgydF4y2Ba50gydF4y2Ba1.1 Mb,大于100 kb的支架有3489个,总2C(二倍体)基因组大小为2,013.4 Mb。gydF4y2Ba
在近交个体中组装单倍体基因组,如AP13,通常会在杂合区域产生两个单倍体拷贝,这就需要计算步骤来将每条染色体表示为单拷贝单倍体,而不会不必要地重复重复副本。我们的初始组装大约是预期的单倍体(1C)基因组大小1.2 Gb的两倍。因此,为了检测假设的减数同源单倍型,我们鉴定并计数了恰好在组装中出现两次的共享24-mers,并相应地将其分类。共鉴定出3,152个较短和冗余的备选单倍型和2,387个重叠的contich末端,包括871.2 Mb的总序列。剩余的1,142.2 Mb序列通过比对2个可用图谱的遗传标记被排序并定向到18个染色体上(补充数据)gydF4y2Ba1gydF4y2Ba)出席MECAT大会;563个连接和57个断裂,10000个Ns代表未大小的间隙序列。总的来说,97.2%的组装序列包含在染色体中。端粒序列用(TTTAGGG)gydF4y2BangydF4y2Ba重复并正确定位。剩余的支架针对GenBank细菌蛋白和细胞器序列进行筛选,如果发现与这些序列匹配,则将其去除。为了解决小重叠区域上的contig末端,相邻的contig末端对齐到另一个使用BLATgydF4y2Ba54gydF4y2Ba;共折叠了47个相邻的重复组对。gydF4y2Ba
我们进行了两轮纠错。首先,我们通过使用bwa mem将Illumina 2 × 150 bp文库与释放共识序列对齐来纠正纯合snp和插入和/或缺失(indels)gydF4y2Ba55gydF4y2Ba并用GATK的统一基因型工具鉴定纯合snp和indelgydF4y2Ba56gydF4y2Ba。共更正了690个纯合snp和80199个纯合索引。其次,我们使用ABI 3730XL毛细管测序仪组合计算完成了来自BAC克隆的11,343个组装contigs的测序gydF4y2Ba57gydF4y2Ba和单指数Illumina克隆池,并将这组柳枝稷克隆与固定snp的基因组进行比对,以找到与其相邻的异相杂合snp。为了解决这些相位开关等位基因,将完整的原始PacBio reads与组装对齐。对于每次读取,确定每个杂合位点的期相,并纠正62,732个异期杂合位点。gydF4y2Ba
为了区分N和K亚基因组,我们使用了从头重复聚类方法,并通过与相关物种的系统发育距离验证了这一点。我们通过水母搜索“诊断”15-mersgydF4y2Ba58gydF4y2Ba在吉普赛,Copia和Pao插入的LTR区域(由RepeatMasker识别)gydF4y2Ba59gydF4y2Ba和LTRHarvestgydF4y2Ba60gydF4y2Ba),以区分每组同源染色体(一个同源染色体≤1命中,另一个同源染色体≥100命中)。具有共同15-mers的LTR序列被分组为超家族,并通过BLAST在每个超家族中进行比对。具有重大BLAST命中的超家庭成员(gydF4y2BaegydF4y2Ba< 0.01,≥90%长度)按Mafft分类并对齐gydF4y2Ba61gydF4y2Ba。LTR家族之间的Jukes-Cantor距离由R ape包计算gydF4y2Ba62gydF4y2Ba,并聚集成两组不同的亚基因组。LTRs和比对的聚类是相同的gydF4y2Bap . rudgeigydF4y2Ba(K.M.D.和E. Kellogg,未发表的数据),它是K亚基因组的古代亲戚gydF4y2Ba17gydF4y2Ba这给了我们很高的信心,我们已经有效地将所有染色体分配到正确的亚基因组中。最后,我们分配了染色体标识符,并通过与之合成来定位每条染色体伪分子gydF4y2BaSetaria italicagydF4y2Ba63gydF4y2Ba。最终发布的单倍体5.0版本包含1125.2 Mb的序列,包含626个contigs,其中contigs N50为5.5 Mb,染色体中组装碱基的总数为97.2%。gydF4y2Ba
基因注释gydF4y2Ba
转录本组装由约20亿对2 × 150-bp的双链Illumina RNA-seq reads、约10亿对2 × 100-bp的双链Illumina RNA-seq reads和454个reads (gydF4y2Ba补充数据3gydF4y2Ba)使用PERTRAN(其详细信息先前已发布)gydF4y2Ba64gydF4y2Ba).简而言之,PERTRAN通过GSNAP进行基因组引导的转录组短读组装gydF4y2Ba65gydF4y2Ba并对拼接进行对齐验证、重新对齐和校正后,构建拼接对齐图。总共约有450万个PacBio Iso-Seq循环共识序列gydF4y2Ba66gydF4y2Ba被纠正并崩溃,产生大约677,000个假定的全长转录片段。随后,使用PASA构建了668,176个转录片段gydF4y2Ba67gydF4y2Ba从RNA-seq reads,全长cDNA, Sanger表达的序列标签,以及校正和折叠的PacBio循环共识序列reads。基因座由EXONERATE测定gydF4y2Ba68gydF4y2Ba柳枝稷转录物组装和蛋白质的比对gydF4y2Ba拟南芥gydF4y2Ba69gydF4y2Ba、大豆gydF4y2Ba70gydF4y2Ba北竹大米gydF4y2Ba71gydF4y2Ba,gydF4y2BaSetaria冬青gydF4y2Ba72gydF4y2Ba,gydF4y2Bap . halliigydF4y2Bavar。gydF4y2BahalliigydF4y2Ba64gydF4y2Ba,gydF4y2Ba高粱二色的gydF4y2Ba73gydF4y2Ba,gydF4y2BaBrachypodium distachyongydF4y2Ba74gydF4y2Ba,葡萄和瑞士普罗特gydF4y2Ba75gydF4y2Ba蛋白质组。使用RepeatMasker对重复软屏蔽柳枝稷基因组完成这些比对gydF4y2Ba59gydF4y2Ba(重复库从RepeatModelergydF4y2Ba76gydF4y2Ba和RepBasegydF4y2Ba77gydF4y2Ba),除非延伸到同一条链上的另一个位点,否则两端的长度可达2,000 bp。不完整的基因模型,即同源性低但转录组不完整的基因模型,或短的单外显子基因(<300 bp的编码DNA序列(CDS)),没有蛋白结构域或良好的表达。gydF4y2Ba
比较基因组学gydF4y2Ba
通过GENESPACE管道推断出两个柳枝稷亚基因组的同源同源物和旁同源物gydF4y2Ba64gydF4y2Ba,使用默认参数和两个outgroups:gydF4y2Bap . halliigydF4y2Bavar。gydF4y2BahalliigydF4y2Ba64gydF4y2Ba和gydF4y2Ba美国二色的gydF4y2Ba73gydF4y2Ba。简而言之,GENESPACE将蛋白质相似度分数解析为合成块并运行正交器gydF4y2Ba78gydF4y2Ba关于synsynconstrained爆炸结果。由此产生的块坐标和合成正交网络为进化推理提供了高置信度的锚点。gydF4y2Ba
为了计算CDS区域的祖先状态,我们首先使用来自Phytozome的基因组确定了具有共同祖先的序列gydF4y2Ba79gydF4y2Ba。柳枝稷基因组的最终匹配数分别为38,960和33,772gydF4y2Bap . halliigydF4y2Ba,gydF4y2Ba美国二色的gydF4y2Ba,分别。对于任意给定的正交网络,我们在matt中建立了两个多序列比对gydF4y2Ba61gydF4y2Ba一个不包括焦点柳枝稷序列(msa)gydF4y2Ba0gydF4y2Ba)和一个强迫msagydF4y2Ba0gydF4y2Ba通过——keeplength参数对准焦序列的坐标系。然后用最大似然算法在Phangorn中提取边缘特征状态gydF4y2Ba80gydF4y2Ba。对于每次重建,只使用最接近柳枝枝条的内部节点作为祖先状态。总体而言,我们分析了40,943个柳枝稷基因模型(216,157外显子),覆盖54.95 Mb (gydF4y2Ba补充数据9gydF4y2Ba).gydF4y2Ba
亚基因组进化和年代测定gydF4y2Ba
为了推断柳枝稷亚基因组和四倍体柳枝稷的年龄,我们采用了一组保守的同源物,它们之间具有简单的2:1:1网络gydF4y2Bap . virgatumgydF4y2Ba,gydF4y2Bap . halliigydF4y2Ba和gydF4y2Ba美国italicagydF4y2Ba。这产生了45,045个柳枝稷蛋白,排列到24,549gydF4y2Bap . halliigydF4y2Ba通过跨物种分析,得到20,496对同源子和4,053个单子(K亚基因组2,396个,N亚基因组1,660个)。我们使用Dialign-TX对这些序列的翻译cd进行比对gydF4y2Ba81gydF4y2Ba。将排列好的CDS序列连接到Gblocks中gydF4y2Ba82gydF4y2Ba使用默认参数。Gblocks将18,044,244个CDS核苷酸筛选到16,321,302个位置,在50,334个区块中。然后在PhyML中使用结果对齐gydF4y2Ba83gydF4y2Ba利用一般时间可逆模型构建最大似然树。这棵树被用作r8s的输入gydF4y2Ba84gydF4y2Ba,计算时间树并校准gydF4y2Ba黍gydF4y2Ba- - - - - -gydF4y2BaSetariagydF4y2Ba树的节点为13.1 MagydF4y2Ba63gydF4y2Ba。到目前为止,亚基因组分化和多倍体柳枝稷物种形成的时间,我们利用了爆发距离,它指的是LTR家族内的所有距离(而成对距离指的是相同插入的5 '和3 ' LTR之间的距离)。N-或k -亚基因组特异性反转录转座子的5 '和3 '距离用于确定这些元件的插入时间。此方法不能用于gydF4y2Bap . virgatumgydF4y2Ba特殊技能或gydF4y2Ba黍gydF4y2Ba因为这些元素最近的扩展主导了分布。相反,我们依靠比较最佳的跨物种比对来估计物种的LTR距离gydF4y2Bap . virgatumgydF4y2Ba- - - - - -gydF4y2Bap . halliigydF4y2Ba和gydF4y2Ba黍gydF4y2Ba- - - - - -gydF4y2BaSetariagydF4y2Ba节点。这样,我们就有了校准点,可以将LTR距离与物种之间更可靠的蛋白质编码基因差异进行比较。gydF4y2Ba
亚功能化和基因表达分析gydF4y2Ba
为了评估在蛋白质编码序列尺度上观察到的亚基因组进化偏差是否在表型中表现出来,我们探索了来自AP13叶片组织的生物复制同源物之间的基因表达偏差(gydF4y2BangydF4y2Ba≥5)在两个地点(TXgydF4y2Ba2gydF4y2Ba和MI)。对Illumina配对末端RNA-seq 150-bp reads进行质量修剪(gydF4y2Ba问gydF4y2Ba≥25),修剪后长度小于50 bp的被丢弃。对高质量序列进行比对gydF4y2Bap . virgatumgydF4y2Bav5.1参考基因组使用GSNAPgydF4y2Ba65gydF4y2Ba使用HTSeq v.0.11.2获得与被注释基因唯一对应的reads计数gydF4y2Ba85gydF4y2Ba。在DESeq2中通过似然比检验进行差异表达检验gydF4y2Ba86gydF4y2Ba。在按亚基因组拆分reads之前计算文库大小;这些大小被用作分析差异表达的大小因子。从似然比检验中,亚功能化被定义为显著的亚基因组与环境的相互作用。利用条件下亚基因组间的事后wald测试对比,对田间花园和注释库的亚基因组表达偏差进行了测试。显著偏倚定义为差异表达假发现率调整gydF4y2BaPgydF4y2Ba< 0.05。用WGCNA对AP13基因注释RNA-seq文库进行加权基因共表达聚类gydF4y2Ba87gydF4y2Ba6的幂。原始计数可在gydF4y2Ba补充数据10gydF4y2Ba。gydF4y2Ba
倍性评估gydF4y2Ba
我们使用LSRFortessa SORP流式细胞仪(BD Biosciences)来测定重测序材料的倍性水平。每株取200-300 mg幼叶组织,用刮片浸泡在培养皿中,用1 ml Cystain PI Absolute P核提取缓冲液(Sysmex流式细胞术)与1 μl 2-巯基乙醇混合处理15 min。用CellTrics 30 μm滤器(Sysmex)过滤样品,分离游离细胞核,用2 ml Cystain PI Absolute P染色缓冲液(Sysmex)、12 μl碘化丙啶和6 μl RNase a在湿冰上处理20 min,流式细胞仪测定细胞核大小,每个样品至少分析10,000个细胞核。流式细胞仪的输出用FlowJo软件(BD Biosciences)进行分析,并根据每个细胞核的平均荧光单位将样品分为三类(补充图2)。gydF4y2Ba1gydF4y2Ba).如果细胞群有4 - 8万单位的荧光,则认为样品的倍性水平为4×, 8 - 10万单位为6×, 10 - 14万单位为8×。用流式细胞术数据建立分仓参数gydF4y2Bap . virgatumgydF4y2Ba已知倍性的材料。gydF4y2Ba
我们还通过双等位基因snp的变异等位基因频率分布来评估样本的倍性(如“变异召唤”所述)。该方法假设四倍体和八倍体遵循不同的等位基因频率分布模式,四倍体为0.5/0.5(参考深度和变异深度),八倍体混合为0.75/0.25和0.5/0.5。如果命中的比例为0.48≤gydF4y2BaxgydF4y2Ba≤0.52 <0.035,认为文库为八倍体;≥0.035,认为文库为四倍体;870个样本中有837个(96.2%)的流式细胞术数据与这些结果相匹配。gydF4y2Ba
变量调用gydF4y2Ba
共对789份四倍体多样性样本进行了重测序,中位深度为59×(范围20×-140×)。其中,732份在过滤了缺失数据、异常杂合度升高和采集位点差异后用于进一步分析。样品采用HudsonAlpha生物技术研究所和联合基因组研究所的Illumina HiSeq X10和Illumina NovaSeq 6000对端测序(2 × 150 bp)进行测序。考虑到不同的库大小,读取被修剪到≤50倍的覆盖率,然后使用bwa-mem映射到v5程序集gydF4y2Ba55gydF4y2Ba。gydF4y2Ba
通过将Illumina reads与AP13参考文献与BWA-mem对齐来调用snp。生成的.bam文件使用Picard (gydF4y2Bahttp://broadinstitute.github.io/picardgydF4y2Ba),并使用GATK 3.0重新排列索引gydF4y2Ba56gydF4y2Ba。多样本SNP调用使用SAMtools堆叠完成gydF4y2Ba88gydF4y2Ba和Varscan V2.4.0gydF4y2Ba89gydF4y2Ba最小覆盖率为8个,最小交替等位基因数为4个。通过二项检验来确定基因型。在24mer重复序列的25bp内的snp从进一步的分析中删除。仅保留了数据缺失率≤20%和较小等位基因频率>0.005的snp,导致75%的基因组中有33,905,042个snp,覆盖深度在8×至500×之间。使用SHAPEIT3进行分相gydF4y2Ba90gydF4y2Ba。gydF4y2BaFgydF4y2Ba圣gydF4y2Ba计算通过vcftools完成gydF4y2Ba91gydF4y2Ba。我们通过生成732个文库和18条染色体每Mb的平均覆盖率来测试亚基因组读图偏差。然后,我们在lme4中对这些数据拟合了一个混合效应线性模型gydF4y2Ba92gydF4y2Ba其中染色体数目(1-9)是随机效应,以检验亚基因组的主要效应。有和没有主效应项的模型通过似然比检验进行比较。gydF4y2Ba
使用HipMer构建了732个短读库的单独从头组装gydF4y2Ba93gydF4y2Ba与一个gydF4y2BakgydF4y2Ba-mer的大小为101,以最大限度地在contigs之间进行单倍型分裂。由于组装体的质量和相邻性各不相同,根据组装体的总大小,将基因存在缺失和结构变异检测的样本集缩小到251个样本(泛基因组集)gydF4y2Ba50gydF4y2Ba每个文库的长度和总基因比对。gydF4y2Ba
为了评估整个泛基因组中基因的存在-缺失变异,我们将所有AP13蛋白和一组独特的6161个来自美国的蛋白质进行了比对gydF4y2BaOropetium thomaeumgydF4y2Ba(gydF4y2BangydF4y2Ba蛋白质gydF4y2Ba= 1476)gydF4y2Ba94gydF4y2Ba,gydF4y2Ba美国italicagydF4y2Ba(gydF4y2BangydF4y2Ba= 1085)gydF4y2Ba63gydF4y2Ba,gydF4y2BaSetaria冬青gydF4y2Ba(gydF4y2BangydF4y2Ba= 891)gydF4y2Ba72gydF4y2Ba,gydF4y2Bap . halliigydF4y2Bavar。gydF4y2Ba菲利普gydF4y2Ba(gydF4y2BangydF4y2Ba= 1048)gydF4y2Ba64gydF4y2Ba,gydF4y2Ba美国二色的gydF4y2Ba(gydF4y2BangydF4y2Ba= 878)gydF4y2Ba95gydF4y2Ba和gydF4y2Bap . halliigydF4y2Bavar。gydF4y2BahalliigydF4y2Ba(gydF4y2BangydF4y2Ba= 772)gydF4y2Ba64gydF4y2Ba。这些独特的基因是从通过正交器推断的单拷贝同源网络中提取出来的gydF4y2Ba78gydF4y2Ba和选择由于缺乏同源的柳枝稷。使用BLAT将所有蛋白质(≥100个氨基酸)与所有从头组装对齐gydF4y2Ba54gydF4y2Ba。如果AP13蛋白的基因序列大于或等于80%的同源性和75%的覆盖率,则认为存在,而其他草蛋白的基因序列大于70%的同源性和75%的覆盖率(允许物种之间更大的差异)。可变(泛基因组外壳)基因(被认为存在于40-60%的人群中);gydF4y2BangydF4y2Ba= 5,432)从存在-缺失变异矩阵中提取,并用于可视化来自大西洋,海湾和中西部亚群的非混合个体之间的差异。在每个亚群中检测显著过度或不足代表的基因是用agydF4y2BaχgydF4y2Ba2gydF4y2Babenjamin - hochberg多重测试修正(gydF4y2BaPgydF4y2Ba≤0.05)。gydF4y2Ba
为了检测整个泛基因组的结构变异,使用nglr将每个文库中的contigs(≥2kb)与AP13参考基因组进行比对gydF4y2Ba96gydF4y2Ba使用PacBio读取的默认设置。使用samtools对生成的.bam文件进行排序gydF4y2Ba88gydF4y2Ba用来用鼻子召唤结构变体gydF4y2Ba96gydF4y2Ba。使用SURVIVOR跨样本合并单个结构变体调用gydF4y2Ba97gydF4y2Ba,最大允许距离为1 kb。使用bcftools过滤得到的.vcf文件gydF4y2Ba88gydF4y2Ba最小等位基因频率为0.1,只考虑长度在100 - 1500 bp之间的插入和缺失。gydF4y2Ba
人口基因组学gydF4y2Ba
评价732份四倍体文库的遗传群体结构(补充资料)gydF4y2Ba4gydF4y2Ba),我们提取了所有具有祖先状态调用的四重退化位点(相对中性)(补充数据)gydF4y2Ba9gydF4y2Ba)从祖先的状态排列。该位点列表代表我们的最高置信度中性位点,然后使用|的阈值对链接不平衡进行修剪gydF4y2BargydF4y2Ba|≤0.6,得到59,789个位点用于R包SNPRelate的下游分析gydF4y2Ba98gydF4y2Ba。gydF4y2Ba
群体的连锁不平衡程度由snp决定gydF4y2Ba99gydF4y2Ba在叮铃声gydF4y2BaOne hundred.gydF4y2Ba。连杆不平衡(gydF4y2BargydF4y2Ba2gydF4y2Ba)使用plink(——ld-window 500——ld-window-kb 2000)计算。的gydF4y2BargydF4y2Ba2gydF4y2Ba每500bp取平均值。在R中使用nls函数对该数据拟合了一个非线性模型,并确定了连杆不平衡(gydF4y2BargydF4y2Ba2gydF4y2Ba非线性曲线稳定。gydF4y2Ba
群体遗传结构分级评价。考虑到研究范围内存在高度分化的生态型,我们首先使用主成分判别分析(DAPC)分析了最广泛的遗传群体结构。gydF4y2Ba101gydF4y2Ba在adegenet v.2.0.1中gydF4y2Ba102gydF4y2Ba。这种方法不依赖于构成许多种群聚类方法基础的常见假设(例如,Hardy-Weinberg均衡和连锁不均衡),因此提供了一种有价值的工具来研究广泛的结构划分。DAPC展示了一套强大的基因库,并将中西部基因型与所有其他基因型区分开来。然后,我们使用structure v.2.3.4中实现的贝叶斯聚类算法评估了剩余非中西部个体的遗传群体结构和潜在混合gydF4y2Ba103gydF4y2Ba通过相关等位基因频率的混合模型。该分析包括20,000个燃烧步骤和30,000个1-6个基因型组的重复,每个重复运行10次。通过SNPRelate的特征向量分解,对所有亚种群的祖先系数进行事后分配。gydF4y2Ba
利用多序列马尔可夫聚结法(MSMCv.2.0)推断柳枝稷样本的人口统计学历史gydF4y2Ba104gydF4y2Ba),这是一种种群遗传学方法,用于从序列数据中推断人口历史和种群结构。该方法模拟了重组下聚结线虫的近似模型,并对种群大小和分化时间进行了检验。每个亚群使用4个单倍型运行MSMC,跳过模糊位点,估计rhoOverMu为0.25,时间段模式为10 × 2 + 20 × 5 + 10 × 2。我们对每个亚群的5组4个单倍型进行了100次迭代,在没有固定重组参数的情况下,估计rhoOverMu的平均值为0.25,并取其平均值。为了估计世代间的尺度分化时间,我们假设突变率为6.5 × 10gydF4y2Ba−8gydF4y2Ba。为了估计初始分化时间,我们比较了相邻的相对交叉聚结率(RCCR)值(从过去到现在)(gydF4y2Ba补充数据11gydF4y2Ba).如果在单个时间段内或在相邻时间段内或在两个交错时间段内出现下降(>0.01;观测范围为0.01 ~ 0.28),以下相邻值几乎为零(≤0.009;观测范围:−0.1-0.009),我们认为这是种群分离的起点。然而,如果在五个时间段内再次出现下降,我们认为后者是人口分离的开始。我们用16组不同的个体对每个亚群进行了重复分析。gydF4y2Ba
通过对距离矩阵的特征向量分解,对snp、结构变异和有无变异的种群结构进行了可视化分析。首先,计算3种变异呼叫类型的0/1/2(参考纯合子、杂合子、替代纯合子)文库×标记矩阵之间的欧氏距离矩阵。然后对欧几里得矩阵进行缩放和集中,通过在R包MDMR中实现的Gower中心相似矩阵去除库间覆盖率方差gydF4y2Ba105gydF4y2Ba。gydF4y2Ba
生态型分类gydF4y2Ba
对成熟柳枝稷开花期或临近开花期的植株进行了16项性状调查(叶片长度、宽度、长宽比、面积、层厚和层/中脉厚度比;整株:分蘖数、分蘖高度、分蘖高度×数乘积、分蘖高度/计数比、穗高、穗高/计数比、叶冠高度、分蘖/叶高比;物候学:变绿日期和穗长出日期),以确定2019年夏季在德克萨斯大学j.j. Pickle研究校园(PKLE;或TXgydF4y2Ba2gydF4y2Ba(Austin, Texas, USA)和密歇根州立大学Kellogg生物站(KBSM;或MI (Hickory Corners, Michigan, USA))普通花园(见补充资料)gydF4y2Ba5gydF4y2Ba获取这些变量的详细描述)。物候测量,包括变绿(当第一个绿色营养结构从根茎冠出现时)和穗出苗(当第一个生殖结构从分蘖出现时),每天进行分析。通过测量叶片的长度和宽度(单位:mm)、中脉和叶片厚度(单位:μm) (Mitutoyo 547-500S卡尺)和叶面积(单位:mm),对每个植物的代表性叶片进行了详细的叶片形态评估gydF4y2Ba2gydF4y2Ba) (Licor 3100C叶面积计)。除了这些数量性状外,我们还为叶片和整株外观生成了定性的高地-低地指数,这些指数是在2019年夏末在德克萨斯州奥斯汀收集的gydF4y2Ba2gydF4y2Ba网站)。每种植物的特征以1-5的等级进行评估,从最低地到最高地。利用已建立的品种Alamo和Dacotah分别进行低地和高地性状的基线测定。植物性状评估包括:分蘖外观,从最厚和最像低地到最薄和最像高地;叶外观,从最宽,最长和最低地状到最短,最薄和最高地状;冠层颜色从最蓝和最典型的低地到深绿色和最典型的高地。这种视觉方法类似于柳枝稷育种者经常使用的基本选择标准。gydF4y2Ba
为了评估这些数据中的表型结构,我们使用了DAPCgydF4y2Ba101gydF4y2Ba。首先利用主成分分析(PCA)对表型数据进行转换,确定前10个主成分gydF4y2BakgydF4y2Ba-means算法将个体分为3个可能的组,目的是最大化组之间的差异。接下来,对10个保留的主成分实施DAPC,使用两个合成变量(即判别函数)提供对生态型集群的有效描述,这两个合成变量是具有最大组间方差和最小组内方差的原始表型变量的线性组合。gydF4y2Ba
我们对调查的651个四倍体基因型中的16个性状在MI和TX进行了分类gydF4y2Ba2gydF4y2Ba通过1层5个单元的低容量神经网络对3个生态型中的1个生态型(34个总特征,32个定量特征和2个定性有序特征)进行分析(补充数据)gydF4y2Ba5gydF4y2Ba).该神经网络采用插入式实现gydF4y2Ba106gydF4y2Ba并对7个已知生态型的栽培品种进行了培训(低地:Kanlow和Alamo;沿海:涨潮和斯图尔特;upland: Summer, Dacotah和Sunburst)和78个额外的基因型在同一个基于snp的遗传簇中(扩展数据图)。gydF4y2Ba3.gydF4y2Ba),与样本品种在表型PCA空间中聚类最紧密。这些高亲和力的范例基因型在补充数据中打印gydF4y2Ba5gydF4y2Ba。对其余582个表型为生态型分类性状的基因型用插入符号进行了生态型预测gydF4y2Ba106gydF4y2Ba。通过使用在代表北部和南部柳枝稷范围的花园收集的性状,我们希望避免当地气候对植物表型和随后的生态型分类的偏见。此外,与DAPC和专家资格相比,神经网络分类方法有一个显著的优势:因为神经网络锚定在已知和已发表的基因型上,包括这些常见品种的实验将能够更有效地概括我们的任务。gydF4y2Ba
混合和渗滤块的计算和年代测定gydF4y2Ba
我们通过两步管道建立了一个外加剂信息snp数据库。首先,祖先系数在“种群基因组学”中从具有相关祖先状态呼叫的四倍退化位点计算。每个亚种群中缺失数据最少且全基因组混合比例≤0.001的30个样本用于确定亚种群特异性等位基因频率。这些文库用于寻找至少有一个成对的snpgydF4y2BaFgydF4y2Ba圣gydF4y2Ba值>0.4,由snpRelate函数snpGdsFst中的' W&C84 '方法计算得出。其次,在每个亚群中对这些全局遗传信息位点进行分析,找出等位基因频率较小的位点(0.05)和缺失位点(0.05)。这些位点首先在亚群体内被进一步修剪到|位点gydF4y2BargydF4y2Ba| < 0.9(10个snp或1000 -bp窗口),则为|gydF4y2BargydF4y2BasnpRelate的| < 0.95 (1000 snp或10000 bp窗口)。这一过程导致每个亚群的SNP和文库计数如下:大西洋,579,468个SNP和284个文库;GULF, 641,975个snp和215个文库;中西部地区,481563个snp和196个图书馆。gydF4y2Ba
为了测试每对亚种群之间混合块的物理位置,我们使用了Ancestry_HMMgydF4y2Ba36gydF4y2Ba,gydF4y2Ba107gydF4y2Ba。这种方法利用假定亲本群体中的等位基因频率来确定测试群体中可能出现基因渗入的区域。对于三个亚种群中的每一个,我们试图确定混合块渗入的时间、程度和当前位置。在每种情况下,我们允许其他两个亚群中的每一个产生两次脉冲。Ancestry_HMM可以优化祖先脉冲发生前的代数和混合脉冲中涉及的个体比例。然而,使用>48万个站点和>150个库进行8参数优化在计算上是不可行的。因此,我们使用40个随机抽样的文库进行参数优化,这些文库的混合系数在混合比例分布的0.2-0.8分位数范围内,并且snp仅在N亚基因组的4号染色体上。我们选择这条染色体作为其他染色体的代表是因为缺乏明显的大频率渗入。所得到的祖先脉冲参数优化是建立在1万代之前的初始未混合种群上的,然后为其他两个亚种群中的每一个建立两个后续的混合脉冲;优化后的脉冲如下(来源-参考):gydF4y2BangydF4y2Ba一代又一代gydF4y2Ba= 8658和gydF4y2BaPgydF4y2Ba混在gydF4y2Ba= 0.001%;67和0.7%),海湾-大西洋(85和1.1%);17和0.25%),大西洋海湾(79和1.9%;11和0.38%),中西部-海湾地区(79和0.86%;大西洋-中西部地区分别为66和0.27%;14和0.036%),墨西哥湾-中西部(71和0.15%;14和0.033%)。这些脉冲提供给包含所有个体和染色体的完整模型,误差概率为0.001,出现之前的最大代数为10,000,有效种群大小为100,000。后验祖先概率被解码成单倍型块,块被分类成相似位置块的簇。gydF4y2Ba
环境基因组学gydF4y2Ba
从“自然地球”网站(gydF4y2Bahttps://www.naturalearthdata.com/gydF4y2Ba).各种绘图例程依赖于sfgydF4y2Ba108gydF4y2Ba和光栅gydF4y2Ba109gydF4y2Ba包在R环境中用于统计计算gydF4y2Ba110gydF4y2Ba。气候数据下载自WorldClimgydF4y2Ba22gydF4y2Ba(19个生物气候变量,0.5-arcmin分辨率1960-2000)gydF4y2Ba21gydF4y2Ba。通过动态聚类探讨了气候变量在不同收集点之间的分布gydF4y2Ba111gydF4y2Ba然后是围绕介质聚类进行分区gydF4y2Ba112gydF4y2Ba与gydF4y2BakgydF4y2Ba= 7。最具代表性的气候变量被定义为与每个簇内的第一个变异特征向量最相关的气候变量。七个集群中有六个包含WorldClim变量。gydF4y2Ba
从NOAA门户网站下载了距离每个花园站点最近的气象站的天气数据,这些站点拥有2018年9月1日至2019年10月31日的完整日温度(最低-最高)和降水数据。每个花园使用的NOAA气象站标识符如下:IL (USC00110338)、MI (USW00014815)、MO (USW00003945)、NE (USC00255362)、OK (USW00053926)、SD (USC00391076)、TXgydF4y2Ba1gydF4y2Ba(USC00414810), TXgydF4y2Ba2gydF4y2Ba(USC00410433), TXgydF4y2Ba3.gydF4y2Ba(USC00418862)和TXgydF4y2Ba4gydF4y2Ba(USW00003901)。gydF4y2Ba
在原始数据和估算数据的基础上,对不同花园的气候表型关联进行了研究。纬度与生存的关系(图2)gydF4y2Ba2 bgydF4y2Ba)对原始数据进行了逻辑回归,通过在R中使用二项家族的glm进行了逻辑回归,在R中使用所有可用表型的近邻完成了输入(gydF4y2BakgydF4y2Ba= 5),专门用于园林等级顺序的检验(图2)。gydF4y2Ba2 c, dgydF4y2Ba).在混合线性模型中,通过lmer实现了气候相似性与生物量的关联gydF4y2Ba92gydF4y2Ba,使用似然比检验将完整模型(固定=气候距离+截距,随机=基因型标识符)与没有气候距离固定效应的简化模型进行比较。gydF4y2Ba
采用物种分布模型(SDM)模拟了青藏高原所有生态型(高地、低地和沿海)的现代潜在分布范围gydF4y2Bap . virgatumgydF4y2Ba。用于建立SDMs的最终数据集包括277个(高地)、199个(沿海)和121个(低地)的发生记录。在我们最终的SDM模型中使用了6个环境预测因子(BIO1 =年平均温度,BIO2 =平均日差,BIO4 =温度季节性,BIO5 =最温暖月份的最高温度,BIO16 =最潮湿季度的降水量,BIO17 =最干燥季度的降水量)。然后用BIOMOD2 v.3.3生成sdmgydF4y2Ba113gydF4y2Ba采用7种建模算法:广义线性模型、增强回归树、人工神经网络、灵活判别分析、随机森林、分类树分析和多元自适应回归样条。对于每个模型,发生率数据与在建模研究区域内随机生成的500个伪缺席数据相耦合,存在和伪缺席的权重相等gydF4y2Ba114gydF4y2Ba。模型用80%的耦合出现和伪缺席数据进行训练,并用剩下的20%进行测试。每种建模算法对总共700个模型运行100次,并通过真技能统计(TSS)对这些模型进行评估。gydF4y2Ba115gydF4y2Ba。TSS值从0.2到0.5被认为是差的,0.6到0.8被认为是有用的,>0.8被认为是好的到优秀的gydF4y2Ba116gydF4y2Ba。基于TSS阈值(高地TSS阈值= 0.96,低地TSS阈值= 0.93,沿海TSS阈值= 0.965),从700个模型中约50个最佳SDMs计算出独特的整体SDMs。最终的总体sdm被投影到当前的气候层上,以显示现代的潜在范围(补充资料)gydF4y2Ba12gydF4y2Ba).gydF4y2Ba
我们通过在素食主义者中实施冗余分析,研究了大西洋亚群中中西部基因渗入的存在如何与气候、地理和亲属关系的独立和共同影响相关联gydF4y2Ba117gydF4y2Ba,gydF4y2Ba118gydF4y2Ba,gydF4y2Ba119gydF4y2Ba,gydF4y2Ba120gydF4y2Ba。为了划分由气候、亲属关系和地理因素引起的渗透存在的可解释方差,我们运行了四个模型:一个完整的模型,具有渗透存在(潜在的渗透块被编码为0,大西洋遗传为1,中西部遗传为1),由气候(即七个代表性气候变量)、亲属关系(从一组假定中性标记计算出的前两个主要成分)和地理(纬度和经度)解释,这三个因素中的每一个都以其他两个因素为条件。比较每个模型约束矩阵的惯性(即方差)值,以确定气候、亲属关系、地理及其联合效应的相对重要性。此外,为了找到与气候和生存校正生物量密切相关的渗入区域,我们从两个额外的模型中提取了冗余分析轴的负荷:(1)一个仅由气候预测,(2)一个仅由生存校正生物量预测。根据排列检验,两个模型均显著(gydF4y2BangydF4y2Ba= 999;gydF4y2BaPgydF4y2Ba均< 0.001),所有轴近似为正态分布。每个轴尾部的snp负载更有可能表明与预测因子(即气候或生存校正生物量)相关的选择,因此我们确定了至少2.5 s.d(双尾)的所有标记gydF4y2BaPgydF4y2Ba= 0.012)作为自然选择下的渗入gydF4y2Ba119gydF4y2Ba。gydF4y2Ba
GWASgydF4y2Ba
由于我们常用的花园数据集的规模很大,我们开发了一个管道- switchgrassGWAS R包(gydF4y2Bahttps://github.com/Alice-MacQueen/switchgrassGWASgydF4y2Ba)——允许在多样性面板上进行快速、低内存密集型的GWAS,并分析在不同位点测量的表型上SNP效应相似或不同的程度。这个包利用了bigsnprgydF4y2Ba121gydF4y2Ba对编码为矩阵的大量SNP阵列进行快速(比TASSEL快300倍)统计分析。它还结合了当前人类遗传学文献中SNP质量控制、修剪和插入以及GWAS群体结构校正的金标准。为了测试在许多条件下(例如,多个地点、气候变量等)许多影响的重要性,我们使用了mashrgydF4y2Ba32gydF4y2Ba这是一种灵活的数据驱动方法,可以共享任何数据集中效应大小和符号模式的信息,这些数据集可以在许多条件和snp的逐个条件基础上估计其影响。我们使用局部错误标记率确定哪些snp具有显著表型效应的证据,这类似于错误发现率,但更保守(因为它们也反映了效应标记估计的不确定性)。gydF4y2Ba122gydF4y2Ba。我们用这些值用log找到snpgydF4y2Ba10gydF4y2Ba-变换后的贝叶斯因子>2。这里,贝叶斯因子是一个SNP出现一个或多个显著表型效应的可能性与该SNP只产生零效应的可能性之比。根据之前的工作gydF4y2Ba33gydF4y2Ba,贝叶斯系数为bb10gydF4y2Ba2gydF4y2Ba被认为是支持SNP具有一个或多个显着表型效应的假设的决定性证据。gydF4y2Ba
为了计算气候和健康相关snp的区域遗传力,我们采用了先前描述的两步法gydF4y2Ba123gydF4y2Ba。方差成分分析使用ASReml (VSN International)软件完成,使用van Raden方法计算的基因组关系矩阵gydF4y2Ba124gydF4y2Ba。计算每个亚群内和整个多样性面板的基因组关系矩阵。基于单变量GWAS中使用的所有snp计算亲属关系矩阵(gydF4y2BaGgydF4y2Ba),以及基于该亚群中与气候显著相关的snp的亲属矩阵(loggydF4y2Ba10gydF4y2Ba-变换后的贝叶斯因子>2;gydF4y2Ba问gydF4y2Ba气候gydF4y2Ba)和基于与该亚种群生物量或冬季存活率显著相关的snp的亲缘关系矩阵(loggydF4y2Ba10gydF4y2Ba-转换贝叶斯因子> 2,或>1.385海湾亚群;gydF4y2Ba问gydF4y2Ba健身gydF4y2Ba).这些亲缘关系矩阵用于区域遗传力作图gydF4y2Ba123gydF4y2Ba就像之前的出版物一样gydF4y2Ba125gydF4y2Ba,使用混合模型的形式:gydF4y2Ba
其中向量gydF4y2BaygydF4y2Ba表示生物量值,gydF4y2BaZgydF4y2Ba是随机效应的设计矩阵,gydF4y2BaugydF4y2Ba是整个基因组的加性遗传效应,gydF4y2BavgydF4y2Ba区域基因组的加性遗传效应和gydF4y2BaegydF4y2Ba是残差。矩阵gydF4y2BaGgydF4y2Ba为全基因组加性效应中使用所有snp的全基因组关系矩阵。矩阵gydF4y2Ba问gydF4y2Ba区域基因组关系是否如上所述得到:之一gydF4y2Ba问gydF4y2Ba气候gydF4y2Ba或gydF4y2Ba问gydF4y2Ba健身gydF4y2Ba。gydF4y2Ba我gydF4y2Ba等级是——gydF4y2BaygydF4y2Ba单位矩阵,其中gydF4y2BaygydF4y2Ba等于生物量值的个数。全基因组、区域基因组和残差方差gydF4y2Ba\({\σ}_{你}^ {2}\)gydF4y2Ba,gydF4y2Ba\({\σ}_ {v} ^ {2} \)gydF4y2Ba和gydF4y2Ba\({\σ}_ {e} ^ {2} \)gydF4y2Ba,分别。表型变异(gydF4y2Ba\({\σ}_ {{rm \ p {}}} ^ {2} \)gydF4y2Ba)是gydF4y2Ba\({\σ}_{你}^ {2}\)gydF4y2Ba+gydF4y2Ba\({\σ}_ {v} ^ {2} \)gydF4y2Ba+gydF4y2Ba\({\σ}_ {e} ^ {2} \)gydF4y2Ba。全基因组遗传力、区域遗传力和总遗传力gydF4y2Ba\ ({h} _{你}^ {2}\)gydF4y2Ba= (gydF4y2Ba\({\σ}_{你}^ {2}\)gydF4y2Ba/gydF4y2Ba\({\σ}_ {{rm \ p {}}} ^ {2} \)gydF4y2Ba),gydF4y2Ba\ ({h} _ {v} ^ {2} \)gydF4y2Ba= (gydF4y2Ba\({\σ}_ {v} ^ {2} \)gydF4y2Ba/gydF4y2Ba\({\σ}_ {{rm \ p {}}} ^ {2} \)gydF4y2Ba),gydF4y2Ba\ ({h} _ {u + v} ^ {2} \)gydF4y2Ba= (gydF4y2Ba\({\σ}_{你}^ {2}\)gydF4y2Ba+gydF4y2Ba\({\σ}_ {v} ^ {2} \)gydF4y2Ba/gydF4y2Ba\({\σ}_ {{rm \ p {}}} ^ {2} \)gydF4y2Ba),分别。gydF4y2Ba
这些模型是在进行亚种群GWAS的三个地点运行的:哥伦比亚,密苏里州;密歇根州的山胡桃角;以及德克萨斯州的奥斯汀。这产生了80个模型:4组种群(全多样性面板和3个亚种群),2种模型类型(一个模型与gydF4y2BaGgydF4y2Ba只有和一个gydF4y2BaGgydF4y2Ba+gydF4y2Ba问gydF4y2Ba模型),10种表型(3个地点的生物量和7个环境变量)。gydF4y2Ba
方差成分分析也用于划分K亚基因组和n亚基因组之间的方差。只有具有祖先状态的snp调用(gydF4y2Ba补充数据9gydF4y2Ba)在该分析中使用,结果每个种群子集使用了460,429个snp。根据特定染色体上的所有snp计算亲属矩阵(gydF4y2Ba问gydF4y2BaChr01KgydF4y2Ba来gydF4y2Ba问gydF4y2BaChr09KgydF4y2Ba,gydF4y2Ba问gydF4y2BaChr01NgydF4y2Ba来gydF4y2Ba问gydF4y2BaChr09NgydF4y2Ba),得到18个亲属矩阵。这些亲属关系矩阵被用于区域遗传力映射,使用混合模型的形式:gydF4y2Ba
其中向量gydF4y2BaygydF4y2Ba表示生物量值,gydF4y2BaZgydF4y2Ba是随机效应的设计矩阵,gydF4y2BavgydF4y2Ba1 kgydF4y2Ba(gydF4y2BavgydF4y2Ba9 kgydF4y2Ba)或gydF4y2BavgydF4y2Ba1 ngydF4y2Ba(gydF4y2BavgydF4y2Ba9 ngydF4y2Ba)(统称为gydF4y2BavgydF4y2Ba我gydF4y2Ba)是染色体特异性基因组加性遗传效应和gydF4y2BaegydF4y2Ba是残差。矩阵gydF4y2Ba问gydF4y2Ba我gydF4y2Ba是N和K亚基因组的9条染色体的染色体特异性基因组关系矩阵。染色体特异性和残差是gydF4y2Ba\({\σ}_ {{v} _{我}}^ {2}\)gydF4y2Ba和gydF4y2Ba\({\σ}_ {e} ^ {2} \)gydF4y2Ba,分别。染色体特异性遗传力是gydF4y2Ba\ ({h} _ {{v} _{我}}^ {2}\)gydF4y2Ba= (gydF4y2Ba\({\σ}_ {{v} _{我}}^ {2}\)gydF4y2Ba/gydF4y2Ba\({\σ}_ {{rm \ p {}}} ^ {2} \)gydF4y2Ba),而亚基因组特异性遗传力是每个亚基因组内9条染色体上这些差异的总和。gydF4y2Ba
候选基因探索gydF4y2Ba
我们整合了多个数据结构,对渗入间隔内的候选基因进行排序,并提供有意义的筛选标准,以及与数量性状位点峰值的物理接近度。在GWAS峰的情况下,候选基因被定义为围绕mashr峰的20 kb间隔内的位点。基因组渗入的候选基因必须至少部分重叠于渗入间隔。由于GWAS和基因渗入的推断是在遗传亚群内进行的,所有统计数据均报道于gydF4y2Ba补充数据7gydF4y2Ba(候选基因列表)也具有亚种群特异性,但基因共表达分析除外(该分析仅对用于注释目的的AP13 rna测序文库进行)(gydF4y2Ba补充数据3gydF4y2Ba)).对于给定的区间,我们给出一组统计量。首先,将峰值位置的物理接近度计算为基因到间隔(渗入)或GWAS峰值位置的中点。其次,由于一个亚群体中GWAS峰值的因果位点在该亚群体中必然是可变的,我们提取了候选基因模型内和邻近的所有snp。这些变体用SNPeff进行了注释gydF4y2Ba126gydF4y2Ba以及三大类变量的加权和(高、中、低;这些的描述可以在gydF4y2Bahttps://pcingola.github.io/SnpEff/se_inputoutput/#effect-prediction-detailsgydF4y2Ba)计算为SNPeff_score =高× 20 +中× 5 +低× 1。第三,对于每个基因,我们计算了结构变异和存在缺失变异的次要等位基因频率。第四,我们包括每个基因的WGCNA集群的身份载体。最后,如果候选基因是先前发表的开花时GWAS候选基因的同源物gydF4y2Ba127gydF4y2Ba,包括重叠区间或基因的身份。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到本文。gydF4y2Ba
数据可用性gydF4y2Ba
序列读取档案的所有RNA和DNA测序文库的加入代码可以在gydF4y2Ba补充数据3gydF4y2Ba和gydF4y2Ba4gydF4y2Ba,分别。v5 AP13基因组已存入DDBJ/ENA/GenBank,代码为JABWAI000000000。基因组,基因和重复注释也可以直接从Phytozome下载gydF4y2Bahttps://phytozome-next.jgi.doe.gov/info/Pvirgatum_v5_1gydF4y2Ba。只要有可能,植物材料将根据要求共享。gydF4y2Ba源数据gydF4y2Ba都提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
用于GWAS和其他分析的自定义管道可从dataverse网站获得gydF4y2Bahttps://doi.org/10.18738/T8/J377KEgydF4y2Ba。gydF4y2Ba
参考文献gydF4y2Ba
罗贝尔,D. B, Schlenker, W. & Costa-Roberts, J. 1980年以来气候趋势与全球作物产量。gydF4y2Ba科学gydF4y2Ba333gydF4y2Ba, 616-620(2011)。gydF4y2Ba
查利诺,A. J.等。气候变化与适应下作物产量的荟萃分析。gydF4y2BaNat,爬。常gydF4y2Ba。gydF4y2Ba4gydF4y2Ba, 287-291(2014)。gydF4y2Ba
罗森茨威格,C.等。基于全球网格化作物模式比对的21世纪气候变化农业风险评估gydF4y2Ba自然科学进展。美国gydF4y2Ba111gydF4y2Ba中文信息学报,368 - 3273(2014)。gydF4y2Ba
波特,j.r.等人gydF4y2Ba气候变化2014:影响、适应和脆弱性。A部分:全球和部门方面(第二工作组对政府间气候变化专门委员会第五次评估报告的贡献)gydF4y2Ba(eds Field, c.b. et al.) 485-533(剑桥大学出版社,2014)。gydF4y2Ba
贝文,m.w.等。作物改良的基因组创新。gydF4y2Ba自然gydF4y2Ba543gydF4y2Ba, 346-354(2017)。gydF4y2Ba
Nelson, R., Wiesner-Hanks, T., Wisser, R.和Balint-Kurti, P.。gydF4y2BaNat, Genet牧师gydF4y2Ba。gydF4y2Ba19gydF4y2Ba, 21-33(2018)。gydF4y2Ba
里瑟,p.g.,伯尼,e.c.和布洛克,h.d.。gydF4y2Ba真正的草原生态系统gydF4y2Ba(Dowden, Hutchinson and Ross, 1981)。gydF4y2Ba
Suyker, a . E.和Verma, S. B.原生高草草原生态系统二氧化碳净交换的全年观测。gydF4y2Ba水珠。改变生物gydF4y2Ba。gydF4y2Ba7gydF4y2Ba, 279-289(2001)。gydF4y2Ba
Schmer, m.r., Vogel, k.p., Mitchell, r.b. & Perrin, r.k.。柳枝稷纤维素乙醇的净能量。gydF4y2Ba自然科学进展。美国gydF4y2Ba105gydF4y2Ba, 464-469(2008)。gydF4y2Ba
Palik, D. J, Snow, A. A., Stottlemyer, A. L, Miriti, M. N. & Heaton, E. A.。柳枝稷非本地品种与本地野生种群的相对表现(gydF4y2Ba黍virgatumgydF4y2Ba)在竞争实验中。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba11gydF4y2Ba, e0154444(2016)。gydF4y2Ba
麦克劳克林,S.等人gydF4y2Ba展望新作物和新用途gydF4y2Ba(编。Janick, J.) 282-299 (ASHS, 1999)。gydF4y2Ba
Vogel, k.p., Schmer, M. R. & Mitchell, R. B.植物适应区:植物材料的生态和气候分类。gydF4y2Ba牧场生态。等内容gydF4y2Ba。gydF4y2Ba58gydF4y2Ba, 315-319(2005)。gydF4y2Ba
Casler等人。柳枝稷种群的纵向和纵向适应性。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba47gydF4y2Ba, 2249-2260(2007)。gydF4y2Ba
Lipka, A. E.等。加速柳枝稷(gydF4y2Ba黍virgatumgydF4y2BaL.利用基因组选择方法的育种周期。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, e112227(2014)。gydF4y2Ba
Poudel, h.p., sanangco, m.d., Kaeppler, s.m., Buell, c.r. & Casler, m.d.美国北部低地柳枝稷冬季生存的基因组预测。gydF4y2BaG3gydF4y2Ba9gydF4y2Ba, 1921-1931(2019)。gydF4y2Ba
劳里,d.b.等人。QTL与环境的相互作用是柳枝稷在大纬度梯度上自适应分化的基础。gydF4y2Ba自然科学进展。美国gydF4y2Ba116gydF4y2Ba中文信息学报,12933-12941(2019)。gydF4y2Ba
Triplett, J. K, Wang, Y., Zhong, J. & Kellogg, E. A.柳枝稷多倍体历史的5个核位点解析(英文)gydF4y2Ba黍virgatumgydF4y2BaL.)和亲属。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba7gydF4y2Ba, e38702(2012)。gydF4y2Ba
Martínez-Reyna, J. M. & Vogel, K. P.柳枝稷的不亲和系统。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba42gydF4y2Ba, 1800-1805(2002)。gydF4y2Ba
Casler, M. D, Vogel, K. P. & Harrison, M.柳枝稷种质资源。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba55gydF4y2Ba中文信息学报,2463-2478(2015)。gydF4y2Ba
埃文斯等人。广泛的遗传多样性存在于北美柳枝稷种质资源中。gydF4y2Ba植物基因组gydF4y2Ba11gydF4y2Ba, 1-16(2018)。gydF4y2Ba
Wang, T., Hamann, A., Spittlehouse, D. & Carroll, C.北美地区历史和未来时期的局部缩小尺度和空间可定制的气候数据。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba11gydF4y2Ba, e0156720(2016)。gydF4y2Ba
Fick, S. E.和Hijmans, R. J. WorldClim 2:全球陆地区域新的1公里空间分辨率气候面。gydF4y2BaInt。j . ClimatolgydF4y2Ba。gydF4y2Ba37gydF4y2Ba, 4302-4315(2017)。gydF4y2Ba
劳里,d.b.等人。生态类型之间的适应和沿环境梯度gydF4y2Ba黍virgatumgydF4y2Ba。gydF4y2Ba点。NatgydF4y2Ba。gydF4y2Ba183gydF4y2Ba, 682-692(2014)。gydF4y2Ba
Casler, M. D, Vogel, K. P., Taliaferro, C. M. & Wynia, R. L.柳枝稷种群的纬度适应。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba44gydF4y2Ba中文信息学报,293-303(2004)。gydF4y2Ba
波特,C. L.高地和低地柳枝稷的变异分析gydF4y2Ba黍virgatumgydF4y2BaL在俄克拉荷马州中部。gydF4y2Ba生态gydF4y2Ba47gydF4y2Ba1966年,980-992。gydF4y2Ba
麦克millan, C.北美四种草原牧草的生态类型分化。1 .移植群落内形态变异。gydF4y2Ba点。j .机器人gydF4y2Ba。gydF4y2Ba51gydF4y2Ba, 1119-1128(1964)。gydF4y2Ba
Grabowski, p.p., Morris, g.p., Casler, m.d.和Borevitz, j.o.群体基因组变异揭示了柳枝稷历史、适应性和倍性的作用。gydF4y2Ba摩尔。生态gydF4y2Ba。gydF4y2Ba23gydF4y2Ba中文信息学报,4059-4073(2014)。gydF4y2Ba
卢,F.等。柳枝稷基因组多样性、倍性和进化:基于网络的SNP发现协议的新见解。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba。gydF4y2Ba9gydF4y2Ba, e1003215(2013)。gydF4y2Ba
Casler, m.d.等人,《柳枝稷和大蓝茎提高生物量产量的30年进展》。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba58gydF4y2Ba, 1242-1254(2018)。gydF4y2Ba
卡斯勒,m.d.和沃格尔,k.p.选择生物量产量在高地,低地,和杂交柳枝稷。gydF4y2Ba作物科学gydF4y2Ba。gydF4y2Ba54gydF4y2Ba, 626-636(2014)。gydF4y2Ba
Suarez-Gonzalez, A, Lexer, C.和Cronk, Q. C. B.适应性渗入:植物的视角。gydF4y2Ba医学杂志。列托语gydF4y2Ba。gydF4y2Ba14gydF4y2Ba, 20170688(2018)。gydF4y2Ba
王国强,王志强,王志强,王志强,王志强,王志强,王志强,王志强,王志强,王志强,王志强,王志强,王志强。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba51gydF4y2Ba, 187-195(2019)。gydF4y2Ba
卡斯,r.e. &拉特里,a.e.贝叶斯因子。gydF4y2Baj。统计协会。gydF4y2Ba。gydF4y2Ba90gydF4y2Ba, 773-795(1995)。gydF4y2Ba
Fournier-Level, A.等。当地适应的地图gydF4y2Ba拟南芥gydF4y2Ba。gydF4y2Ba科学gydF4y2Ba334gydF4y2Ba, 86-89(2011)。gydF4y2Ba
张勇,等。冰期后的演化gydF4y2Ba黍virgatumgydF4y2Ba: SSR标记和cpDNA序列揭示的多样性中心和基因库。gydF4y2Ba遗传gydF4y2Ba139gydF4y2Ba, 933-948(2011)。gydF4y2Ba
Corbett-Detig, R. & Nielsen, R.使用任意倍性样本的下一代序列数据同时估计本地祖先和混合时间的隐马尔可夫模型方法。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba。gydF4y2Ba13gydF4y2Ba, e1006529(2017)。gydF4y2Ba
Todesco, M.等。大量单倍型是向日葵生态型分化的基础。gydF4y2Ba自然gydF4y2Ba584gydF4y2Ba, 602-607(2020)。gydF4y2Ba
Lucek, K., Lemoine, M. & Seehausen, O.在最近的范围扩张期间,当代生态型分化是由适应性渐渗促进的。gydF4y2Baj .另一个星球。医学杂志gydF4y2Ba。gydF4y2Ba27gydF4y2Ba, 2233-2248(2014)。gydF4y2Ba
惠特尼,k.d.等。数量性状位点定位鉴定了参与野生向日葵适应性渗入和范围扩展的候选等位基因。gydF4y2Ba摩尔。生态gydF4y2Ba。gydF4y2Ba24gydF4y2Ba, 2194-2211(2015)。gydF4y2Ba
多倍体的优点和缺点。gydF4y2BaNat, Genet牧师gydF4y2Ba。gydF4y2Ba6gydF4y2Ba; 836-846(2005)。gydF4y2Ba
Mattenberger, F., Sabater-Muñoz, B., Toft, C. & Fares, M. A.gydF4y2Ba酿酒酵母gydF4y2Ba以及适应的起源。gydF4y2BaG3gydF4y2Ba7gydF4y2Ba, 63-75(2017)。gydF4y2Ba
克拉克,J. W.和多诺霍,P. C. J.全基因组复制与植物宏观进化。gydF4y2Ba植物科学动态gydF4y2Ba。gydF4y2Ba23gydF4y2Ba, 933-945(2018)。gydF4y2Ba
多倍体、杂交和新生境的入侵。gydF4y2Ba安。密苏里州机器人。加尔省gydF4y2Ba。gydF4y2Ba72gydF4y2Ba, 824(1985)。gydF4y2Ba
Bird, K. A, VanBuren, R., Puzey, J. R.和Edger, P. P.杂交和新近多倍体亚基因组显性的原因和后果。gydF4y2Ba新植醇gydF4y2Ba。gydF4y2Ba220gydF4y2Ba, 87-93(2018)。gydF4y2Ba
温德尔,李文杰。异源四倍体棉花物种形成的进化速率变异、基因组优势和重复基因表达进化。gydF4y2Ba新植醇gydF4y2Ba。gydF4y2Ba186gydF4y2Ba, 184-193(2010)。gydF4y2Ba
埃德格,p.p.等人。草莓八倍体基因组的起源和进化。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba51gydF4y2Ba, 541-547(2019)。gydF4y2Ba
陈振杰等。5个基因组的多样性gydF4y2BaGossypiumgydF4y2Ba异源多倍体品种及其对棉花改良的影响。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba52gydF4y2Ba中文信息学报,525-533(2020)。gydF4y2Ba
Session, a.m.等。异源四倍体青蛙的基因组进化gydF4y2Ba非洲爪蟾蜍光滑的gydF4y2Ba。gydF4y2Ba自然gydF4y2Ba538gydF4y2Ba, 336-343(2016)。gydF4y2Ba
Nieto Feliner, G., Casacuberta, J. & Wendel, J. F.杂交和多倍体进化新颖性的基因组学。gydF4y2Ba前面。麝猫gydF4y2Ba。gydF4y2Ba11gydF4y2Ba, 792(2020)。gydF4y2Ba
Davis, M. B.和Shaw, R. G.第四纪气候变化的范围转移和适应性响应。gydF4y2Ba科学gydF4y2Ba292gydF4y2Ba, 673-679(2001)。gydF4y2Ba
rnaturalearthdata:“rnaturalearth”中使用的来自自然地球的世界矢量地图数据。R包版本0.1.0。gydF4y2Bahttps://CRAN.R-project.org/package=rnaturalearthdatagydF4y2Ba(2017)。gydF4y2Ba
肖,C.-L。et al。MECAT:用于单分子测序读取的快速制图,纠错和从头组装。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 1072-1074(2017)。gydF4y2Ba
下巴,c。et al。非杂交,完成微生物基因组组装从长读取SMRT测序数据。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 563-569(2013)。gydF4y2Ba
Kent, W. J. blat - BLAST-like alignment tool。gydF4y2Ba基因组ResgydF4y2Ba。gydF4y2Ba12gydF4y2Ba, 656-664(2002)。gydF4y2Ba
Li, H. & Durbin, R.基于Burrows-Wheeler变换的快速准确短读对齐。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 1754-1760(2009)。gydF4y2Ba
麦肯纳等人。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。gydF4y2Ba基因组ResgydF4y2Ba。gydF4y2Ba20.gydF4y2Ba, 1297-1303(2010)。gydF4y2Ba
Sharma, M. K.等。柳枝稷BAC文库筛选和序列分析确定了预测生物量和胁迫应答相关基因。gydF4y2BaBioenerg。ResgydF4y2Ba。gydF4y2Ba9gydF4y2Ba, 109-122(2016)。gydF4y2Ba
的快速,无锁的方法,用于有效的并行计数的发生gydF4y2BakgydF4y2Ba即。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 764-770(2011)。gydF4y2Ba
史密特,a.f.,哈布利,r.g。gydF4y2BaRepeatMaskergydF4y2Ba,gydF4y2Bahttp://www.repeatmasker.org/gydF4y2Ba(1996)。gydF4y2Ba
elinghaus, D., Kurtz, S.和Willhoeft, U. LTRharvest,一种用于LTR反转录转座子重新检测的高效且灵活的软件。gydF4y2BaBMC生物信息学gydF4y2Ba9gydF4y2Ba, 18(2008)。gydF4y2Ba
Katoh, K. & Standley, D. M. MAFFT多序列比对软件版本7:性能和可用性的改进。gydF4y2Ba摩尔。杂志。另一个星球gydF4y2Ba。gydF4y2Ba30.gydF4y2Ba, 772-780(2013)。gydF4y2Ba
类人猿5.0:现代系统发育和进化分析的环境。gydF4y2Ba生物信息学gydF4y2Ba35gydF4y2Ba, 526-528(2019)。gydF4y2Ba
Bennetzen, j.l.等。模式植物参考基因组序列gydF4y2BaSetariagydF4y2Ba。gydF4y2BaNat。gydF4y2Ba。gydF4y2Ba30.gydF4y2Ba, 555-561(2012)。gydF4y2Ba
洛弗尔等人。植物对自然干旱胁迫的分子反应的基因组景观gydF4y2Ba黍halliigydF4y2Ba。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba9gydF4y2Ba, 5213(2018)。gydF4y2Ba
Wu, T. D.和Nacu, S.短读段中复杂变异和剪接的快速和耐snp检测。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 873-881(2010)。gydF4y2Ba
左,C.等。PacBio长读测序揭示柳枝稷转录组的复杂性。gydF4y2BaBiotechnol。生物燃料gydF4y2Ba11gydF4y2Ba, 170(2018)。gydF4y2Ba
哈斯,b.j.等人。改善gydF4y2Ba拟南芥gydF4y2Ba使用最大转录物比对组装的基因组注释。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba31gydF4y2Ba, 5654-5666(2003)。gydF4y2Ba
Slater, g.s.c.和Birney, E.生物序列比较的自动启发式生成。gydF4y2BaBMC生物信息学gydF4y2Ba6gydF4y2Ba, 31(2005)。gydF4y2Ba
Lamesch, P.等人。的gydF4y2Ba拟南芥gydF4y2Ba信息资源(TAIR):改进的基因注释和新工具。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba40gydF4y2Ba, d1202-d1210(2012)。gydF4y2Ba
Schmutz等人。古多倍体大豆基因组序列。gydF4y2Ba自然gydF4y2Ba463gydF4y2Ba, 178-183(2010)。gydF4y2Ba
Jain, R.等。模式水稻品种KitaakeX基因组序列分析。gydF4y2BaBMC基因组学gydF4y2Ba20.gydF4y2Ba, 905(2019)。gydF4y2Ba
Mamidi, S.等。绿谷子的基因组资源gydF4y2BaSetaria冬青gydF4y2Ba能够发现有农艺价值的基因座。gydF4y2BaNat。gydF4y2Ba。gydF4y2Ba38gydF4y2Ba[j] .自然科学学报,2020。gydF4y2Ba
帕特森,a.h.等。的gydF4y2Ba高粱二色的gydF4y2Ba基因组和草的多样化。gydF4y2Ba自然gydF4y2Ba457gydF4y2Ba; 551-556(2009)。gydF4y2Ba
戈登,s.p.等人。广泛的基因含量变异gydF4y2BaBrachypodium distachyongydF4y2Ba泛基因组与种群结构相关。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba8gydF4y2Ba, 2184(2017)。gydF4y2Ba
UniProt财团。UniProt:蛋白质知识的全球中心。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba47gydF4y2Ba, d506-d515(2019)。gydF4y2Ba
史密斯,A.和哈布利,R.。gydF4y2BaRepeatModeler开放- 1.0gydF4y2Ba,gydF4y2Bahttp://www.repeatmasker.org/gydF4y2Ba(2010)。gydF4y2Ba
鲍伟,Kojima, K. K. & Kohany, O. rebase更新:真核生物基因组重复元件数据库。gydF4y2Ba暴徒。DNAgydF4y2Ba6gydF4y2Ba, 11(2015)。gydF4y2Ba
Emms, D. M. & Kelly, S. OrthoFinder:解决全基因组比较中的基本偏差显著提高了正位群推断的准确性。gydF4y2Ba基因组医学杂志gydF4y2Ba。gydF4y2Ba16gydF4y2Ba, 157(2015)。gydF4y2Ba
古德斯坦博士等人。Phytozome:绿色植物基因组学比较平台。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba40gydF4y2Ba, d1178-d1186(2012)。gydF4y2Ba
Schliep, k.p. phangorn: R的系统发育分析。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 592-593(2011)。gydF4y2Ba
Subramanian, A. R, Kaufmann, M. & Morgenstern, B. DIALIGN-TX:基于片段的多序列比对的贪婪和渐进方法。gydF4y2Ba算法学报。生物学报gydF4y2Ba。gydF4y2Ba3.gydF4y2Ba, 6(2008)。gydF4y2Ba
Talavera, G.和Castresana, J.从蛋白质序列比对中去除歧义和模糊对齐块后系统发育的改进。gydF4y2Ba系统。医学杂志gydF4y2Ba。gydF4y2Ba56gydF4y2Ba, 564-577(2007)。gydF4y2Ba
Guindon, S.等。估计最大似然系统发育的新算法和方法:评估PhyML 3.0的性能。gydF4y2Ba系统。医学杂志gydF4y2Ba。gydF4y2Ba59gydF4y2Ba, 307-321(2010)。gydF4y2Ba
在没有分子钟的情况下,推断分子进化的绝对速率和分化时间。gydF4y2Ba生物信息学gydF4y2Ba19gydF4y2Ba, 301-302(2003)。gydF4y2Ba
Anders, S., Pyl, P. T. & Huber, W. htseq -一个处理高通量测序数据的Python框架。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 166-169(2015)。gydF4y2Ba
Love, M. I, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志gydF4y2Ba。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
Langfelder, P. & Horvath, S. WGCNA:加权相关网络分析的R包。gydF4y2BaBMC生物信息学gydF4y2Ba9gydF4y2Ba, 559(2008)。gydF4y2Ba
李,H.等。序列比对/映射格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 2078-2079(2009)。gydF4y2Ba
科博尔特等人。VarScan 2:通过外显子组测序发现癌症的体细胞突变和拷贝数改变。gydF4y2Ba基因组ResgydF4y2Ba。gydF4y2Ba22gydF4y2Ba, 568-576(2012)。gydF4y2Ba
奥康奈尔,J.等。生物库规模数据集的单倍型估计。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba48gydF4y2Ba, 817-820(2016)。gydF4y2Ba
Danecek, P.等。变体调用格式和VCFtools。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 2156-2158(2011)。gydF4y2Ba
Bates, D. Mächler, M., Bolker, B. & Walker, S.使用lme4拟合线性混合效应模型。gydF4y2BaJ. Stat. softwaregydF4y2Ba。gydF4y2Ba67gydF4y2Ba,gydF4y2Bahttps://doi.org/10.18637/jss.v067.i01gydF4y2Ba(2015)。gydF4y2Ba
Azad, A, Pavlopoulos, G. A, Ouzounis, C. A, Kyrpides, N. C. & Buluç, A. HipMCL:大规模网络中马尔可夫聚类算法的高性能并行实现。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba46gydF4y2Ba, 33(2018)。gydF4y2Ba
VanBuren, R.等。耐干燥草的单分子测序gydF4y2BaOropetium thomaeumgydF4y2Ba。gydF4y2Ba自然gydF4y2Ba527gydF4y2Ba, 508-511(2015)。gydF4y2Ba
麦考密克等。的gydF4y2Ba高粱二色的gydF4y2Ba参考基因组:改进组装、基因注释、转录组图谱和基因组组织的特征。gydF4y2Ba植物JgydF4y2Ba。gydF4y2Ba93gydF4y2Ba中文信息学报,338-354(2018)。gydF4y2Ba
Sedlazeck, F. J.等。利用单分子测序精确检测复杂结构变异。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba, 461-468(2018)。gydF4y2Ba
杰弗里斯等人。瞬时结构变异对裂变酵母的数量性状和生殖隔离有重要影响。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba8gydF4y2Ba, 14061(2017)。gydF4y2Ba
郑旭,等。一个用于SNP数据相关性和主成分分析的高性能计算工具集。gydF4y2Ba生物信息学gydF4y2Ba28gydF4y2Ba, 3326-3328(2012)。gydF4y2Ba
雷明顿等人。玉米基因组连锁不平衡结构与表型关联。gydF4y2Ba自然科学进展。美国gydF4y2Ba98gydF4y2Ba[j] .中文信息学报(英文版),2001。gydF4y2Ba
Purcell, S.等。PLINK:全基因组关联和基于群体的连锁分析的工具集。gydF4y2Ba点。j .的嗡嗡声。麝猫gydF4y2Ba。gydF4y2Ba81gydF4y2Ba; 559-575(2007)。gydF4y2Ba
Jombart, T., Devillard, S. & Balloux, F.主成分的判别分析:一种分析遗传结构群体的新方法。gydF4y2BaBMC麝猫gydF4y2Ba。gydF4y2Ba11gydF4y2Ba, 94(2010)。gydF4y2Ba
Jombart, T.和Ahmed, I. adegenet 1.3-1:全基因组SNP数据分析的新工具。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 3070-3071(2011)。gydF4y2Ba
Pritchard, J. K, Stephens, M. & Donnelly, P.使用多位点基因型数据推断种群结构。gydF4y2Ba遗传学gydF4y2Ba155gydF4y2Ba, 945-959(2000)。gydF4y2Ba
Schiffels, S. & Durbin, R.从多个基因组序列推断人类种群规模和分离历史。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba46gydF4y2Ba[j], 2014。gydF4y2Ba
李建军,李建军,李建军,等。基于效应量和检验统计量渐近零分布的多元距离矩阵回归。gydF4y2Ba心理测量学gydF4y2Ba82gydF4y2Ba, 1052-1077(2017)。gydF4y2Ba
库恩,M.等。注:分类和回归训练。R包版本6.0-78。gydF4y2Bahttps://CRAN.R-project.org/package=caretgydF4y2Ba(2016)。gydF4y2Ba
Medina, P., Thornlow, B., Nielsen, R. & Corbett-Detig, R.在局部祖先推断中估计多个混合脉冲的时间。gydF4y2Ba遗传学gydF4y2Ba210gydF4y2Ba, 1089-1107(2018)。gydF4y2Ba
R的简单特性:对空间矢量数据的标准化支持。gydF4y2BaR JgydF4y2Ba。gydF4y2Ba10gydF4y2Ba, 439(2018)。gydF4y2Ba
Hijmans, r.j.等。栅格:地理数据分析和建模。R包版本3.4-5。gydF4y2Bahttps://CRAN.R-project.org/package=rastergydF4y2Ba(2015)。gydF4y2Ba
R核心团队。统计计算语言与环境;gydF4y2Bahttps://www.r-project.org/gydF4y2Ba(R Foundation for Statistical Computing, 2013)。gydF4y2Ba
Langfelder, P., Zhang, B.和Horvath, S. dynamictreeccut:层次聚类树形图中聚类的检测方法。R包版本1.63-1。gydF4y2Bahttps://CRAN.R-project.org/package=dynamicTreeCutgydF4y2Ba(2014)。gydF4y2Ba
梅切勒,M.等。聚类:聚类分析基础和扩展。R包版本1-56。gydF4y2Bahttps://CRAN.R-project.org/package=clustergydF4y2Ba(2012)。gydF4y2Ba
张建军,张建军,张建军,张建军,张建军。基于gis的生物多样性研究进展[j]。R包版本3.3-7。gydF4y2Bahttps://CRAN.R-project.org/package=biomod2gydF4y2Ba(2016)。gydF4y2Ba
Barbet-Massin, M, Jiguet, F, Albert, C. H. & Thuiller, W.选择物种分布模型的伪缺席:如何,在哪里,有多少?gydF4y2Ba生态方法。另一个星球gydF4y2Ba。gydF4y2Ba3.gydF4y2Ba中文信息学报,327-338(2012)。gydF4y2Ba
李建军,李建军,李建军,等。物种分布模型的准确性评估:流行率、kappa和真技能统计(TSS)。gydF4y2Baj:。生态gydF4y2Ba。gydF4y2Ba43gydF4y2Ba, 1223-1232(2006)。gydF4y2Ba
Coetzee, b.w.t, Robertson, m.p., Erasmus, b.f.n, van Rensburg, b.j. & Thuiller, W.集合模型预测,在气候变化的影响下,非洲南部重要鸟类保护区对保护特有鸟类的效果将会降低。gydF4y2Ba水珠。生态。BiogeogrgydF4y2Ba。gydF4y2Ba18gydF4y2Ba; 701-710(2009)。gydF4y2Ba
Oksanen, J, Blanchet, F, Kindt, R, Legendre, P. & Minchin, R. vegan:社区生态包。R包版本2.0-10。gydF4y2Bahttps://CRAN.R-project.org/package=vegangydF4y2Ba(2013)。gydF4y2Ba
古格,陈永平,池上,M.和Sork, V. L.晚第四纪气候变化对谷栎遗传变异模式的影响,gydF4y2BaQuercus兜水母目gydF4y2Ba娘家姓的。gydF4y2Ba摩尔。生态gydF4y2Ba。gydF4y2Ba22gydF4y2Ba科学通报,3598-3612(2013)。gydF4y2Ba
Napier, J. D, de Lafontaine, G. & Hu, F. S.研究干旱胁迫下的基因组变异gydF4y2Ba云杉马里亚纳gydF4y2Ba人群。gydF4y2Ba生态。另一个星球gydF4y2Ba。gydF4y2Ba10gydF4y2Ba中文信息学报,9271-9282(2020)。gydF4y2Ba
福瑞斯特,B. R, Lasky, J. R, Wagner, H. H. & Urban, D. L.检测多位点适应与多基因型环境关联的方法比较。gydF4y2Ba摩尔。生态gydF4y2Ba。gydF4y2Ba27gydF4y2Ba, 2215-2233(2018)。gydF4y2Ba
privprive, F., Aschard, H., Ziyatdinov, A. & Blum, M. G. B.使用两个R软件包:bigstatsr和bigsnpr进行大规模全基因组数据的有效分析。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba中文信息学报,2781-2787(2018)。gydF4y2Ba
虚假发现率:一项新协议。gydF4y2Ba生物抑制剂gydF4y2Ba。gydF4y2Ba60gydF4y2Ba, kxw041(2016)。gydF4y2Ba
George, a.w., Visscher, p.m.和Haley, c.s.在复杂谱系中绘制数量性状位点:两步方差成分方法。gydF4y2Ba遗传学gydF4y2Ba156gydF4y2Ba, 2081-2092(2000)。gydF4y2Ba
VanRaden, p.m.等。北美荷斯坦公牛基因组预测的可靠性。gydF4y2Ba乳品科学gydF4y2Ba。gydF4y2Ba92gydF4y2Ba, 16-24(2009)。gydF4y2Ba
圣安东尼奥,n.n;詹宁克,j.l。一种低分辨率上位定位方法鉴定异源六倍体小麦染色体臂相互作用。gydF4y2BaG3gydF4y2Ba9gydF4y2Ba, 675-684(2018)。gydF4y2Ba
Cingolani, P.等人。一个用于注释和预测单核苷酸多态性(SnpEff)影响的程序。gydF4y2Ba飞(奥斯丁)gydF4y2Ba6gydF4y2Ba, 80-92(2012)。gydF4y2Ba
格拉博斯基,p.p.等。利用外显子组捕获测序数据研究柳枝稷开花时间与全基因组的关联。gydF4y2Ba新植醇gydF4y2Ba。gydF4y2Ba213gydF4y2Ba, 154-169(2017)。gydF4y2Ba
Schnable, p.s.等。B73玉米基因组:复杂性、多样性和动态。gydF4y2Ba科学gydF4y2Ba326gydF4y2Ba, 1112-1115(2009)。gydF4y2Ba
m.m accaferri等人。硬粒小麦基因组突出了过去的驯化特征和未来的改良目标。gydF4y2BaNat麝猫。gydF4y2Ba。gydF4y2Ba51gydF4y2Ba, 885-895(2019)。gydF4y2Ba
邹,C.等。高粱的基因组。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba10gydF4y2Ba, 436(2019)。gydF4y2Ba
VanBuren, R.等。异源四倍体埃塞俄比亚谷物苔麸的亚基因组稳定性和功能分化。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba11gydF4y2Ba, 884(2020)。gydF4y2Ba
霍夫迈斯特,b.t.等人。野生长寿多年生植物的基因组组装及体细胞遗传和表观遗传突变率gydF4y2Ba杨树trichocarpagydF4y2Ba。gydF4y2Ba基因组医学杂志gydF4y2Ba。gydF4y2Ba21gydF4y2Ba, 259(2020)。gydF4y2Ba
马拉诺,A.等。高质量的核桃染色体尺度组装(gydF4y2Ba胡桃regiagydF4y2BaL.参考基因组。gydF4y2BaGigasciencegydF4y2Ba9gydF4y2Ba[j],[2020]。gydF4y2Ba
致谢gydF4y2Ba
植物收集是与J. Randall(北卡罗莱纳植物园)合作进行的,通过种子成功计划,A. Stottlemeyer(俄勒冈州立大学)和USDA-NIFA生物技术风险评估资助计划,编号。(2010-33522-21703), T. Quedensley, M. Donahue, D. Schemske和J. M. Reyna。我们感谢Brackenridge野外实验室、Ladybird Johnson野花中心和junger实验室在植物护理和繁殖方面的支持。多纳休负责多元化小组的管理、宣传和维护。P. Duberney、S. Reeder、K. Turner、M. Carey、T. Arredondo、N. Ryan、B. Watson、B. Battershell、N. Albert、H. Wilson、L. Simon、J. Sanley、L. Vormwald、T. Bortnem、S. Hofmann、M. Iceberg、C. Lamb和T. Vugteveen也进行了实地调查。从J. G.门罗,D.胡佛,P. Edger, J. Lasky, E. Kellogg, J. Vogel, G. Sarath和J. Tuskan的建议帮助制作实验设计,测序策略和早期版本的文本。R. VanBuren, P. Edger, H. Zheng, D. Ware和L. Cattivelli提供了基因组比较信息。我们感谢HudsonAlpha基因组服务实验室装载Illumina X10测序运行。本研究得到了美国能源部奖励DESC0014156给T.E.J, DE-SC0017883给D.B.L., DE-SC0010743给K.M.D,大湖生物能源研究中心(奖励DESC0018409和DE-FC02-07ER64494)和生物能源创新中心(奖励DE-AC05-000R22725)的支持。资金由美国国家科学基金会PGRP奖IOS0922457和IOS1444533提供给T.E.J., IOS1402393提供给J.T.L.。美国能源部联合基因组研究所的工作由美国能源部科学办公室支持,合同号为DE-AC02-05CH11231。 The work conducted by the Joint BioEnergy Institute is supported by the Office of Science of the US Department of Energy under contract no. DE-AC02-05CH11231. The work conducted by Argonne National Laboratory is supported by the Office of Science of the US Department of Energy under contract DE-AC02-06CH11357. J.S. thanks T. Marsh for transferring his passion for ecological science to J.S. T.E.J. thanks K. Robertson for introducing him to prairie habitats and plant diversity.
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
贡献gydF4y2Ba
K.D.B’变为贬义词,m·萨哈L.E.B,司仪,K.M.D, D.B.L, D.S.R, J.G。T.E.J.和J.S.二人主持设计的研究。mit获得M.H。,j .江推力马力IV,酸处理,J.Z J.M.M.-R。,P。R., C.M.T., M.U., M.C., K.M.D. and D.B.L. contributed plant material and resources. J. Jenkins, C.P. and S.S. assembled and annotated the genome. J.B., A.R.B., P.A.F., F.B.F., J.D.J., D.B.L., J.L.-R., R.M., R.B.M., F.M.R. Jr, M. Saha, Y.W. and T.E.J. designed and executed field experiments. K.B., L.B., C.D., S.D., A.E., D.K., A.L., E.V.S., D.S., M. Sharma, R.S., A. Stewart, V.R.S., Y.T., J.W., X.W., M.W., Y.Y., M.Z. and R.A.W. conducted sequencing and data acquisition. J.T.L., A.H.M., S.M., J.D.N., A. Session, A. Sreedasyam, P.P.G., T.H., A.H., P.Q., C.A.S., G.A.W. and L.Z. conducted statistical and computational analyses. The manuscript was written by J.T.L., A.H.M., T.E.J. and J.S. with contributions from all authors.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
同行评议信息gydF4y2Ba自然gydF4y2Ba感谢Emily Heaton, Todd Michael, Ian Stavness和其他匿名审稿人对这项工作的同行评审所做的贡献。同行评审报告是可用的。gydF4y2Ba
出版商的注意gydF4y2Bab施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。gydF4y2Ba
扩展数据图和表gydF4y2Ba
图1基因组组装和注释。gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba、基因组邻近度(gydF4y2Ba一个gydF4y2Ba)及图书馆覆盖范围(gydF4y2BabgydF4y2Ba)证明v5释放物是一个非常完整的基因组,是目前最好的植物参考基因组之一(gydF4y2BacgydF4y2Ba),与玉米相比gydF4y2Ba128gydF4y2Ba硬质小麦gydF4y2Ba129gydF4y2Ba、黍gydF4y2Ba130gydF4y2Ba,画眉草gydF4y2Ba131gydF4y2Ba,杨树gydF4y2Ba132gydF4y2Ba、大豆gydF4y2Ba70gydF4y2Ba、棉花gydF4y2Ba47gydF4y2Ba、核桃gydF4y2Ba133gydF4y2Ba和草莓gydF4y2Ba46gydF4y2Ba。gydF4y2BadgydF4y2Ba在4向定位群体的两个杂交(标记数= 4,701)中,标记序之间的共线性是明显的。gydF4y2BaegydF4y2Ba,基因组注释统计呈现出与组装体一样完整的基因注释。gydF4y2BafgydF4y2Ba在我们的732个文库重测序小组中,观察到杂合度在<4到bb0 10%之间。gydF4y2BaggydF4y2Ba的几乎整个单拷贝基因组gydF4y2Bap . halliigydF4y2Ba与两个柳枝稷亚基因组都是同源的;淡蓝色的多边形代表亚基因组和gydF4y2Bap . halliigydF4y2Ba。唯一的例外是先前已知的在Chr. 03和Chr. 08上代表ρ重复的过度保留区域gydF4y2Ba64gydF4y2Ba。gydF4y2Ba
图2常见园林和生态型之间的表型和气候梯度。gydF4y2Ba
一个gydF4y2Ba这十个常见的花园跨越了柳枝稷的地理分布,并在我们的柳枝稷多样性小组中引起了非常不同的表型反应。对于每个花园,我们给出了地理参考位置和种植在那里的植物的一些基本数量遗传属性。gydF4y2BabgydF4y2Ba为了说明冬季死亡率的气候背景,我们提出了整个研究期间最低日温度的7天滚动平均值。线条颜色与颜色键相匹配gydF4y2Ba一个gydF4y2Ba。gydF4y2BacgydF4y2Ba为了研究每个花园的气候属性,我们对来自WorldClim的46个气候变量(变量命名为bio1-19)进行了聚类gydF4y2Ba22gydF4y2Ba)及ClimateNAgydF4y2Ba21gydF4y2Ba使用多样性小组的地理参考位置;标识符(左)和描述(右)伴随每一行。这七个簇,在热图中被分隔开来,由七个气候变量表示,这些气候变量与每个簇的第一个主成分特征向量(以粗体标记)最密切相关。gydF4y2BadgydF4y2Ba为了研究生态型进化,我们将多样性小组的每个成员概率地分配到三种生态型中的一种(gydF4y2BangydF4y2Ba高地gydF4y2Ba= 221,gydF4y2BangydF4y2Ba沿海gydF4y2Ba= 157,gydF4y2BangydF4y2Ba低地gydF4y2Ba= 129)使用一组形态学(gydF4y2BangydF4y2Ba= 16(2个花园)及质素(gydF4y2BangydF4y2Ba= 2)表型;区分生态型的线性判别函数在这里与两种定性生态型分类的特征向量一起提出。每个点代表在两个TX中生长的单一基因型gydF4y2Ba2gydF4y2Ba及MI花园(gydF4y2BangydF4y2Ba= 509)。线性判别分析。gydF4y2BaegydF4y2Ba定性生态类型评估的专家提出了TXgydF4y2Ba2gydF4y2Ba2019年的花园。的gydF4y2BaygydF4y2Ba-轴尺度是五类有序的,但点是抖动的,使观测的密度更明显。然后通过神经网络分类对点进行上色gydF4y2BadgydF4y2Ba。gydF4y2BafgydF4y2Ba其他16个变量(横跨2个花园)的负荷在相同的尺度和轴上绘制gydF4y2BadgydF4y2Ba。为了区分变量,我们将每个变量归为四组中的一组,分别代表叶片(深绿色)(3)、整株(红色)(1)以及这些变量的组合的变化。gydF4y2BaggydF4y2Ba中的标签的图例gydF4y2BafgydF4y2Ba,其中每个变量都在MI和TX中测量gydF4y2Ba2gydF4y2Ba花园。更详细的表型描述可在补充数据中找到gydF4y2Ba5gydF4y2Ba。gydF4y2BahgydF4y2Ba对于七个气候变量中的每一个,我们都校正了收集点与每个公共花园之间的气候距离。二次模型拟合(gydF4y2BargydF4y2Ba2gydF4y2Ba),给出了每个变量和生态型。gydF4y2Ba
图3亚种群和生态型之间的种群和数量遗传分化和进化。gydF4y2Ba
一个gydF4y2Ba,两两gydF4y2BaFgydF4y2Ba-每个亚种群按生态型组合之间的统计数据以及每个亚种群所有生态型之间的统计数据。gydF4y2BabgydF4y2Ba交叉聚结(Cross coalescence, RCCR)是定义发散的另一种方法。这里,将16个RCCR剖面的bootstrap转换为发生发散的生成时间。介绍了各个自举的统计数据。gydF4y2BacgydF4y2Ba,物理距离的链接-不平衡非线性函数和整个样本中标记物之间的预测相关系数。将每500bp区间的线性模型预测绘制为黑色开点;2-bp-interval意味着gydF4y2BargydF4y2Ba2gydF4y2Ba值是背景中的浅灰色点。gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba,群体遗传结构显示为结构变异的缩放和中心距离矩阵的主坐标(gydF4y2BadgydF4y2Ba)、存在-不存在变体(gydF4y2BaegydF4y2Ba)及SNPs (gydF4y2BafgydF4y2Ba),按图中亚种群分配的颜色编码。gydF4y2Ba3.gydF4y2Ba。gydF4y2BaggydF4y2Ba、Positions和−loggydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba值)的前2000个GWAS点击显示了2个花园,3个亚种群(颜色为gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba)和整体运行(黑分)。gydF4y2Ba
图4跨DNA、表达和数量性状的亚基因组偏差。gydF4y2Ba
一个gydF4y2Ba生物量snp遗传力差异(gydF4y2BahgydF4y2Ba2gydF4y2Ba)对每个花园逐个亚种群组合的亚基因组之间的估计。带空单元格的逐亚种群组合表明模型不收敛。gydF4y2BabgydF4y2Ba在这里进行的所有基因组分析中都存在亚基因组偏差。颜色表示使用的数据集。gydF4y2BacgydF4y2Ba,用于构建的计数和比率gydF4y2BabgydF4y2Ba,对变量进行更长的描述。gydF4y2BadgydF4y2Ba,非同义(gydF4y2BaKgydF4y2Ba一个gydF4y2Ba),同义的(gydF4y2BaKgydF4y2Ba年代gydF4y2Ba)和每个亚基因组相对于的四倍简并翻转变位率(4DTv)gydF4y2Bap . halliigydF4y2Ba。gydF4y2BaegydF4y2Ba的每个色仓中的基因数目之和gydF4y2BafgydF4y2Ba。gydF4y2BafgydF4y2Ba在基因组注释RNA-seq数据集中显示了每个组织的K > N(蓝色)和N > K(红色)的表达热图。gydF4y2Ba
补充信息gydF4y2Ba
补充图gydF4y2Ba
流式细胞术中估计倍性的门控策略的例子。gydF4y2Ba
补充数据1gydF4y2Ba
两个基因图谱的定位。遗传连锁群(LG)和厘根(cM)定位在前两列。v5基因组物理染色体和碱基对(bp)位置为第三列和第四列。标记的身份和遗传图谱在第五列和第六列(最后一列)找到。这两张地图是堆叠的,Noble地图(553377个标记)位于UGA地图(4252个标记)之上。gydF4y2Ba
补充数据2gydF4y2Ba
柳枝稷K、N亚基因组的同源性数据库gydF4y2Bap . halliigydF4y2Ba和gydF4y2Ba美国二色的gydF4y2Ba外围集团。这是一个带注释的blast格式文件,其中(子)基因组的每个成对组合都是堆叠的。除了标准blast格式列外,还提供了查询(1)和目标(2)的以下数据:基因组,基因ID (' ID '),染色体(' chr '),以及每个基因组中的基因“开始”(bp),“结束”(bp),“链”和基因“顺序”。最后两列是' orthology.network ',它定义了子图(正交群),和' hit. net '。类型'在这里指定了418,768个同源和58,430个同源的命中。gydF4y2Ba
补充数据3gydF4y2Ba
84个rna测序文库的元数据。在这里,我们提出了加入号,实验设计,覆盖率和相关统计数据。实验、组织和复制在“样品”栏中指定。“Library”列与补充数据10中的原始读取计数相匹配。SRA标识符可以在最后两列中找到。gydF4y2Ba
补充数据4gydF4y2Ba
732个DNA重测序文库的元数据。在这里,我们提出了加入编号,地理参考收集地点,亚种群分类,生态型分配,覆盖率和相关统计数据。SRA标识符在最后一列中显示。gydF4y2Ba
补充数据5gydF4y2Ba
模型规范、特征描述和变量重要性gydF4y2Ba在网上gydF4y2Ba生态型分类。用于训练的基因型(前两列),神经网络层(5-6列)和变量字典(4gydF4y2BathgydF4y2Ba列)从训练的神经网络模型中提取。LDA和DAPC变量加载伴随着神经网络结果。gydF4y2Ba
补充数据6gydF4y2Ba
在大西洋、海湾和中西部亚种群中,分别对627,563、523,501和541,889个GWAS命中点进行了GWAS峰值位置和效果的统计。物理染色体和位置见于前两列。亚种群的测试分类在第三、第四和第五列,混合效应估计(“Bhat”)、标准误差(“Shat”)和显著性(“log10bf”)在最后三列。gydF4y2Ba
补充数据7gydF4y2Ba
候选基因的渗入间隔和GWAS峰组合数据库。在GWAS峰值10kb以内或渗入区间内的所有基因及其基因组坐标(列1-5),以及发现显著关联的亚群体、统计检验、候选区间的起始和结束坐标以及候选基因与区间中心的物理距离(列6-10)。列11-13包含了加权SNPeff评分的亚种群特异性统计数据、缺失基因文库的比例以及候选基因100bp内(如果有的话)SVs的大小和类型。基因共表达模块(来自WGCNA)的身份被打印在14gydF4y2BathgydF4y2Ba列。在最近的开花时间GWAS论文中,那些与候选基因同源的基因被标记在15gydF4y2BathgydF4y2Ba列。gydF4y2Ba
补充数据8gydF4y2Ba
生物量和存活率的基因型和普通花园。第一列(' plant_id ')对应于补充数据4中的' plant_id '列。其他列名跟在garden ID _ phenotype后面。Garden id对应于扩展数据图2a中的第一列。表型要么是“生物量”(干生物量,g)生物量的自然对数“LOGBIOMASS”或生存“SRV”(0 =死,1 =活)。缺失值表明该植物不是在该特定地点生长的。当一个基因型的多个克隆重复在一个位点生长时,该值代表所有观察数据的平均值。因此,一些生存值是> 0或< 1。gydF4y2Ba
补充数据9gydF4y2Ba
基因序列祖先状态重建。文件格式为gzip压缩fasta,其中每个序列头是柳枝稷基因组中单拷贝基因的主要转录物ID。gydF4y2Ba
补充数据10gydF4y2Ba
rna测序原始计数。库(列)与元数据文件(补充数据3)中的库匹配。每行显示80,278个基因的计数。gydF4y2Ba
补充数据11gydF4y2Ba
来自MCMS运行的相对交叉聚结数据。对于运行中的每一步(行),计算出的代数(4gydF4y2BathgydF4y2Ba列)和相对交叉聚结(RCCR)。这些被转换成步骤之间的差异,发散时间在第七列中被标记。gydF4y2Ba
补充数据12gydF4y2Ba
柳枝稷各生态型的空间分布模型。它们被保存为压缩到单个存档中的R对象。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接从版权所有者处获得许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
洛弗尔,j.t.,麦昆,a.h.,马米迪,S。gydF4y2Baet al。gydF4y2Ba多倍体生物能源柳枝稷气候适应的基因组机制。gydF4y2Ba自然gydF4y2Ba590gydF4y2Ba, 438-444(2021)。https://doi.org/10.1038/s41586-020-03127-1gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-020-03127-1gydF4y2Ba
这篇文章是由gydF4y2Ba
柳枝稷丛枝菌根真菌应答microrna的鉴定gydF4y2Ba
BMC基因组学gydF4y2Ba(2022)gydF4y2Ba
柳枝稷(Panicum virgatum L.) Foxtail mosaic virus诱导的基因沉默(VIGS)gydF4y2Ba
工厂方法gydF4y2Ba(2022)gydF4y2Ba
柳枝稷穗部性状变异的遗传基础gydF4y2Ba
理论与应用遗传学gydF4y2Ba(2022)gydF4y2Ba
调查草原岛屿:阿巴拉契亚柳枝稷(Panicum virgatum L.)的遗传和性能gydF4y2Ba
遗传资源与作物进化gydF4y2Ba(2022)gydF4y2Ba
2个低地柳枝稷群体春绿、植株活力和生物量的qtl定位gydF4y2Ba
分子育种gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba