文摘
详细的知识在人类基因组的序列多样性如何影响表型多样性取决于一个全面和可靠的特征序列和表型变异。在过去的十年中,洞察这种关系已从whole-exome测序获得或大群丰富的表型数据的全基因组测序1,2。在这里,我们描述了全基因组测序分析英国生物库的150119人3。这就构成了一组高质量的变异,包括585040410个单核苷酸多态性,代表所有可能的人类单核苷酸多态性的7.0%,和58707036年indels。这大量的变异使我们能够描述选择基于序列变异在一个人口通过消耗排名分数沿基因组的windows。消耗排名分析表明,编码外显子代表一小部分地区基因组序列保护强。我们定义三个军团在英国生物库:一个大型英国爱尔兰队列,一个较小的非洲队列和南亚群组。单体型参考面板,允许提供可靠的归责的变异由三个或三个以上测序个体。我们确定了895055结构变异和2536688微卫星,组变异通常被排除在大规模的全基因组测序研究。使用这个强大的新资源,我们提供几个例子罕见变异影响大的特征关联未找到之前通过研究基于whole-exome测序和/或非难。
主要
英国生物库(UKB)3文档表型变异的500000名参与者在英国,有一个健康的志愿者的偏见4。UKB全基因组测序(WGS)财团是整个基因组测序的所有参与者的平均深度至少23.5×。这里我们报告第一个数据组成的一组庞大的序列变异,包括单核苷酸多态性(snp),短的插入或删除(indels),微卫星和结构变异(sv),基于WGS 150119人。所有变量调用进行共同在个人,允许比较一致的结果。由此产生的数据集提供了一个无与伦比的机会来研究人类的序列多样性及其对表型变异的影响。
先前的研究UKB产生了全基因组SNP数组的数据5和whole-exome测序(韦斯)数据6,7。尽管SNP数组通常只捕获的一小部分基因组中常见变异,当结合参考面板的WGS个人8在这些人,一个更大的变异可以通过污名调查。污名,然而,错过变体私人个人只在SNP类型化数组和提供不可靠的结果变异与运营商之间的单体型不足共享引用和归责集。差估算变异通常是罕见的,高度可变的或与复杂的单体型结构基因组区域,通常是由于结构性的变化。
韦斯区域主要局限于翻译,从而揭示了只有一小部分(2 - 3%)的序列变异在人类基因组中。是相对简单的分配功能变体编码蛋白质的区域内,但有充分的证据,变异外编码外显子功能也很重要9,解释了一大部分的遗传特征10。
大规模测序的努力通常专注于识别单核苷酸多态性和短indels。虽然这些是最丰富的变异类型在人类基因组中,其他类型,包括sv和微卫星,影响更多的碱基对,因此更有可能有一个功能的影响11,12。甚至重叠外显子是很难确定的sv韦斯由于更大的可变性的深度序列覆盖率韦斯研究比WGS研究因为捕获步骤的目标排序。微卫星多态1 - 6 bp的串联重复序列,也通常不检查在大规模序列分析研究。
这里我们强调的一些见解从这个巨大的新资源的WGS数据挑战或不可能确定从韦斯和SNP数组数据集。
单核苷酸多态性和indels
整个150119 UKB参与者的基因组测序的平均覆盖率32.5×(至少23.5×个体;补充图。1)使用Illumina公司NovaSeq测序机器解码遗传学(90667人)和维尔康姆基金会桑格研究所(59452人)。人伪随机从组中选择UKB参与者和分裂的两个序列之间的中心。变体中使用的所有150119个人被发现,13个人在重复测序,11个人撤回同意不时的测序分析和微阵列数据没有提供给我们的有135人,留下149960人进行后续分析。
顺序读取映射到人类基因组GRCh38参考13使用BWA14。单核苷酸多态性和短indels共同称为个人使用图像电传机15和GATK HaplotypeCaller16分别在655928639年和710913648年,结果变异。我们用几种方法来比较两个变体呼叫者的准确性,包括策划比较数据集17(补充表1和补充图。2),传播的等位基因在三人小组(补充表2和3),比较参数精度(补充表4)和韦斯数据比较(补充表5)。这些比较表明,图像电传机提供更精确的基因型。例如,尽管图像电传机变异减少7.7%,我们估计图像电传机叫做真阳性变异在三人小组,有9.4%多4.5%比GATK更可靠地将变异。所以我们限制后续分析图像电传机短变异的基因型,尽管进一步的见解可能获得共同探索这些调用集。包含假阳性的数量,图像电传机使用逻辑回归模型,分配每个变体得分(AAscore),预测概率,这是一个真正的积极。我们专注于643747446(98.14%)高质量的图象电传机变异,表明AAscore高于0.5,以下称为GraphTyperHQ。
美国大学医学遗传学和基因组学(ACMG)建议报告列表中可操作的基因型与疾病相关的基因的高度渗透和行之有效的干预是可用的18。我们发现4.1%的149960人携带一个可行的基因型在73个基因根据ACMG之一18v3.0。使用韦斯6和ACMG v2.0(59基因),据报道,2.0%的人携带一个可行的基因型,当限制我们的分析ACMG v2.0和相同的标准,我们发现基于WGS 2.5%,增加可操作的基因型检测的数量在一个大群体,在一定程度上,它可以有一个显著的影响社会疾病负担。
变异的数量确定每个个体的数量是40倍变异通过韦斯研究确定相同的UKB个人(表1;方法)。虽然被称为“韦斯”,我们发现,韦斯主要捕获编码外显子和最怀念变异的外显子转录但不翻译,失踪的72.2%和89.4%的(UTR) 5′和3′端非翻译区,分别。甚至目前策划的内部编码外显子编码19我们估计,10.7%的变异是由韦斯(表错过1)。手动检查缺失变异在韦斯认为,这些失踪是由于缺少覆盖在一些地区和基因分型过滤器。相反,几乎所有的变异与韦斯WGS发现(表1)。
功能重要的地区
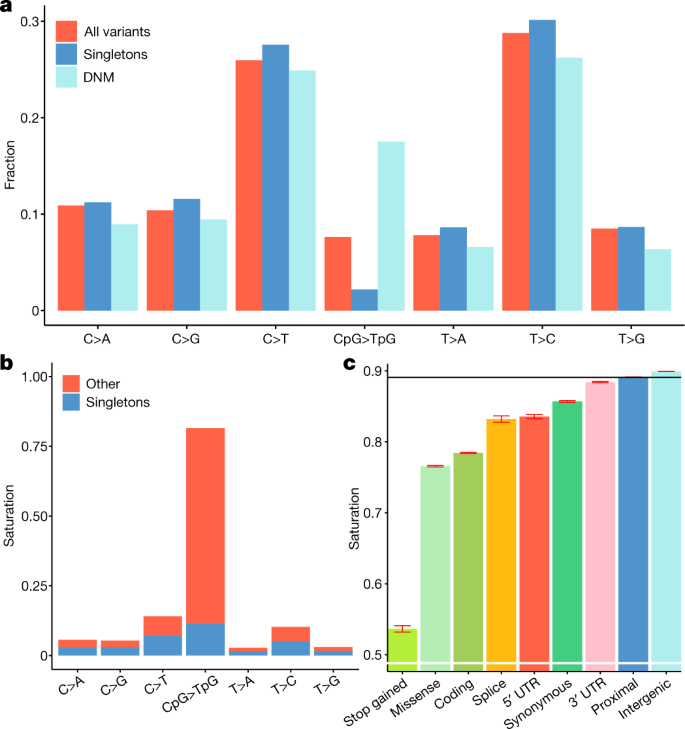
snp的数量在我们的研究发现对应于平均每4.8个基点,在区域的基因组可映射序列较短的读取。这相当于7.0%的理论上可行的检测单核苷酸多态性在这些地区(衡量饱和)。我们观察到所有可能的常染色体CpG > TpG变异的81.5%,其他的11.8%转换和只有4.0%的颠换(补充表6)。限制分析17345777种系常染色体CpG二核苷酸甲基化9过渡,我们观察到的变量在89.1%的甲基化论文认定。CpG突变是严重饱和(无花果。1)、转换与颠换比率(1.66)低于发现在较小的WGS集1和新生突变研究20.。
绝大多数的所有变体识别罕见的(补充表7),46.0%和40.6%的单核苷酸多态性和短indels,分别是单例(由一个测序个体),和96.6%和91.7%频率低于0.1%。推理的单体型和归责通常涉及识别变异将共享一个共同的祖先(由血统是相同的)。由于UKB WGS数据的规模,一个观察相同的等位基因无关的人并不总是意味着身份的后裔。明确表明,只有14%的高饱和CpG > TpG变异单例对象,与其他47%的单核苷酸多态性(无花果。1 b)。描述了这些复发现象在其他样本集之间使用共享罕见变异不同的子集2,21。我们使用一个新创突变组从2976年冰岛的三人小组20.直接评估复发,变异出现在设置和UKB必须至少来自两个突变事件。的194687冰岛新创突变,我们发现53859年UKB集(27.7%),提供一个直接的观察序列变异来自至少两个突变事件。正如预期的那样,我们发现CpG > TpG突变是最丰富的突变类重叠,由于他们的高突变率22和饱和度UKB设置(图。1 b)。
基因组变异的速度和模式是相关的丰富信息,塑造了基因组变异和选择的过程23。外显子组序列变异的数量被用来根据他们的公差等级基因丧失功能(LoF)和错义变异21,24。的关注外显子组是因为韦斯的可用性数据和编码变异的相对简单的功能解释。保护跨广泛的物种25是用来推断选择的影响超出了外显子组,利用广泛的突变积累了数百万年。然而,这些统计数据是只有部分信息对序列保护特定的人类26。人类的序列变异27,28可以用来描述人类保护,但需要大样本大小准确推断,尽可能多的减少突变单独比不同物种对人类。
大量饱和CpG > TpG在甲基化变异论文认定在大型韦斯军团已经被用于识别基因组注释或位点的缺席可能表明消极的选择21,29日。与先前的报道21,我们看到不饱和stop-gain CpG > TpG变异比同义(无花果。1 c)。同义突变通常被认为是影响选择(中性)29日;然而,我们发现的CpG > TpG突变不饱和(85.7%)比那些基因间(89.9%)、支持假设人类的密码子使用的限制30.。
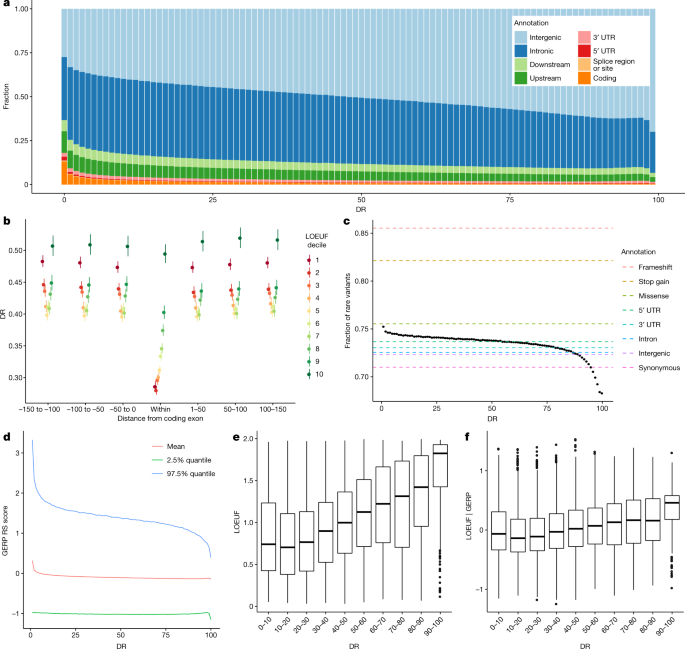
扩展这种方法中,我们使用序列变异数UKB寻求守恒的地区在500 - bp windows整个人类基因组。我们在背后的方法论构建上下文相关的公差得分(上)27,将它应用到更大的数据集。更具体地说,我们在每个窗口列表的数量变异,这个数字比预期多聚体核苷酸组成的七聚物的窗口和分数与整个基因组序列变异及其突变类。然后我们分配一个等级(消耗排名(DR))从0(大多数损耗)到100(损耗)为每个500 - bp窗口。正如预期的那样,编码外显子有一个低博士(博士的意思= 28.4),但大量的非编码区域显示更低博士(损耗),包括非编码调控元素。博士在最低的1%的地区,13.0%是编码和87.0%非编码,群体中拼接,UTR、基因上游和下游地区(图。2)。博士随距离编码外显子(无花果。2 b)。后删除编码外显子,在1%的最低和最高的地区博士得分,我们看到GWAS的变异传播的3.2倍和0.4倍,分别(表2博士),这表明分数可以在GWAS之前是一个有用的分析31日。编码9候选人独联体监管元素比预期更有可能偶然发现在耗尽博士(低)地区(表3)。值得注意的是,候选人独联体监管元素位于转录起始点的距离,也就是说,近端enhancer-like promoter-like序列,浓缩在枯竭的地区比远端enhancer-like序列。
一个,部分地区陷入功能注释类,通过运用基因映射的定义,作为博士的函数。b博士分数作为一个外显子和LOEUF等分距离的函数。误差线代表95%可信区间,使用正常的近似计算,每个基因治疗(n范围在1206 - 1848年间)作为一个独立的观察。c,部分罕见(四个或更少的运营商)变体作为博士的函数。d,平均GERP的分数在500 - bp windows作为RS博士的函数,拒绝替换。e,fLOUEF (e)和LOEUF | GERP (f在博士)的函数e和f,中间栏显示平均,铰链是第25和第75分位数,黑点显示异常,须延长到1.5四分位范围从铰链到最大或最小值。博士的基因数量或观察的范围如下:n(0 - 1)= 1234,n(0.1 - -0.2)= 3202,n(0.2 - -0.3)= 4474,n(0.3 - -0.4)= 3888,n(0.4 - -0.5)= 2476,n(0.5 - -0.6)= 1384,n(0.6 - -0.7)= 863,n(0.7 - -0.8)= 522,n(0.8 - -0.9)= 374,n(0.9 - 1)= 427。
地区下强烈的负面选择预计将有更大比例的罕见变异(FRV;这里定义为变种最多由四个WGS个人)比其余的基因组28。我们观察到更大的FRV博士在最贫地区(< 5)比最不枯竭地区>博士(95年):74.8%和69.1%(图2摄氏度和补充图。3)。这也是当限制只有一个非编码区(74.6%比69.2%)。使用带注释的FRV编码变异作为参考(图。2摄氏度),我们发现最贫地区(< 1)博士FRV与错义突变(75.5%)。
总的来说,之间存在弱相关博士和种间保护以基因组进化速率分析(GERP)25(线性回归r2= 0.0050,两面P< 2.2×10−308;无花果。二维)。我们发现了一个更强的相关性和GERP博士编码外显子(线性回归r2= 0.0498,两面P< 2.2×10−308),而不是外部(线性回归r2= 0.0012,两面P< 2.2×10−308博士),这表明之间的相关性和GERP主要是由于大多数高度保守的元素,如编码外显子,用来计算GERP 36哺乳动物,与较弱的相关性在保守区域。
博士是否消极的选择反映了人类不是被GERP,我们聚合相比,外显子和博士从Gnomad LOEUF度量21(无花果。2 e)。LOEUF措施不耐受LoF突变的基因,但它不衡量不耐受外编码外显子。我们发现博士与LOEUF(线性回归r2= 0.085,两面P< 2.2×10−16)。LOEUF与基因常染色体显性遗传21;符合这一点,我们发现相关博士(线性回归r2= 0.0027,两面P= 6.6×10−12据人类)和常染色体显性基因32(补充表8)。造型LOEUF度量GERP的函数和提取残差的线性,我们得到一个衡量人类特有的LoF不耐受(LOEUF | GERP)。我们发现博士与LOEUF | GERP(线性回归r2= 0.024,两面P< 2.2×10−16;无花果。2 f博士),这表明人类序列约束不被GERP的措施。我们在上博士相比27,这是一个衡量序列约束类似于这里介绍的,和CADD33,特征34和LINSIGHT35措施的功能影响,结合跨物种保护(扩展数据图。1)。使用跨物种保护的约束指标形成一个关联块(CADD GERP,特征和LINSIGHT)不与博士和上相关块(补充表9)。博士得分最低的地区显示类似的浓缩在所有指标(扩展数据图。1)。总体而言,我们的研究结果表明,博士可以用来帮助识别基因组区域约束下整个基因组,因此提供了一个宝贵的资源,确定非编码序列的功能的重要性。
多个军团在UKB
许多GWAS36使用UKB数据都是基于一个子集5那些自认为“英国白人”的409559名参与者。更好的利用价值UKB的更广泛的参与者,我们定义了三种组包括450690个人(补充表10),基于遗传聚类的微阵列基因型通知自称种族和监督祖先推理(方法)。最大的群体,XBI(扩展数据图。3),包含431805个人,包括409559年规定英国白人的99.6%,加上之前增派约23900个人被排除在外,因为他们不确定是英国白人(其中13000人确认为“白色的爱尔兰”)。我们相信这个扩展将增加电力协会研究,但没有详细研究这组是否有其他潜在的优点或缺点。主成分分析与WGS 132000 XBI个人数据(方法),基于460万位点,揭示了一个非常精细的地理分化的英国爱尔兰群岛基因库(扩展数据图。2)。
我们定义了两个其他军团基于血统:非洲(XAF;n= 9633;扩展的数据图。4)、南亚(XSA;n= 9252;扩展的数据图。5)(图。3得了)。37598 UKB个人不属于XBI, XAF或XSA被分配到组超视距(他人)。WGS XAF队列的数据代表非洲序列变异的一个最全面的调查,与报道的出生地,其成员包括44个国家的31个撒哈拉沙漠以南的非洲大陆(扩展数据图。4)。由于非洲人口相当大的遗传多样性,以及由此而产生的连锁不平衡的模式的差异,XAF队列可能证明有价值的精细定位协会信号由于多个强烈相关变异XBI或其他非非洲人群的识别。
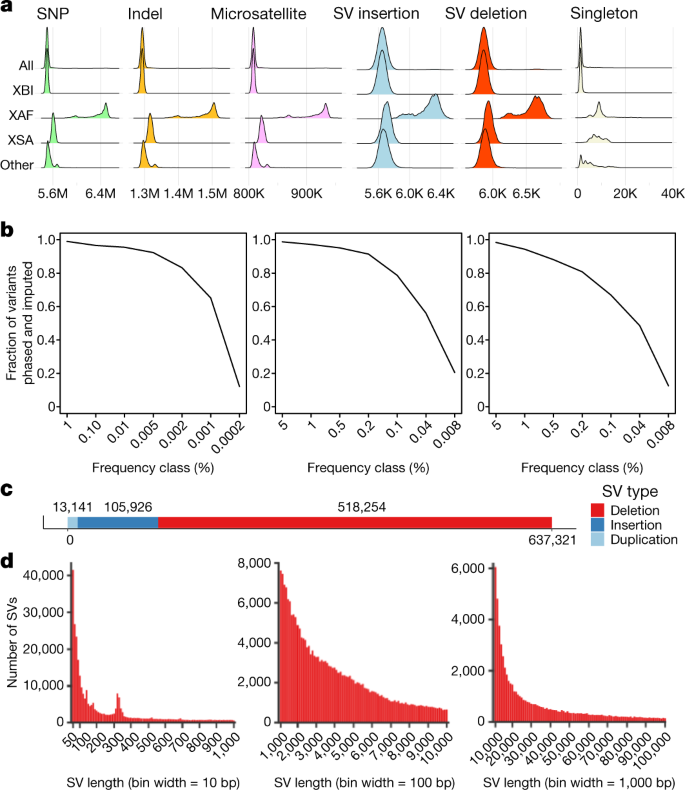
一个,WGS样本的数量分析表型在我们的研究中。b,UMAP情节产生的前40的主成分UKB参与者,彩色的自我报告的种族:蓝色阴影下的民族标签的白色类别(XBI),个人(XAF)红色色调为黑色和绿色色调南亚人(XSA);全彩色的传说,知道了补充图17。c、联合频谱之间的变异20号染色体上所有成对的人群。d- - - - - -f、特点的XBI队列在大不列颠和爱尔兰所示:单件的数量由个人在XBI队列作为出生地的函数(d);由行政区划逼供的亲戚的平均数(e);UKB评估中心的位置和周围的人口估计分数招募UKB (f)。单件计数和逼供的亲戚的数量的差异可能是由于密度采样UKB评估中心附近的个人生活。图3 d-f K.H.S.M.
我们走过GraphTyperHQ变异与外显子注释和发现,平均而言,大约在30个人是罕见的纯合子(小等位基因频率小于1%)LoF突变纯合状态和杂合的罕见的LoF数的中位数是24 /个人。我们在19105年发现罕见的LoF变异基因,在这2017个基因纯合的载体罕见LoFs(个人n= 5102)。明显不同数量的纯合子LoFs航空公司被发现在人群之间,XSA有最大的纯合子分数LoF运营商(扩展数据图。6 b)。XSA队列的显著特点是高基因组近亲繁殖,可能由于同系交配37,尤其是在认同巴基斯坦人38(扩展数据图。6)。
平均而言,个人进行另外的3410510个snp等位基因和indels(无花果。4),每单倍体基因组。更多的变异通常发现在欧洲以外的个人出生(扩展数据图。7),因为人类的参考基因组主要是来自欧洲血统的人13。XAF个人携带最大数量的替代等位基因(图。4)。我们建造cohort-specific DRs,发现XAF显示更大的损耗比XBI外显子左右和XSA(扩展数据图。8)。很大程度上由于变异个体采样的数量,平均每个个体的单件数量变化明显的祖先(无花果。4)。因此,个人从XBI XAF和XSA军团平均有1330个,单变量,分别为9623人和8340人。在XBI,单件计数(无花果。3 d)表明,预期每个基因组的新的变体发现数量仍然是实质性的,但不同地域,平均在英格兰北部大约1000和2000年在英格兰东南部。这种模式在很大程度上是用密集采样的一些地区(图来解释。3 e, f),而不是地区血统的差异。
归责
我们能够可靠地归咎于变异成整个UKB样本集到非常低的频率(图。4 b)。我们估算分阶段基因型,它允许分析取决于阶段识别等复合LoF杂合的。一个参考面板是用来在UKB转嫁到所有参与者的基因组,但结果分别给出了三个军团(补充表11)。这个参考面板可用于准确的归责个人从英国和许多其他人群。在XBI人群中,98.5%的变异频率高于0.1%和65.8%的变异频率的0.001 - -0.002%(代表3 - 5 WGS运营商)可以可靠地估算(无花果。4 b和补充图13)。变体也估算精度高XAF和XSA军团(无花果。4 b),97.5%和94.9%的变异频率1 - 5%,56.6%和48.9%的变异由3 - 5测序个体可以估算,分别。大量的变异,尤其是罕见的,比在使用替代估算所有军团归罪面板5(补充表12)。因此可能UKB参考面板提供了一个最好的选择将基因型为非洲和南亚人口样本。
我们发现了很多临床上重要的变体,可以估算的数据集。这些包括rs63750205 (NM_000518.5 (HBB): 110 c。* _ * 111 del)的3′UTRHBB,一个变种在ClinVar注释39作为β-thalassaemia可能致病。rs63750205-TTA 0.005%频率的估算XBI队列(0.98)的归责信息和相关较低意味着南达科他州微粒体积2.88。(95% CI 2.43 - -3.33,两面P= 1.5×10−36,χ2)。
XSA队列,我们发现rs563555492-G,先前报道40错义变体在PIEZO1XAF XSA(频率= 3.65%,0.046%和0.0022% XBI)与高血红蛋白浓度,影响0.36他(95% CI 0.28 - -0.44,两面P= 8.9×10−19,χ2)。变体可以估算与归责XSA人口信息为0.99。
XAF队列,我们发现stop-gain变体rs28362286-C (p.Cys679Ter)PCSK9(XBI XAF频率= 0.93%,0.00016%和0.0070% XSA)估算XAF群体污名信息为0.93。变异降低南达科他州non-HDL胆固醇0.92。(95% CI 0.75 - -1.09,两面P= 2.3×10−26,χ2)。我们发现一个纯合子的变体的载体,有2.5 s.d non-HDL胆固醇低于总体均值,61岁,似乎是健康的。
SNP和indel协会韦斯
我们测试了估算图像电传机SNP / indel,微卫星和SV协会共有8180数据集,1291年和459年在XBI表型,分别XAF和XSA军团。我们对特征的突出例子,可能不容易发现韦斯或SNP数组数据,从三个例子的SNP和indel协会XBI队列。
第一个是一个协会之间的XBI队列罕见variant-rs117919628-A(频率= 0.32%;归责信息0.90),在启动子区域GHRH编码生长激素释放激素,接近它的一个转录开始女王的高度(南达科他州效果=−0.32 (95% CI 0.27 - -0.36),双面P= 1.6×10−39,χ2)。GHRH神经肽是由下丘脑分泌的刺激生长激素(GH)的合成。我们注意到影响美国南达科他州(−0.32或−3厘米)的rs117919628大于任何变体在大高度全基因组关联研究(GWAS;大约1200相关的变体)41,42,43。除了降低高度,rs117919628-A与降低血清胰岛素生长因子1 (IGF1);南达科他州效果=−0.36 (95% CI 0.32 - -0.40),双面P= 3.2×10−58,χ2)。生产刺激IGF1的GH和介导GH对儿童成长的影响,进一步支持的假设GHRH介导rs117919628-A的影响。由于它大约50个基点上游的位置GHRH5′UTR,这种变体不是针对UKB韦斯,也不是唯一的强烈相关变体rs372043631 (intronic)。rs117919628-A不是与rs763014119-C(没有个人携带的微小等位基因变异),先前报道44非常罕见的移码删除GHRH(Phe7Leufster2;频率= 0.0092%),与身高和IGF1水平降低(=−0.63年代高度影响。d (95% CI 0.36 - -0.89),双面P= 4.6×10−6;南达科他州IGF1效果=−0.74 (95% CI 0.49 - -0.99),双面P= 4.9×10−9,χ2)。
第二个例子是rs939016030-A,一种罕见的3′UTR必不可少的拼接速激肽受体基因的变体编码3 (TAC3;频率= 0.033%;c。* 2-1G > T NM_001178054.1和NM_013251.3)。没有找到这种变体在UKB韦斯45和都是两个高度相关的变体:一个intronic (rs34711498)和一个基因间(rs368268673)。的微小等位基因3′UTR必不可少的剪接变体,rs939016030-A,与后来月经初潮年龄,南达科他州的影响为0.57 (95% CI 0.41 - -0.74)或11个月。(两面P= 1.0×10−11,χ2)。罕见的编码变异TAC3及其受体TACR3已报告导致hypogonadotropic性腺机能减退46常染色体隐性遗传。然而,在UKB,协会的3′UTR拼接受体变异仅仅是由杂合子(大约1500人)没有检测到该等位。我们复制这一发现39360年的丹麦人,南达科他州的影响为0.70。(95% CI 0.34 - -1.06,频率= 0.05%,两面P= 0.00014,χ2)。
第三个例子是一种罕见的变异(rs1383914144-A;染色体的着丝粒附近频率= 0.40%)1 (1 q)开始,尿酸水平较低的同事(南达科他州效果=−0.43。(95% CI 0.40 - -0.46)或dl−0.58毫克−1(95% CI 0.54 - -0.62),双面P= 8.1×10−170,χ2),防止痛风(或= 0.36 (95% CI 0.28 - -0.46),双面P= 4.2×10−15,χ2)。第二个变种rs1189542743 4 Mb下游的染色体1 p强烈与rs1383914144 (r2= 0.68),收益率与尿酸类似的协会。在这个地区没有协会报道尿酸GWAS47。rs1383914144-A对尿酸的影响比任何变体的报道,在最新的GWAS荟萃分析的特征。我们复制这些发现冰岛人(rs1383914144-A频率= 0.47%;尿酸:两面P= 8.0×10−37,χ2南达科他州,效果=−0.51 (95% CI 0.43 - -0.59);痛风:两面P= 0.0018,χ2= 0.31 (95% CI 0.15 - -0.64))。
结构变异
我们确定了sv在每个使用外套48和这些变异与读相结合研究49和七个人的程序集50。我们基因分型结果895055 sv(无花果。4摄氏度)图像电传机50,其中637321人被认为是可靠的。
平均而言,我们认为7963 sv可靠/个人,4185删除和3778插入(图。4)。这些数字是与7439 sv /个人Gnomad-SV发现51,另一个短内容研究,但是大大小于22636优质sv发现读测序研究49,主要是由于重复插入和sv的代表名额不足的地区。sv显示类似的频率分布snp indels和类似的变异在群体分布(图。4)。
我们提出四个与sv表型关联的例子,在韦斯数据不容易发现。首先,一种罕见的(频率= 0.037%)14154 -英国石油删除,删除第一外显子PCSK9之前发现的使用读测序在冰岛人口和non-HDL胆固醇水平较低的关联49。有32颗WGS卫星运营商XBI队列(频率= 0.012%)和72个运营商XBI估算组(频率= 0.0087%)1.22其中。(95% CI 0.90 - -1.55) non-HDL胆固醇水平低于非承运人(两面P= 1.2×10−13,χ2)。
第二个例子是4160年英国石油公司删除(XBI频率= 0.037%),消除了从4300年到140年英国石油公司上游的启动子区域铝青铜基因编码蛋白。不足为奇的是,运营商删除有明显的低水平的血清白蛋白(南达科他州效果= 1.50 (95% CI 1.35 - -1.62),双面P= 9.5×10−118,χ2)。与特征相关的变体也与白蛋白水平;航空公司有低水平的钙和胆固醇:0.62他(95% CI 0.50 - -0.75,两面P= 2.9×10−22,χ2南达科他州)和0.45 (95% CI 0.30 - -0.59,两面P= 1.1×10−9,χ2),分别。
第三个SV的例子是16411 - bp删除(XBI频率= 0.0090%),删除最后两个外显子(4和5)GCSH,甘氨酸解理系统H蛋白质编码。运营商的删除在UKB明显更高水平的甘氨酸代谢组学数据集(南达科他州效果= 1.45 (95% CI 1.01 - -1.86),双面P= 1.2×10−10,χ2)。
最后一个例子是一个罕见的(XBI频率= 0.892%)754 - bp删除重叠的外显子6NMRK2编码产生烟酰胺核苷激酶2,从转录RNA中删除72个基点,对应于一个24个氨基酸在坐标系中删除翻译蛋白质。运营商的删除南达科他州0.22。(95% CI 0.18 - -0.27)早些时候绝经的年龄(两面P= 1.1×10−26,χ2)。附近是变体rs147068659,据报道,与这种特质有关52,南达科他州的影响为0.20 (95% CI 0.16 - -0.24)早期绝经的年龄(两面P= 2.0×10−20,χ2XBI队列中)。删除和rs147068659相关(r2= 0.67);后删除仍然显著(双面条件分析P= 6.4×10−8,χ2),而rs147068659不(两面P= 0.39,χ2),这表明缺失是导致变异位点。NMRK2主要是心脏和肌肉组织中表达53。在我们的心脏右心房组织的数据集,一个人一组169 RNA-sequenced个人是这个删除的载体。正如预期的那样,我们观察到表达下降的外显子6个人和增加的记录片段跳过外显子6(扩展数据图。9)。
微卫星通常被忽视
我们确认14321152等位基因在2536688使用popSTR微卫星位点54在150119 WGS个人携带,平均而言,810606年非引用微卫星等位基因。非引用等位基因的数量进行每个显示了一个类似的分布在UKB组其他变体类型在这项研究中(图特点。4)。微卫星是最快速变异的变异在人类基因组和遗传变异的来源,在GWAS通常被忽视。重复扩张已知与表型联系起来,包括脆性X综合征55。我们可以归罪于微卫星降到很低的频率(补充图4)在所有三个组,提供了一个估算微卫星的第一次大规模数据集。
我们组内的微卫星CACNA1A基因,编码电压门控钙通道subunit-α1 a。个人20或更多重复的微卫星通常患有终身影响大脑的条件,包括家族性偏瘫的偏头痛1型、2型和脊髓小脑的共济失调型癫痫、情景性共济失调6 (ref。56)。运营商在XBI群22份microsattelite对遗传性共济失调(重复在更大的风险频率= 0.0071%,或= 304,两面P= 1.1×10−31,χ2)。
我们也证实了一个协会之间的微卫星的3′UTR内DMPK,编码DM1蛋白激酶、肌强直性营养不良XBI队列。的表达DMPK已被证明是负相关与微卫星重复的数量吗57。肌强直性营养不良的风险增加而重复拷贝数的迅速增长与重复的数量由单个的或161个人携带39或多个重复(扩展数据图10补充表13)。
讨论
提供的数据集的全基因组测序大约150000 UKB参与者是前无古人的大小和提供最广泛的特征序列的一个人口的生殖系基因的多样性。我们一组广泛的序列变异特征在WGS个人,提供两套SNP和indel数据,以及微卫星和SV数据,变量的类经常不会在GWAS审问。单核苷酸多态性和indels 40倍的数量大于从韦斯相同的个体。即使在注释编码外显子,韦斯错过10.7%的变异,通过WGS发现。韦斯错过大部分基因组的其余部分,包括utr功能重要,启动子区域和外显子进行注释。这些地区的重要性以罕见的非编码序列变异的发现更大影响身高和初潮比任何变体GWAS中描述。
我们希望这里给出的分数博士是一个重要的资源识别基因组区域的功能的重要性,虽然进一步评估应采取了解它的属性,以及它如何影响比较其他措施保护和序列的约束。虽然编码外显子显然是在强大的净化选择,博士为代表的低分数,他们只代表一小部分地区低分数博士。这里描述的大规模测序,测序整个UKB持续努力,承诺将大大增加我们对非编码基因的作用和影响。当结合UKB广泛的表型多样性的特征,这些数据应该大大提高我们理解人类基因组变异和表型多样性之间的关系。
方法
数据集
UKB数据
UKB表现型和基因型数据收集从所有参与者获得知情同意。西北研究伦理委员会审查和批准了科学协议和操作规程(REC参考号:06 / MRE08/65) UKB。在这项研究中获得的数据以及研究UKB应用程序许可下进行数字24898,52293年、68574年和69804年。序列数据处理中描述补充讲义1 - 4,补充无花果。5 - 8和16日和17日补充表。
表型从UKB下载。8180、1291和459年表型XBI建造,分别XAF和XSA军团。这里给出的例子被选为值得注意的代表协会的例子。表型这里给出的处理,参照身份UKB数据展示,在提供补充表15。
冰岛的数据
痛风的样本集60冰岛,共有1740人,是通过多种渠道招募的。的一个子集,这些人一般用户anti-gout药物对应的解剖治疗化学分类系统类M04 (ATC-M04)。个人使用ATC-M04当时通过问卷调查确定进入遗传学项目指挥部提供的解码和Heahth条目的处方药的登记注册(2005 - 2020)或RAI评估和最小数据集(MDS)居民和疗养院(1993 - 2018)的申请者。此外,大约一半收到了痛风的临床诊断(274年国际疾病分类:ICD-9代码或icd -代码M10)在1984年和2019年之间在Landspitali,冰岛的国立大学医院,诊所,或在两个风湿病或这样的诊断是由检查拉伊和MDS医疗记录。
血清尿酸水平的血液样本95086冰岛人从Landspitali获得,国立大学医院的冰岛和冰岛医学中心(Laeknasetrid)实验室Mjodd (RAM)在1990年和2020年之间。血清尿酸的规范化使用quantile-quantile规范化标准正态分布,然后调整性别、出生年份和年龄测量。对个人来说,多个测量是可用的,我们使用归一化值的平均值。血清尿酸水平测定的酶反应中尿酸酶氧化尿酸盐尿囊素和过氧化氢,而过氧化物酶的帮助下,染料,形成一个颜色复杂,可以用光度计在波长670纳米。
所有参与的人捐献的血液签署知情同意。使用第三方参与者的身份加密系统和监控通过冰岛数据保护权威。这项研究是冰岛国家生物伦理委员会的批准(批准号vsn - 15 - 023)后评价冰岛数据保护权威。所有数据处理符合数据保护机构的指令(PV_2017060950ÞS)。
RNA序列数据分析经冰岛数据保护权威和冰岛的国家生物伦理学委员会(没有。VSNb2015030021)。
丹麦的数据
从丹麦献血者提供研究数据(dbd)61年。dbd基因研究已通过丹麦国家卫生研究伦理委员会(nvk - 1700407)和丹麦首都地区的数据保护办公室(p - 2019 - 99)。
SNP与图像电传机indel调用
所有输入压缩图像电传机运行之前,我们预处理reference-oriented对齐地图(塞)指数(CRAI)指数提取一个大型的单一文件包含所有与样本CRAI索引条目ID 50 kb窗口(1 kb填充在每个地区)的所有样品。对于每个区域,然后,我们创建了一个切碎CRAI为每个样本处理大文件的相应区域,大大减少CRAI索引条目阅读。
此外,我们创建了一个序列缓存使用的seq_cache_populate FASTA文件的引用。pl脚本分布式samtools 1.9。在每个地区,我们复制相应的序列缓存到本地磁盘,用它阅读补习文件通过设置“REF_CACHE”环境变量。
我们跑图像电传机(v2.7.1)使用“基因型”子命令。完整的命令,我们跑的格式:
图像电传机基因型$ {UKBIO_REFERENCE}——sam = $ {sams} sams_index = $ {CRAI_TMP} / crai_filelist。txt avg_cov_by_readlen = ${保险}——地区= ${地区}——线程= ${线程}——冗长
在哪里UKBIO_REFERENCE是GRCh38_full_analysis_set_plus_decoy_hlaFASTA序列文件,地空导弹是所有输入BAM /补习的列表文件,CRAI_TMP是一个路径切碎CRAI本地磁盘上的文件,保险是覆盖除以每个输入文件的读取长度,地区基因分型地区吗线程使用线程的数量。
SNP和indel调用GATK给出补充注5。详细的对比图像电传机,GATK叫集提供了补充Notes 6和7,补充无花果。9 - 12和补充表21页。
运行时间
所有工作运行使用12芯保留60 GB的RAM。大约1%的工作岗位重新使用24芯和120 GB内存保留的。几个工作需要更多的内核和内存,与单个作业完成48芯和1000 GB的RAM。总保留CPU时间集群580万CPU时间和总有效CPU计算时间为500万小时。这些数字的差异的解释是,并非所有的核心的程序不能利用所有在同一时间。
SV调用外套和图像电传机
我们运行了一个SV型管道类似于我们先前应用于49962年冰岛人50。总之,我们跑外套48v1.6发现sv型组中的所有150119人。我们还创建了一组高度自信共同sv(归罪信息高于0.95,频率在0.1%以上)从我们先前的研究使用Illumina公司短的读取50和牛津纳米孔读数据49。最后,我们推断出一组sv从六个公开使用dipcall组装数据集62年如前所述,50。我们使用svimmer50合并不同的SV数据集,我们称为结果SV使用图像电传机502.7.1版。通过将数据从读数据和高质量的组件,我们可以调用比只使用短读真正的sv,特别是对常见的sv。
总共有895054个变异,其中637321变种被注解为“通过”。提出了变异数变异注释的图像电传机为“通过”,除非另有注明。
大多数的sv删除(81.3%);然而,我们观察到仅略比插入和删除重复平均每个(图。4)。这是因为许多插入源读取和组装数据,因此许多稀有插入失踪。删除通常更容易发现短内容数据。个人属于XAF队列携带更多sv比其他人群(无花果。4 b)。
归责和逐步
UKB样本SNP芯片与一组定制Affymetrix芯片,英国BiLEVE公理,在第一个50000人63年数组,Affymetrix UKB公理64年在剩下的参与者。我们使用现有的远程定相的SNP chip-genotyped样本5。
我们excluced SNP和indel序列变异中,至少50%的样品没有报道(《GQ》得分= 0),如果哈迪温伯格P值小于10−30或者杂合的过剩是小于0.5或大于1.5。
我们用剩下的序列变异和long-range-phased芯片数据来创建一个单体型参考面板使用内部工具1,65年。然后我们估算单体型参考面板变异到chip-genotyped样品使用先前描述的内部工具和方法1,65年。
归责由估计,对于每一个单体型,单体型与单分享在单体型参考面板中,给每个单体型单体型的重量。这些权重以及等位基因概率为每个单体型与李单体型参考面板允许归责和斯蒂芬斯66年模型类似于一个用于IMPUTE2 (ref。67年)。单体型权重估计是基于long-range-phased芯片单。
迭代序列变异逐步由冠阶段在每个测序样品基于其他测序样品和上次迭代估计阶段。估算基因型以及原始的基因型,一起加权估计新等位基因单体型的概率。归责完成如上所述。
我们计算分析r2评分(L1oR2)平方相关(r2值)的原始基因型电话,与每个样本的基因型估算不包括样品的原始基因型的污名输入。
批测序中心的影响被发现在两个原始基因型(补充表21)和估算数据(补充表22)。
识别功能的重要地区
识别功能的重要地区,我们开始通过估算是否可靠basecalls有望在基因组中每个站点。在每个碱基对序列覆盖在GRCh38计算为每个1000随机选择的个体。在每个碱基对,然后我们计算均值和停工的覆盖1000人。碱基对平均覆盖率至少20和南达科他州的报道最多12被认为是可靠的碱基对。只在GraphTyperHQ变体(AAscore > 0.5)被认为是在分析。
复发性突变和光谱下饱和
使用从上面SNP变体的分类,我们计算比率的SNP GraphTyperHQ落入每个类别。然后,我们做了相同的限制单件,即计算每个突变类单例落入的比例。相比之下,我们计算每个SNP类的分数从194687年策划的所有181258个SNP新创突变在2976年冰岛三人小组20.。我们使用这种分布突变类计算过渡到tranversion比率在每种情况下。
复发性突变的列表,我们加入这个列表与GraphTyperHQ新创突变。
饱和突变类
我们限制我们的分析上面描述的可靠的碱基对和分组碱基对及其补充和考虑每个或T基地基因组突变的机会T >, T > C或T > G突变。同样,我们认为每个G或C基地作为一个潜在的C > C > G和C > T突变,将C > T拆分为两类基于CpG是否发生在上下文。然后我们计算饱和度的观察突变GraphTyperHQ除以突变的机会在可靠的碱基对。分别计算了对常染色体和染色体x 95%独联体使用正常的近似二项分布计算,将每个站点作为一个独立的观察。
甲基化在生殖系的网站
我们确定网站GRCh38中甲基化的生殖细胞系使用编码亚硫酸氢全基因组测序9人类睾丸和卵巢的数据样本。更准确地说,我们使用示例ENCFF946UQB和ENCFF157ZPP睾丸和ENCFF561KYJ ENCFF545XYI和ENCFF515OOQ卵巢。
我们假设甲基化链对称,计算每个CpG甲基化率二核苷酸由制表的数量在给定的组织类型读取支持甲基化或non-methylation在每个二核苷酸,对一个给定的组织类型的所有样品求和,然后计算读取的部分甲基化的支持。
我们认为一个网站在参考基因组甲基化CpG二核苷酸的种系如果其甲基化比率至少是0.7在睾丸和卵巢,并结合深度至少20为卵巢、睾丸和30或10倍数量的样本在每个组织类型。这导致了17902255年CpG的列表(17345777常染色体)二核苷酸,35804510(34691554常染色体)CpG > TpG突变的机会。
在甲基化CpG地点饱和
为每一个潜在的CpG > TpG在甲基化位点,我们评估了其最重要的潜在后果预测变量的影响68年v . 100。对于多个这样的后果,我们选择了按字母顺序。我们也分类基于上述功能分类。对于每个类,我们估计饱和度作为功能类的变体的比率GraphTyperHQ除以突变的机会。95%独联体使用正常的近似二项分布计算,将每个站点作为一个独立的观察。
消耗排名
我们跟着方法类似于先前发表的研究27。消耗一个变体得分计算了一组重叠的基因组中500 - bp windows 50个基点的步长。49104026 500 - bp windows中至少450 bp被认为是可靠的碱基对被认为是进行进一步分析。我们统计出现的次数可能七聚物(H)和中央碱基对的次数在七聚物观察SNP (S),在第一组重叠的窗口。考虑到区域在基因组突变模式69年基因组,我们一分为二成两个互斥的子集,内部和外部C > G-enriched区域(补充表12在裁判。69年)。S:比H被解释为预期的突变率七聚物,分别为每个两个子集。对于每一个窗口,然后我们计算变量的观察值(O),然后减去预期数量的变异(E),鉴于其七聚物。这种差异是期望值除以根号((O−E) /√E)。我们从分析那个窗口的平均AAscore低于0.85窗口内的变异。这些((O−E) /√E)数字排序和窗口我th损耗最低分数是100年分配一个博士(我−0.5)/n,在那里nwindows的总数。
计算限制博士军团,我们应用同样的方法限制序列变异存在于每个XBI, XSA和XAF同志们。
协会测试
我们检测与定量特征基于BOLT-LMM实现的线性混合模型70年。我们使用BOLT-LMM leave-one-chromosome计算出残差,然后使用简单线性回归测试协会。我们使用逻辑回归测试序列变异和二进制特征之间的联系。下我们测试变异为协会添加剂模型使用预期的等位基因数作为协变量量化特征和集成在可能的基因型二进制特征。测序状态(无论是个人的WGS个人)和其他可用的个人特征与特征模型中还包括:性别、年龄和主要组件(20 XBI和XAF 45 XSA)调整人口分层。关联分析和XAF XSA种族样本大小的不到10000人,因此被直接用线性回归,而不是BOLT-LMM。使用的校正因子的拦截每个回归分析。
我们使用连锁不平衡(LD)得分回归占分布数据集的通货膨胀由于神秘的亲缘和人口分层71年。使用110万个变异,我们退化χ2统计数据从我们的GWAS对LD拦截作为校正因子得分和使用。尺度效应基于leave-one-chromosome残差缩小,我们新他们基于110万个变异的收缩中使用LD得分回归。补充表24全基因组关联分析列表统计数据给出每个协会的信号。曼哈顿情节,quantile-quantile情节和直方图inverse-normal-transformed值的协变量调整后,年龄、性别和40个主要组件中可以找到补充无花果。14和15分别对定量和二进制表型。轨迹情节尿酸和初潮协会中可以找到补充图16。人类32和开放的目标72年注释的基因提供了补充表14。
没有统计方法被用来预先确定协会测试的样本大小。所有协会报告估算基因型。作为比较,协会也直接进行基因型。协会测试执行的直接的基因标记,协变量的同一组被使用,除了测序状态(所有人都测序),以及测序中心(解码,桑格主要,桑格先锋)作为协变量。补充表25展示了原始之间的相关性和估算基因型和批处理效果为测序中心XBI队列。
一个人被认为是一个等位基因的载体,如果个体进行等位基因的概率至少0.9。协会的分析仅限于标记中至少一个(XAF XSA), 2 (XBI,估算数据集)或3 (XBI原始基因型)个人带着轻微的等位基因。协会测试往往局限于个人的数据集的一个子集,该协会分析进一步限制在那些至少有标记载体之一的个人联想测验。估算数据集,协会测试进一步限制在那些标记与归责信息> 0.5和原始基因型将这些标记与测序信息> 0.8 (ref。1)。
定义组
大多数研究UKB数据到目前为止一直在进行的409554年英国白人的个人创造的UKB英国白人的基础上自我认同和集群遗传主成分来自微阵列基因型5。像一些最近的研究44,73年,74年,我们希望利用UKB的多样性。为了达到这个目标,我们定义了三个军团基于最常见的血统确定参与者中,使用的组合(1)均匀歧管近似和投影(UMAP)提供的40个基因主成分降维UKB,和(2)掺合料分析监督五个参考人口和自我报告的种族的信息。
定义三个军团,我们跟着以前的工作75年并应用UMAP 40遗传UKB提供的主成分。UMAP进行R使用UMAP: UMAP v0.2.3()使用默认参数,值得注意的是,n_neighbours 15和min_dist 0.1。UMAP放置在一个二维的个人潜在的空间有几个集群和细丝。这些结构显示对应自称种族(补充图17)。
提供一个单独的祖先,我们可以用通知的解释UMAP集群,我们从监督掺合料叠加的结果58分析UKB微阵列基因型(补充部分掺合料),使用五个训练人口从1000人基因工程8从犹他州:CEU(北欧),慢性乙肝(汉族在北京),ITU(印度在英国泰卢固语),图像的基本单位(秘鲁首都利马)和套(尼日利亚伊巴丹、约鲁巴语)。我们观察到一个明确的对应UMAP坐标和祖先交办的比例掺合料(补充无花果。18和19)。使用此信件和指导下自我种族信息,我们定义了军团通过手动描述地区UMAP潜在空间仅限于个人British-Irish祖先(XBI;n= 431805),南亚祖先(XSA;n= 9633)和非洲血统(XAF;n= 9252)。这使得37598人与基因型数据,分配给任意队列,我们称之为超视距(其他)。祖先的分布估计使用掺合料在一年的四个军团(补充图18)。
XBI队列最系统的区别和流行的UKB-defined英国白人是我们加入XBI大约12500个人识别爱尔兰那样白。这显然是合理的,因为已知种群的地理和文化距离英国和爱尔兰。更重要的是,我们的分析(和以前的出版物)清楚地揭示他们之间广泛的基因流动的证据。因此,爱尔兰的主要基因集群出现在主成分分析作为一个集成组件的连续变异在英国(扩展数据图。2),和其他没有明确分开。XBI队列的另一个主要区别相对于常用的英国白人,是增加了约10900人没有确定英国白人,但是我们推导出祖先区别British-Irish个人。我们注意到更大的规模XBI队列应该提供更多的统计能力检测genotype-phenotype关联。进一步详细地描述队列定义补充笔记16 - 22和补充无花果。-。
报告总结
进一步研究信息设计是可用的自然研究报告摘要与本文有关。
数据可用性
WGS,基因型数据,分阶段和估算数据可以通过访问UKB研究分析平台(RAP):https://ukbiobank.dnanexus.com/landing。研究分析平台是开放的研究人员列出合作者UKB-approved访问应用程序。摘要统计信息对GWAS可以下载,为了科学目的,https://www.decode.com/summarydata/。分数是博士作为补充数据。丹麦的汇总统计数据复制表型可以在请求O.B.P.冰岛的汇总统计数据复制表型可以勾选上请求K.S.人类参考基因组GRCh38可以发现:http://ftp.1000genomes.ebi.ac.uk/vol1/ftp/technical/reference/GRCh38_reference_genome/。瓶中基因组WGS样品可以发现:https://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/。运用:https://m.ensembl.org/info/data/mysql.html。
代码的可用性
下面列出了我们使用公开可用的软件(url)结合上面的描述算法。BamQC (v 1.0.0):https://github.com/DecodeGenetics/BamQC。图像电传机(v2.7.1):https://github.com/DecodeGenetics/graphtyper。GATK资源包(v4.0.12): gs: / / genomics-public-data /资源/ / hg38 / v0广阔。Svimmer (v0.1):https://github.com/DecodeGenetics/svimmer。popSTR (v2.0):https://github.com/DecodeGenetics/popSTR。Dipcall (v0.1):https://github.com/lh3/dipcall。轮胎式龙门吊工具(v3.8.4):https://github.com/RealTimeGenomics/rtg-tools。bcl2fastq (v2.20.0.422):https://support.illumina.com/sequencing/sequencing_software/bcl2fastq-conversion-software.html。Samtools (v1.9):http://www.htslib.org/。Samblaster (v0.1.24):https://github.com/GregoryFaust/samblaster。我们用R (v3.6.0;https://www.r-project.org/)广泛分析数据和创建的情节。
引用
配图,d . f . et al。冰岛人口的大规模的全基因组测序。Nat,麝猫。47,435 - 444 (2015)。
Taliun, d . et al . 53831不同基因组的测序从NHLBI TOPMed程序。自然590年,290 - 299 (2021)。
Sudlow, c . et al .英国生物库:一个开放存取资源识别多种复杂疾病的原因中、老年。科学硕士。12e1001779 (2015)。
炒,等。比较社会人口和健康相关特征的英国生物库参与者与一般人群。点。j .论文。186年,1026 - 1034 (2017)。
Bycroft, c . et al。英国生物库资源深表现型和基因数据。自然562年,203 - 209 (2018)。
范·胡特,c . v . et al .外显子组测序和描述英国生物库的49960人。自然586年,749 - 756 (2020)。
Szustakowski, j . d . et al .推进人类遗传学研究和药物发现通过英国生物库的外显子组测序。Nat,麝猫。53,942 - 948 (2021)。
Auton, et al。全球参考人类遗传变异。自然526年,68 - 74 (2015)。
摩尔,j . e . et al .扩大百科全书在人类和小鼠基因组DNA的元素。自然583年,699 - 710 (2020)。
Gazal, s . et al .功能体系结构低频变异突出强度的负选择编码和非编码注释。Nat,麝猫。50,1600 - 1607 (2018)。
Weischenfeldt, J。西蒙斯,O。,Spitz, F. & Korbel, J. O. Phenotypic impact of genomic structural variation: insights from and for human disease.Nat,启麝猫。14,125 - 138 (2013)。
Sudmant, p . h . et al。一个集成的地图2504年人类基因组结构变异。自然526年,75 - 81 (2015)。
施耐德,v . a . et al .评价GRCh38和新创单倍体基因组装配演示了持久的参考装配的质量。基因组Res。27,849 - 864 (2017)。
李,h &杜宾,r .快速和准确的读与burrows - wheeler变换。生物信息学26,589 - 595 (2010)。
Eggertsson,惠普et al .图像电传机使人口规模的使用pcr pangenome图表。Nat,麝猫。49,1654 - 1660 (2017)。
麦肯纳,a . et al .基因组分析工具包:一个MapReduce框架分析下一代DNA测序数据。基因组Res。20.,1297 - 1303 (2010)。
祖克,j . m . et al .开放资源准确的基准测试小变体和引用调用。生物科技Nat。》。37,561 - 566 (2019)。
米勒,d . t . et al . ACMG科幻v3.0列表报告的二次研究在临床外显子组和基因组测序:美国大学的政策声明的医学遗传学和基因组学(ACMG)。麝猫。地中海。23,1381 - 1390 (2021)。
邓纳姆,等。一个集成的百科全书在人类基因组中DNA的元素。自然489年57 - 74 (2012)。
Halldorsson b . v . et al .人类遗传学:描述复合诱变的影响通过sequence-level遗传图谱。科学363年eaau1043 (2019)。
Karczewski, k . j . et al . 141456年从变异突变约束谱量化人类。自然581年,434 - 443 (2020)。
琼森,h . et al . 15220冰岛人的全基因组序列多样性的特征。科学。数据4170115 (2017)。
Seplyarskiy v . b . et al .人口测序数据揭示的突变过程在人类生殖细胞系。科学373年,1030 - 1035 (2021)。
豪夫里洛,j . M。,Pedersen, B. S., Layer, R. M. & Quinlan, A. R. A map of constrained coding regions in the human genome.Nat,麝猫。51,88 - 95 (2018)。
库珀、通用汽车等分布和强度的约束在哺乳动物基因组序列。基因组Res。15,901 - 913 (2005)。
胡贝尔,c, D。,Kim, B. Y. & Lohmueller, K. E. Population genetic models of GERP scores suggest pervasive turnover of constrained sites across mammalian evolution.公共科学图书馆麝猫。16e1008827 (2020)。
di Iulio j . et al .定义的人类基因组非编码基因的多样性。Nat,麝猫。50,333 - 337 (2018)。
杜克勒,N。,Mughal, M. R., Ramani, R., Huang, Y.-F. & Siepel, A. Extreme purifying selection against point mutations in the human genome. Preprint atbioRxivhttps://doi.org/10.1101/2021.08.23.457339(2021)。
Agarwal, i & Przeworski m .饱和突变在人类健身效果CPG网站。eLife10e71513 (2021)。
Dhindsa, r S。,Copeland, B. R., Mustoe, A. M. & Goldstein, D. B. Natural selection shapes codon usage in the human genome.点。j .的嗡嗡声。麝猫。107年,83 - 95 (2020)。
Sveinbjornsson, g . et al .加权序列变异增加基于注释的全基因组关联研究。Nat,麝猫。48,314 - 317 (2016)。
道斯,R。,Lek, M. & Cooper, S. T. Gene discovery informatics toolkit defines candidate genes for unexplained infertility and prenatal or infantile mortality.NPJ染色体组。地中海。48 (2019)。
Rentzsch, P。威滕,D。,Cooper, G. M., Shendure, J. & Kircher, M. CADD: predicting the deleteriousness of variants throughout the human genome.核酸Res。47D886-D894 (2019)。
Ionita-Laza,我。Mccallum, K。,Xu, B. & Buxbaum, J. D. A spectral approach integrating functional genomic annotations for coding and noncoding variants.Nat,麝猫。48,214 - 220 (2016)。
Gronau,我。,Arbiza, L., Mohammed, J. & Siepel, A. Inference of natural selection from interspersed genomic elements based on polymorphism and divergence.摩尔。杂志。另一个星球。30.,1159 - 1171 (2013)。
Sirugo G。,Williams, S. M. & Tishkoff, S. A. The missing diversity in human genetic studies.细胞177年26-31 (2019)。
Nakatsuka: et al。发现特定人群疾病有关的基因在南亚的承诺。Nat,麝猫。49,1403 - 1407 (2017)。
Arciero、大肠等。精细的人口结构和人口巴基斯坦裔英国人的历史。Commun Nat。127189 (2021)。
它,m . j . et al . ClinVar:公共档案馆的解释临床相关的变体。核酸Res。44D862-D868 (2016)。
太阳,问:et al。分析生物标志物特征在不同的英国生物库参与者识别关联错过了以欧洲为中心的分析策略。j .的嗡嗡声。麝猫。67年,87 - 93 (2022)。
Yengo, l . et al .荟萃分析的全基因组关联研究身高和体重指数∼700000人的欧洲血统。嗡嗡声。摩尔,麝猫。27,3641 - 3649 (2018)。
Marouli、大肠等。罕见和低频编码变异改变成人身高。自然542年,186 - 190 (2017)。
Asgari, et al。积极选择FBN1错义变异降低高度秘鲁人。自然582年,234 - 239 (2020)。
巴顿,a。R。,Sherman, M. A., Mukamel, R. E. & Loh, P.-R. Whole-exome imputation within UK Biobank powers rare coding variant association and fine-mapping analyses.Nat,麝猫。53,1260 - 1269 (2021)。
麦卡锡,s . et al .参考小组64976个单基因型污名。Nat,麝猫。48,1279 - 1283 (2016)。
Topaloglu, a . k . et al . TAC3 TACR3突变家族hypogonadotropic性腺机能减退揭示神经激肽B的一个关键的角色在中央控制的繁殖。Nat,麝猫。41,354 - 358 (2008)。
锡、et al。目标基因、变异、组织和转录途径影响人类血清尿酸盐的水平。Nat,麝猫。51,1459 - 1474 (2019)。
陈,x et al .外套:快速检测生殖系和癌症的结构变异和indels测序应用程序。生物信息学32,1220 - 1222 (2016)。
Beyter, d . et al . 3622年读测序冰岛人提供了洞察结构变异的作用在人类疾病和其他特征。Nat,麝猫。53,779 - 786 (2021)。
Eggertsson,惠普et al . GraphTyper2使人口规模的基因结构变异使用pangenome图表。Commun Nat。105402 (2019)。
柯林斯,r . l . et al .结构性变化参考医疗和种群遗传学。自然581年,444 - 451 (2020)。
露丝,k . s . et al .遗传洞察人类卵巢衰老生物学机制管理。自然596年,393 - 397 (2021)。
GTEx财团。Genotype-Tissue表达式(GTEx)项目。Nat,麝猫。45,580 - 585 (2013)。
Kristmundsdottir, S。,Sigurpálsdóttir, B. D., Kehr, B. & Halldórsson, B. V. popSTR: population-scale detection of STR variants.生物信息学33,4041 - 4048 (2017)。
Verkerk, a . j . et al .鉴定的基因(FMR-1)包含一个CGG重复重合与断点集群地区表现出脆性X综合征的长度变化。细胞65年,905 - 914 (1991)。
罗,x et al .临床严重CACNA1A等位基因影响突触功能和神经退行性变的不同。公共科学图书馆麝猫。13e1006905 (2017)。
卷起,D。,Lemieux, D., Taneja, K. & Puymirat, J. Decreased levels of myotonic dystrophy protein kinase (DMPK) and delayed differentiation in human myotonic dystrophy myoblasts.Neuromuscul。Disord。11,728 - 735 (2001)。
亚历山大·d·H。,Novembre, J. & Lange, K. Fast model-based estimation of ancestry in unrelated individuals.基因组Res。19,1655 - 1664 (2009)。
Szustakowski, j . d . et al .推进人类遗传学研究和药物发现通过英国生物库的外显子组测序。Nat,麝猫。53,942 - 948 (2021)。
Sulem, p . et al .识别低频变异与痛风和血清尿酸水平。Nat,麝猫。43,1127 - 1130 (2011)。
汉森,t . f . et al . dbd基因组群,未来的全面的资源整合和时序分析的遗传,环境和生活方式因素影响献血者的健康。BMJ开放9e028401 (2019)。
李,h . et al . synthetic-diploid基准精确variant-calling评估。Nat方法。15,595 - 597 (2018)。
北斗七星,l . v . et al。小说对吸烟行为的遗传学,肺功能,和慢性阻塞性肺疾病(英国BiLEVE):英国生物库的遗传关联研究。和出版的《柳叶刀》杂志上。地中海。3,769 - 781 (2015)。
威尔士,S。,Pe一个kman, T., Sheard, S. & Almond, R. Comparison of DNA quantification methodology used in the DNA extraction protocol for the UK Biobank cohort.BMC基因组学1826 (2017)。
香港,et al。检测共享的后裔,远程逐步和单体型归责。Nat,麝猫。40,1068 - 1075 (2008)。
李:&斯蒂芬斯,m .建模连锁不平衡和确定使用单核苷酸多态性数据重组热点。遗传学165年,2213 - 2233 (2003)。
豪伊,b . N。,Donnelly, P. & Marchini, J. A flexible and accurate genotype imputation method for the next generation of genome-wide association studies.公共科学图书馆麝猫。5e1000529 (2009)。
麦克拉伦、w . et al .运用变异效果预测。基因组医学杂志。17122 (2016)。
琼森,h . et al .父母的影响对人类生殖系新创突变三人小组从1548年冰岛。自然549年,519 - 522 (2017)。
Loh, P.-R。et al。高效的贝叶斯混合模型分析增加与大群体力量的联系。Nat,麝猫。47,284 - 290 (2015)。
Bulik-Sullivan, b . k . et al . LD得分回归混杂有别于polygenicity全基因组关联研究。Nat,麝猫。47,291 - 295 (2015)。
Ghoussaini, m . et al .打开目标基因:trait-associated基因系统标识的使用大规模的遗传学和功能基因组学。核酸Res。49D1311-D1320 (2021)。
35 Sinnott-Armstrong: et al .遗传学血液和尿液生物标志物在英国生物库。Nat,麝猫。53,185 - 194 (2021)。
王,问:et al .罕见的变异对人类疾病的贡献在281104年英国生物库外。自然597年,527 - 532 (2021)。
Diaz-Papkovich,。,Anderson-Trocmé, L., Ben-Eghan, C. & Gravel, S. UMAP reveals cryptic population structure and phenotype heterogeneity in large genomic cohorts.公共科学图书馆麝猫。15e1008432 (2019)。
确认
我们感谢UKB的参与者。UKB测序的WGS 450000个人,包括150119年这里描述已经由UKB WGS财团组成的英国政府的研究和创新机构,英国研究和创新(UKRI),通过基金产业战略挑战,威康信托基金会和安进制药公司,阿斯利康、葛兰素史克、强生(Johnson & Johnson)。欢迎信任桑格研究所进行DNA测序和解码遗传学。
作者信息
作者和联系
财团
贡献
本文作者是B.V.H. K.S.,来自H.P.E.输入,K.H.S.M.,O.E.,D.F.G., O.T.M., G.M., U.T., A.H., H.J. and P.S. K.H.S.M. and A.H. defined the cohorts. O.E. and H.J. identified functionally important regions. F.J. and U.T. were responsible for laboratory operations. DNA sequencing was performed by D.N.M., S.S., K.K. and O.T.M. Sample isolation was performed by E.S. and J.S. G.M. was responsible for the sequence analysis pipeline, developed by B.V.H., A.G., P.I.O., M.A., S.T.S., F.Z. and S.A.G., and run by G.T.S. B.V.H., H.P.E., H.Hauswedell, G.P., S.K., G.H. and S.A.G. developed analysis tools. Association analysis was performed by B.V.H., M.O.U., A.O., B.O.J., S.K., B.D.S., D.B., V.T., U.S. and P.S. Phenotypes were defined by M.O.U., V.T., G.T., I.J., T.R., H.Holm, H.S. and P.S. SNP and SV genotyping was performed by H.P.E., P.I.O. and B.S. Microsatellite genotyping was performed by S.K. Data analysis was performed by B.V.H., H.P.E., H.Hauswedell, G.P., A.O., O.A.S., G.S. and K.N. RNA sequencing data were analysed by G.H.H., supervised by P.M. D.F.G. supervised association data and DR analysis. Figures were drawn by M.T.H. and K.H.S.M. H.J., A.J.G., I.O. and P.J. collected clinical data in Iceland. O.B.P., C.E., S.B., S.R.O. and the DBDSGC collected clinical data in Denmark. The study was supervised by B.V.H. and K.S. All authors agreed to the final version of the manuscript.
相应的作者
道德声明
相互竞争的利益
B.V.H.,H。P。E., K.H.S.M., H.Hauswedell, O.E., M.O.U., G.P., M.T.H., A.O., B.O.J., S.K., B.D.S., O.A.S., D.B., G.H., V.T., A.G., P.I.O., F.Z., M.A., S.T.S., B.S., S.A.G., G.T.S., G.H.H., G.S., K.N., U.S., D.N.M., S.S., K.K., E.S., G.T., F.J., P.M., I.J., T.R., H.Holm, H.S., J.S., D.F.G., O.T.M., G.M., U.T., A.H., H.J., P.S. and K.S. are employees of deCODE genetics/Amgen.
同行评审
同行审查的信息
自然由于瑞安层和其他匿名审稿人(s)为他们的贡献的同行评审工作。同行审查报告是可用的。
额外的信息
出版商的注意施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
扩展数据图1平均分数在500个基点windows作为损耗军衔的函数。
一个CADD,b特征,c上,d,LINSIGHT。绿线代表平均分数,蓝色和红色线95。
扩展数据图2
地理分布载荷的前四个主成分的PCA XBI人口。
扩展数据图3
Cartogram-pies表明个人出生在每个国家的比例(名称显示在派)XBI队列。馅饼放置大约根据他们的国家在世界地图上的立场。灰色和白色的方块分别代表了海洋和陆地。
扩展数据图4 Cartogram-pies表明个人出生在每个国家的比例(名称显示在派)XAF队列。
馅饼放置大约根据他们的国家在世界地图上的立场。灰色和白色的方块分别代表了海洋和陆地。
扩展数据图5 Cartogram-pies表明个人出生在每个国家的比例(名称显示在派)XSA队列。
馅饼放置大约根据他们的国家在世界地图上的立场。灰色和白色的方块分别代表了海洋和陆地。
扩展数据图6功能丧失。
一个,每个样本相关性LoF基因的数量和比例的基因纯合性的运行。阴影区域代表95%置信区间。b纯合基因功能丧失(LoF)的数量,样品。数的纯合子基因注释高影响频率< 1%。结果提出了XBI、XAF XSA扣除个人认同巴基斯坦,个人自认为巴基斯坦的XSA队列等等。
扩展数据图7替代等位基因的地区。
数字在括号中地区名称下方显示计数个体全基因组测序的出生地。国家分配给区域是几乎相同的分类显示在队列统计图派人物,除了所有欧洲地区合并成一个地区在这个图。竖线下面密度曲线代表第0,25日,50,第75和第100百分位数。
扩展数据图8博士作为距离的函数编码外显子分区由LOEUF22十分位数。
结果显示分别为整体数据集(所有)和个体群,XBI, XAF XSA。
扩展数据图9 NMRK2 RNA分析。
一个,覆盖块RNA-sequenced读取从心脏组织从169年基因NMRK2心脏组织样本。一个人是754个基点的航母删除描述NMRK2灰色矩形,包括外显子6。承运人(蓝色)的RNA-coverage相比低外显子6位数的报道非承运人(绿色)。阴影是删除。b,直方图的分数RNA-sequenced跳过第6外显子片段NMRK2所有碎片调整施主能级的外显子5受体部位的外显子6或外显子7。野生型个体的平均分数碎片跳过0.035和0.57 754个基点的承运人删除。
扩展数据图10优势比肌强直性营养不良的风险的函数在微卫星重复长度的DMPK 3 '端非翻译区。
运营商至少39.7副本的微卫星重复主题有一个162倍增加肌强直性营养不良的风险。误差线代表95%的置信区间,n = 431079。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
Halldorsson,帐面价值,Eggertsson, H.P., Moore, K.H.S.et al。英国生物库的150119人基因组的序列。自然607年,732 - 740 (2022)。https://doi.org/10.1038/s41586 - 022 - 04965 - x
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586 - 022 - 04965 - x
本文引用的
多通道生物医学人工智能
自然医学(2022)
数据库的5305名健康韩国个人揭示基因对东亚人口和临床意义
实验与分子医学(2022)
非编码变异破坏组织的监管元素HK1引起先天性高胰岛素症
自然遗传学(2022)
全基因组序列分析> 66000人的血脂水平
自然通讯(2022)
饱和的地图常见基因变异与人类身高有关
自然(2022)