摘要gydF4y2Ba
DNA甲基化是一个基本的表观遗传标记,控制基因表达和染色质组织,从而为细胞身份和发育过程提供了一个窗口gydF4y2Ba1gydF4y2Ba.目前的数据集通常只包括甲基化位点的一小部分,并且通常基于在培养中发生巨大变化的细胞系或包含未指定的细胞混合物的组织gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba.在这里,我们描述了一个基于深度全基因组亚硫酸氢盐测序的人类甲基组图谱,允许对从205个健康组织样本中分类的39种细胞类型的数千个独特标记进行片段级分析。相同细胞类型的复制具有99.5%以上的同一性,证明了细胞识别程序对环境扰动的鲁棒性。图谱的无监督聚类概括了组织个体发生的关键要素,并确定了自胚胎发育以来保留的甲基化模式。在单个细胞类型中唯一未甲基化的位点通常位于转录增强子中,并包含组织特异性转录调控因子的DNA结合位点。独特的高甲基化位点是罕见的,并且在CpG岛、Polycomb靶点和CTCF结合位点上富集,这表明在形成细胞类型特异性染色质环方面具有新的作用。该图谱为基因调控和疾病相关遗传变异的研究提供了必要的资源,并为液体活检提供了大量潜在的组织特异性生物标志物。gydF4y2Ba
主要gydF4y2Ba
理解相同的DNA序列如何在不同类型的细胞中被不同地解释是生物学的一个基本挑战。基因表达,DNA可及性和染色质包装是公认的细胞表型的基本决定因素。DNA甲基化是一种稳定的表观遗传标记,它支撑着细胞身份的终身维护。gydF4y2Ba
现有的人类DNA甲基化数据集存在很大的局限性。多项研究已经确定了甲基组在胚胎发育、分化、癌症或其他环境中的特征gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba已经依赖于Illumina BeadChip平台,该平台仅限于预定义的45万或86万CpG甲基化位点子集,仅占人类基因组中约3000万CpG位点的3%gydF4y2Ba10gydF4y2Ba.此外,通过独立测量每个CpG位点,这样的分析忽略了DNA甲基化发生在区块的协调模式,DNA甲基化的关键功能单位gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba.gydF4y2Ba
大多数DNA甲基化分析主要针对大块组织,因此排除了少数细胞类型的研究,如组织常驻免疫细胞、成纤维细胞或内皮细胞,而其他分析的培养细胞可能包含体外引入的非生理甲基化模式gydF4y2Ba13gydF4y2Ba.作为部分解决方案,最近的研究利用来自全组织的单细胞RNA测序数据来鉴定在特定细胞类型中表达的标记基因,然后鉴定出甲基化与表达反相关的特定CpGs。这些可以用于阵列甲基组来解卷散装组织和评估细胞类型组成或样品纯度gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,但对于在液体活检中鉴定罕见细胞的贡献可能不够准确。对人类甲基组的一些研究确实使用全基因组亚硫酸氢盐测序(WGBS)分析了分离的原代细胞,但其范围有限gydF4y2Ba2gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba.gydF4y2Ba
为了克服这些限制并准确地表征人类细胞甲基组,我们在平均测序深度为30× (6.62×或更大)的荧光激活细胞分选器(FACS)纯化的39个人类细胞类型组的群体中,使用成对端150碱基对(bp)长reads进行了深度全基因组测序,从新鲜分离的成人健康组织中获得。我们将整个基因组的甲基化模式合并为均匀甲基化的CpG位点块,并使用这些块来研究不同细胞类型的甲基化模式的变化。在这里,我们识别和描述了以组织或细胞类型特异性方式独特甲基化的基因组区域,提供了它们可能的生物学功能的小片段,并引入了一种片段级反卷积算法,其应用包括基于循环细胞无DNA甲基化的临床诊断。gydF4y2Ba
人类细胞类型甲基化图谱gydF4y2Ba
为了描述多种细胞类型的全基因组DNA甲基化,我们对来自137名自愿供体的代表77种原代细胞类型的205个样本进行了WGBS (150 bp长成对端读,平均深度至少为30倍)。这些被仔细分类并映射到人类基因组(hg19, hg38)。通过流式细胞仪、基因表达和DNA甲基化分析,平均样品纯度(即所需细胞类型的物质比例)超过90%。一些样品纯度较低(例如,结肠成纤维细胞78%,平滑肌细胞82%,内皮细胞86%或脂肪细胞87%)。样品分离和纯度估计的详细描述以及样品信息见补充表gydF4y2Ba1gydF4y2Ba,补充图。gydF4y2Ba1gydF4y2Ba- - - - - -gydF4y2Ba3.gydF4y2Ba而且gydF4y2Ba补充信息gydF4y2Ba.gydF4y2Ba
分析的细胞类型(图;gydF4y2Ba1gydF4y2Ba)代表了大多数主要的人类细胞类型,允许生理系统的复合视图(例如,胃肠道,造血细胞和胰腺),以及不同环境中相似细胞类型的比较(例如,组织驻留巨噬细胞)。gydF4y2Ba
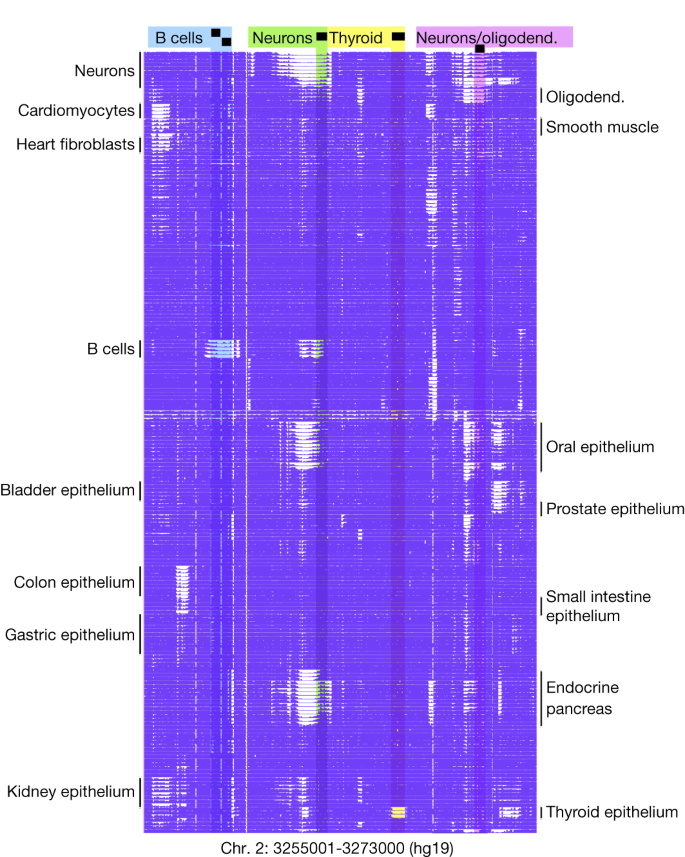
如图所示,205个甲基组在复制之间表现出极大的相似性,细胞类型之间以块状方式发生显著变化。gydF4y2Ba1gydF4y2Ba.我们试图识别特定细胞类型中甲基化差异的基因组区域,以阐明特定细胞类型的生物过程,定义细胞身份,并促进甲基化生物标志物的开发,以确定循环cfDNA片段的细胞起源gydF4y2Ba1gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba.gydF4y2Ba
我们开发了wgbstools,一个计算机器学习套件,以表示,压缩,可视化和分析WGBS数据(gydF4y2Bahttps://github.com/nloyfer/wgbs_toolsgydF4y2Ba).通过在多种条件下识别DNA甲基化模式的变化点,我们将基因组分割为7,104,162个不重叠的连续块。每个块横跨高度相关的CpG位点,在每个样本中类似甲基化,但可能在细胞类型中共变(gydF4y2Ba补充信息gydF4y2Ba).我们保留了2,783,421个至少3个cpg的甲基化区块,平均长度为544 bp(四分位间距(IQR) = 565 bp)和8个cpg (IQR = 5 cpg)。对这些紧密的基因组单元的稳健分析比单个CpG位点更直接,而且由于甲基化的区域性性质,可以被视为人类DNA甲基化的生物“原子”gydF4y2Ba12gydF4y2Ba.gydF4y2Ba
甲基化的个体间变异gydF4y2Ba
甲基化模式在不同的个体中非常稳健。对于大多数细胞类型,0.5%或更少的块显示不同供体之间的差异为50%或更多,而不同细胞类型样本之间的差异为4.9%(扩展数据图)。gydF4y2Ba4gydF4y2Ba).供体之间DNA甲基化的高度相似性与估计的个体间基因组序列变异性相当gydF4y2Ba22gydF4y2Ba.尽管50%的定义有些武断,但其他阈值(35-50%)显示出类似的趋势,具有0.5%或更少的变量块。在不同实验室的重复中观察到相似的个体间变异(补充表gydF4y2Ba1gydF4y2Ba).引人注目的是,对于细胞类型gydF4y2BangydF4y2Ba≥3个生物重复,197个样本中有195个(99%)与另一个复制(而不是来自同一供体的另一种细胞类型)的相似性最高。这些结果证明了制剂的可重复性,但也与以前的研究一致gydF4y2Ba6gydF4y2Ba,强调了DNA甲基化主要由细胞谱系和细胞类型特异性程序决定的基本生物学现象,而不是由遗传或环境因素决定的。gydF4y2Ba
甲基化记录了发育历史gydF4y2Ba
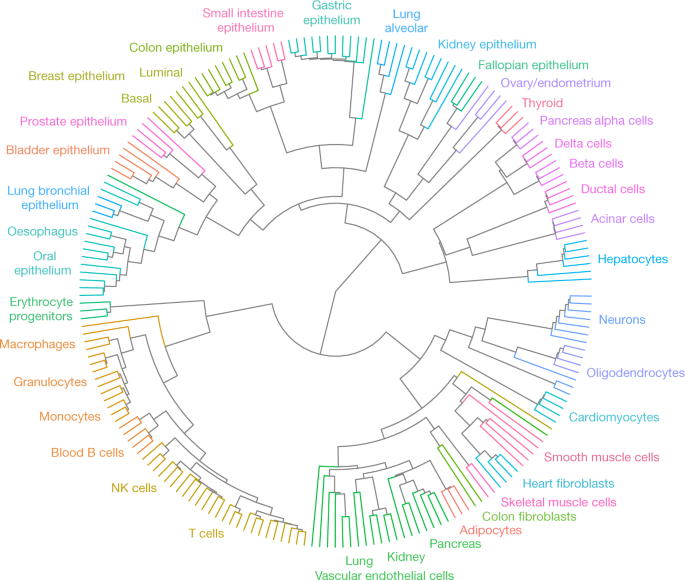
尽管DNA甲基化模式反映了细胞的功能特征,但它们也可以用来追踪细胞的发育历史。为了确定早期祖细胞的后代共享的模式,我们计算了至少四个cpg的区块内的平均甲基化,并选择了在所有样本中表现出最高变动性的区块(21,000个区块,前1%;补充表gydF4y2Ba2gydF4y2Ba).然后,我们使用一种无监督凝聚算法对所有205个甲基聚类,该算法迭代地识别并连接两个最近的样本,而不管它们的标签gydF4y2Ba23gydF4y2Ba.该分析系统地将相同细胞类型的生物样本分组(图2)。gydF4y2Ba2gydF4y2Ba),类似于纯化的人类血细胞的阵列聚类gydF4y2Ba6gydF4y2Ba.这支持了细胞分离的可重复性,并表明每种正常细胞类型的三到四次重复就足以推断其甲基化模式,用于生物标志物鉴定等实际应用。gydF4y2Ba
引人注目的是,由此产生的扇形图概括了人类组织之间谱系关系的关键要素。例如,胰岛细胞类型(α, β和δ),它们起源于相同的胚胎内分泌祖细胞gydF4y2Ba24gydF4y2Ba,密集地聚集在一起。与甲基组反映谱系而不是功能一致,胰岛细胞进一步与胰管和腺泡细胞聚集,然后与肝细胞聚集,它们与肝细胞共享内胚层起源。相反,内胚层来源的胰岛细胞不与外胚层来源的神经元聚集gydF4y2Ba25gydF4y2Ba尽管常见的组织特异性基因调控和胞吐机制gydF4y2Ba26gydF4y2Ba.gydF4y2Ba
其他的例子包括胃、小肠和结肠上皮细胞的聚集;聚类:所有血细胞类型的聚集;多种中胚层细胞类型包括血管内皮细胞、脂肪细胞和骨骼肌细胞的聚集。有趣的是,肺支气管上皮与食管和口腔上皮聚集在一起,而肺泡上皮与肠上皮聚集在一起,这与肺泡细胞谱系早期发育起源的证据一致gydF4y2Ba27gydF4y2Ba.gydF4y2Ba
一些甲基化模式在早期发育阶段形成的谱系中是常见的。例如,在早期内胚层衍生的上皮细胞中,892个区域未甲基化,而在中胚层和外胚层衍生的细胞中,892个区域甲基化(gydF4y2Ba方法gydF4y2Ba).我们认为这些在内胚层中去甲基化,衍生的细胞类型在几十年后仍保留这些模式(扩展数据图)。gydF4y2Ba5gydF4y2Ba).由于内胚层衍生物不具有共同的功能或基因表达,这提供了甲基化模式作为稳定谱系标记的另一个例子。gydF4y2Ba
最后,我们将同样的分割和聚类方法应用于路线图表观基因组学项目中发表的甲基化图谱gydF4y2Ba4gydF4y2Ba.该算法没有对相关的细胞类型进行分组,经常根据供体身份对样本进行聚类。这进一步强调了仔细纯化同质细胞类型的重要性,避免混合细胞群(扩展数据图。gydF4y2Ba5 bgydF4y2Ba).gydF4y2Ba
细胞类型特异性甲基化标记gydF4y2Ba
接下来,我们转向以细胞类型特异性的方式研究不同甲基化的基因组区域。我们将205个样本分为39组特定的细胞类型,包括血细胞类型(B、T、自然杀伤细胞(NK)、粒细胞、单核细胞和组织巨噬细胞)、乳腺上皮细胞(基底和腔内)、肺上皮细胞(肺泡和支气管)、胰腺内分泌细胞(α、β和δ)和外分泌细胞(腺泡和导管)、来自各种来源的血管内皮细胞、心肌细胞和心脏成纤维细胞等。我们还定义了12个超群,其中相关细胞类型被分组,包括肌肉细胞、胃肠道上皮细胞、胰腺等(补充表gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
然后,我们专注于由五个或更多cpg组成的差异甲基化块,这些cpg在一组细胞类型中未甲基化,但在所有其他样本中甲基化,反之亦然。有趣的是,在一种细胞类型中,几乎所有区域(97%)都是未甲基化的,而在所有其他细胞类型中都是甲基化的。然后,我们根据靶细胞类型与所有其他样本甲基化的绝对差异对这些差异区域进行了分类(gydF4y2Ba方法gydF4y2Ba而且gydF4y2Ba补充信息gydF4y2Ba).gydF4y2Ba
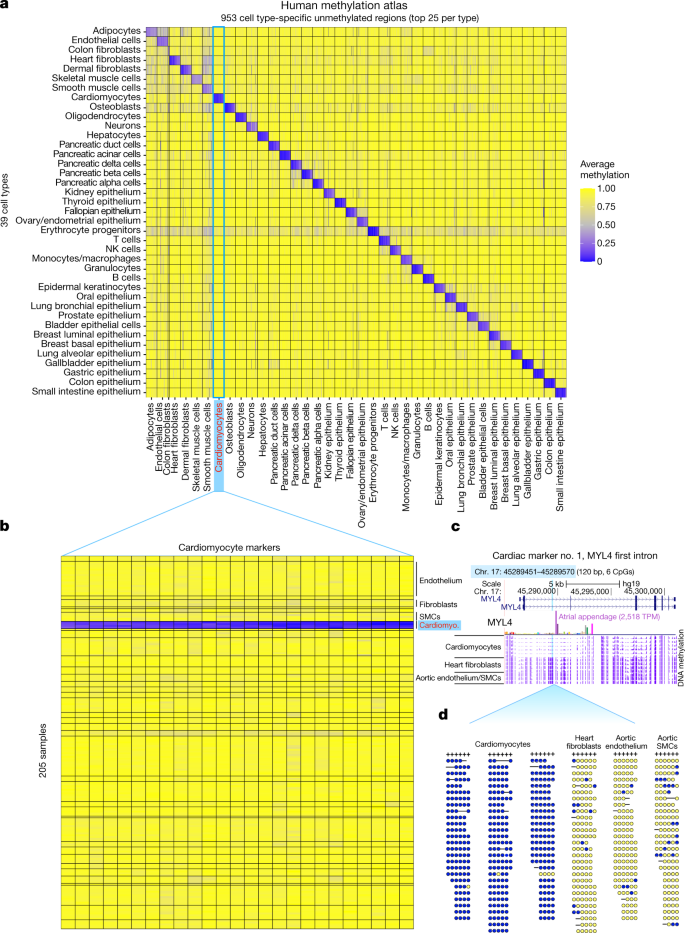
每种细胞类型的前25个差异未甲基化区域包括一个包含1246个标记的人类细胞类型特异性甲基化图谱。gydF4y2Ba3.gydF4y2Ba及补充表gydF4y2Ba4gydF4y2Ba).这些区域在特定细胞类型中是唯一未甲基化的(平均甲基化13%),在所有其他样本中是甲基化的(平均甲基化91%),并且可以作为敏感的生物标记物,用于量化混合物中特定细胞类型DNA的存在。这些标记包括953个细胞类型特异性的未甲基化位点,以及另外293个在少数相关细胞类型中未甲基化的位点。片段水平的分析进一步表明,这些区域的绝大多数DNA片段在目标细胞类型中是未甲基化的,而在所有其他细胞类型中几乎没有(扩展数据图)。gydF4y2Ba6gydF4y2Ba).该图谱有多种应用,包括分析循环的无细胞DNA片段gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba.重要的是,只有约1%的细胞类型特异性标记被还原亚硫酸氢盐测序(RRBS)覆盖,4-8%的甲基测序混合捕获板覆盖,14-24%的细胞类型特异性标记被单cpg 450K/EPIC阵列表示gydF4y2Ba10gydF4y2Ba,强调了全基因组测序对彻底鉴定生物标志物的好处。gydF4y2Ba
一个gydF4y2Ba共953个基因组区域未甲基化的细胞类型特异性方式。图中的每个细胞标记了39种细胞类型(行)中每一种基因组区域(柱)的平均甲基化。每个细胞类型最多显示25个区域,每个区域的平均长度为356 bp (9 cpg)。gydF4y2BabgydF4y2Ba,前25个心肌细胞区。对于每个区域,我们绘制了图谱中所有205个样本中每个CpG位点(列)的平均甲基化,与之前一样分为39种细胞类型。gydF4y2BacgydF4y2Ba心肌细胞中特异性未甲基化的位点。该标记(淡蓝色突出显示)长120 bp(6个CpGs),位于心脏特异性基因MYL4的第一个内含子(心耳中每百万转录本(TPM)表达2518,GTEx插入)。基因组快照描述了6个心肌细胞样本、4个心脏成纤维细胞样本和3个主动脉样本(2个内皮细胞和1个SMC)的平均甲基化(紫色轨道)。gydF4y2BadgydF4y2Ba,从三个心肌细胞样本、一个心脏成纤维细胞样本和两个主动脉样本(内皮和SMC)中可见亚硫酸氢盐转化的碎片。所示为映射到chr的读。17: 45289451-45289570 (hg19),至少有三个覆盖的cpg。黄色和蓝点分别表示甲基化和非甲基化的CpG位点。gydF4y2Ba
人类细胞类型特异性调控图gydF4y2Ba
接下来,我们转向描述这些细胞类型特异性差异非甲基化区域。为此,我们确定了每种细胞类型的前250个未甲基化标记(补充表)gydF4y2Ba4 bgydF4y2Ba),并使用GREAT来识别与每组标记相邻的基因,并测试它们对各种基因集注释的富集程度gydF4y2Ba31gydF4y2Ba.特定细胞类型中唯一未甲基化的位点附近的基因通常反映了该细胞类型的功能特征。例如,B细胞形态、分化、IgM水平和淋巴细胞生成富集了B细胞标记物附近的基因;NK细胞标志物与NK细胞介导的细胞毒性、造血系统、细胞毒性和淋巴细胞生理有关;卵膜、卵黄周隙等输卵管标记物富集;心脏舒张、收缩压、肌肉发育和肥厚的心肌细胞标志物(补充表)gydF4y2Ba5gydF4y2Ba).gydF4y2Ba
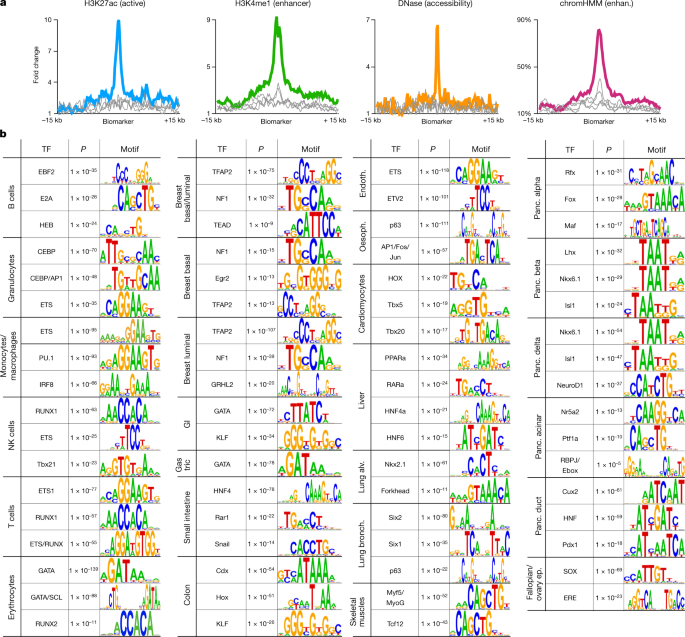
然后,我们分析了细胞类型特异性标记物的DNA可及性和染色质包装,这些标记物由使用测序(ATAC-seq)和DNaseI超敏位点测序(DNase - seq)的转座酶可及性染色质测定所定义。gydF4y2Ba4gydF4y2Ba,gydF4y2Ba32gydF4y2Ba组蛋白标记表明有活跃的启动子和增强子gydF4y2Ba4gydF4y2Ba.单核细胞和巨噬细胞的前250个未甲基化标记是高度可及的,在单核细胞中以H3K27ac和H3K4me1为特征,而其他细胞类型的标记在单核细胞中没有富集(图2)。gydF4y2Ba4gydF4y2Ba),对其他细胞类型的标记也有类似的结果(扩展数据图。gydF4y2Ba7gydF4y2Ba).我们还显示了在细胞类型特异性标记上的chromHMM增强子注释的强协调富集gydF4y2Ba33gydF4y2Ba(无花果。gydF4y2Ba4gydF4y2Ba).这些发现与先前有关组织特异性去甲基化与基因增强子的研究一致gydF4y2Ba1gydF4y2Ba,gydF4y2Ba34gydF4y2Ba.gydF4y2Ba
一个gydF4y2Ba,活性调控标记H3K27ac的平均ChIP-seq信号,增强子标记H3K4me1, DNA可达性和chromHMM增强子注释的前250个细胞类型特异性的单核/巨噬细胞非甲基化标记。其他血细胞类型(粒细胞和B、T和NK细胞)的前250个标记物的平均信号显示为灰色线,用于比较。gydF4y2BabgydF4y2Ba细胞类型特异性标记富集调控基序。所示为使用HOMER motif分析的顶级TF结合位点基序,在每种细胞类型的前1000个差异未甲基化区域中富集。主题类似于以前(更重要的)不包括命中。显示的是HOMER二项gydF4y2BaPgydF4y2Ba值。Alv。,一个lveolar; Bronch., bronchial; Endoth., endothelium; Ep., epithelium; Oesoph., oesophagus; Panc., pancreas.
为了进一步评估细胞类型特异性的未甲基化区域的生物学重要性,我们研究了它们与转录因子(tf)的关系,这些转录因子既可以影响DNA甲基化,也可以以细胞类型特异性的方式结合DNA,这取决于甲基化和染色质gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba.我们确定了每种细胞类型的前1000个未甲基化标记(补充表gydF4y2Ba4摄氏度gydF4y2Ba)并利用HOMER进行motif分析gydF4y2Ba39gydF4y2Ba计算已知TF结合基序的富集(补充表gydF4y2Ba6gydF4y2Ba).对于大多数细胞类型,顶部基序包括主调控因子和关键tf(图2)。gydF4y2Ba4 bgydF4y2Ba).例如,B细胞富集Ebf2/HEB/E2A,粒细胞富集CEBP/AP1/ETS, T细胞富集ETS/RUNX。这种细胞类型特异性的非甲基化区域和TF结合基元之间的联系可以识别新的基因调节回路,并暴露在特定细胞类型中活跃的远端增强子。gydF4y2Ba
我们的目的是鉴定由细胞类型特异性去甲基化标记的假定增强子的靶基因。顶级标记通常落在内含子区域内,并可能调节这些基因(例如,胰α细胞中的胰高血糖素,心肌细胞中的NPPA和MYL4和少突胶质细胞中的MBP;补充表gydF4y2Ba7gydF4y2Ba),或靠近可能的靶标(例如,距离胰岛素基因5kb的β细胞标记)。其他标记与它们的目标基因距离更远。我们设计了一种计算算法来识别在匹配条件下显示增加基因表达水平的细胞类型特异性标记附近的基因(gydF4y2Ba方法gydF4y2Ba).这突出了许多细胞类型的标志基因,并为每种细胞类型的前25个未甲基化标记中的许多提出了假定的靶点。例如,肝细胞标志物与APOE、APOC1、APOC2和胰高血糖素受体相关。同样,心肌细胞标记物与NPPA、NPPB和肌球蛋白基因相关;胰岛标志物与胰岛素和胰高血糖素基因(补充表gydF4y2Ba7gydF4y2Ba).这些发现进一步支持了一个原则,即特定细胞类型中未甲基化的位点可能是积极调节该细胞类型中表达的基因的增强子,通常控制相邻基因。然而,我们注意到,在特定的细胞类型中,与特定的未甲基化位点相邻的基因通常在该细胞类型之外广泛表达(gydF4y2Ba讨论gydF4y2Ba).gydF4y2Ba
为了生成每种细胞类型中假定的调控区域的目录,我们对每种细胞类型的所有样本应用了片段级分析,独立于其他细胞类型。我们扫描了整个基因组,并确定了至少85%具有至少四个cpg的DNA片段未甲基化的基因组区域(gydF4y2Ba方法gydF4y2Ba).在分析的39个细胞类型组中,每一组都确定了一组未甲基化的基因组区域,平均包括36111个区域(补充数据集)gydF4y2Ba1gydF4y2Ba).然后对这些区域进行基因组特征注释,显示平均56%重叠CpG岛,46%靠近启动子区域,44%重叠CTCF结合位点,从而突出了未甲基化位点的调控和结构作用。当有条件时,我们用ENCODE的染色质免疫沉淀测序(ChIP-seq)峰交叉这些区域gydF4y2Ba5gydF4y2Ba以及表观基因组学路线图gydF4y2Ba4gydF4y2Ba在匹配条件下,包括H3K4me3, H3K27ac, H3K4me1, H3K27me3, CTCF和ATAC-seq,并生成了假定的增强子区域的细胞类型特定目录,其中包括与H3K27ac重叠而不与H3K4me3重叠的非甲基化区域(补充数据集gydF4y2Ba2gydF4y2Ba).对这些区域的Motif分析识别出每种细胞类型中的关键tf,与图中所示相似。gydF4y2Ba4gydF4y2Ba(补充表gydF4y2Ba6 b, cgydF4y2Ba).gydF4y2Ba
细胞类型特异性高甲基化位点gydF4y2Ba
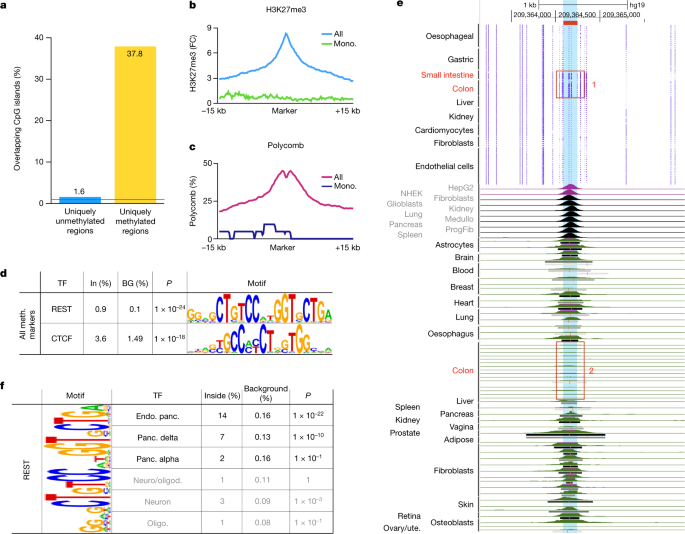
我们研究了那些在一种细胞类型中甲基化,但在人体其他地方未甲基化的基因组区域。它们富集于CpG岛(38%的甲基化区域与1.7-2.7%的细胞类型特异性未甲基化区域相比),并且在其他细胞类型中由H3K27me3和Polycomb标记(图2)。gydF4y2Ba5 a - cgydF4y2Ba),正如先前报道的癌症和发育过程gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba.这些细胞类型特异性的高甲基化区域通常对motif富集不太显著(与独特的未甲基化区域相比)。有趣的是,只有大约3%的细胞类型特异性差异甲基化区域是高甲基化的。gydF4y2Ba
一个gydF4y2Ba38%的顶级细胞类型特异性高甲基化标记(3613个中的1363个,二项gydF4y2BaPgydF4y2Ba< 1 × 10gydF4y2Ba-100年gydF4y2Ba)覆盖CpG岛屿。相比之下,1.6%的细胞类型特异性低甲基化区域(11714中的189个)重叠CpG岛,占基因组的不到0.9%(黑线)。gydF4y2BabgydF4y2Ba在其他细胞类型中,这些区域通常富集H3K27me3。在所有细胞类型特异性高甲基化区域(蓝色)或单核/巨噬细胞特异性高甲基化区域(单核;绿色)。gydF4y2BacgydF4y2Ba,单核细胞和巨噬细胞(chromHMM)中Polycomb注释的类似图,用于所有或单核细胞/巨噬细胞特异性标记。gydF4y2BadgydF4y2Ba对细胞类型特异性高甲基化区域(每种细胞类型前100位)的Motif分析鉴定了已知的CTCF和REST/NSRF Motif。荷马二项gydF4y2BaPgydF4y2Ba值显示出来。gydF4y2BaegydF4y2Ba,其中一个站点的ChIP-seq数据分析(chr。1: 209364093-209364250,蓝色高亮,hg19),在小肠和结肠上皮特异性甲基化(红框1),其他部位未甲基化。如下图所示,该位点在多种细胞类型和组织中结合,但在体内胃和结肠上皮中大多未结合(红框2)。gydF4y2BafgydF4y2Ba, REST/NSRF基序存在于内分泌胰腺前100个细胞类型特异性高甲基化区域的14%、顶端δ细胞标记物的7%和顶端α细胞标记物的2%,而背景序列中约为0.1%,与REST靶蛋白在内分泌胰腺中的表达一致。荷马二项gydF4y2BaPgydF4y2Ba值显示出来。Alv。,一个lveolar; bronch., bronchial; Endo. panc., endocrine pancreas; Ep., epithelium; Oesoph., oesophagus; Oligo, oligodendrocytes; Panc., pancreas; Ute., uterus.
在汇集了所有细胞类型特异性的高甲基化区域后,我们发现了染色质调节因子CTCF的目标序列的强富集(gydF4y2BaPgydF4y2Ba≤1 × 10gydF4y2Ba-18年gydF4y2Ba;无花果。gydF4y2Ba5 dgydF4y2Ba).这表明CTCF结合位点的DNA甲基化可以作为一个组织特异性的调节开关来调节其结合,潜在地影响组织特异性的三维基因组组织gydF4y2Ba35gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba.为了验证这一想法,我们将CTCF位点的DNA甲基化模式与特定组织中的全基因组CTCF蛋白结合进行了比较。数字gydF4y2Ba5 egydF4y2Ba显示了甲基化模式,并在体内公布了CTCF占据的一个位点,该位点在结肠和肠道中被特异性甲基化。与DNA甲基化阻止CTCF结合一致,ChIP数据显示结肠中该位点CTCF结合的选择性缺失。此外,神经基因的转录抑制因子re1沉默TF/神经元限制性沉默因子(REST/NRSF)的靶点在特定细胞类型中甲基化的位点富集(gydF4y2BaPgydF4y2Ba≤1 × 10gydF4y2Ba-24年gydF4y2Ba),这在胰岛细胞的甲基组中最为显著(图。gydF4y2Ba5 fgydF4y2Ba).尽管DNA甲基化尚未被证明会影响REST的结合或活性,但这一发现提出了一种有趣的可能性,即胰岛中REST靶点的甲基化可以独立于REST抑制而允许内分泌分化。gydF4y2Ba
片段级甲基组反褶积gydF4y2Ba
最后,我们开发了一种用于DNA甲基化测序数据的计算片段级反褶褶算法,并使用为每种细胞类型定义的前25个标记(共1246个标记)来研究从复合组织样本和cfDNA中获得的甲基组。简单地说,我们生成了一个图谱,其中为每个细胞类型(列)中的每个标记(行)计算未甲基化片段的百分比。然后使用非负最小二乘(NNLS)算法拟合输入样本并估计其相对贡献(gydF4y2Ba补充信息gydF4y2Ba).gydF4y2Ba
为了估计我们的片段级方法的准确性,我们使用了硅混合序列读。对于每一种细胞类型,我们应用一种留一的方法在白细胞reads中混合一个保留样本,然后使用反褶积算法来推断混合物中的细胞组成。我们以0到10%的浓度重复这个过程。如图所示。gydF4y2Ba6gydF4y2Ba,我们发现,1246个标记(每种细胞类型的前25个)允许以大约0.1%的分辨率准确检测来自给定来源的DNA,与基于阵列的方法相比,提高了近一个数量级gydF4y2Ba28gydF4y2Ba.四向硅混合,其中内皮细胞和肝细胞甲基组也包括在内,以逼真地模拟cfDNA组成,产生了类似的结果(扩展数据图。gydF4y2Ba8gydF4y2Ba).gydF4y2Ba
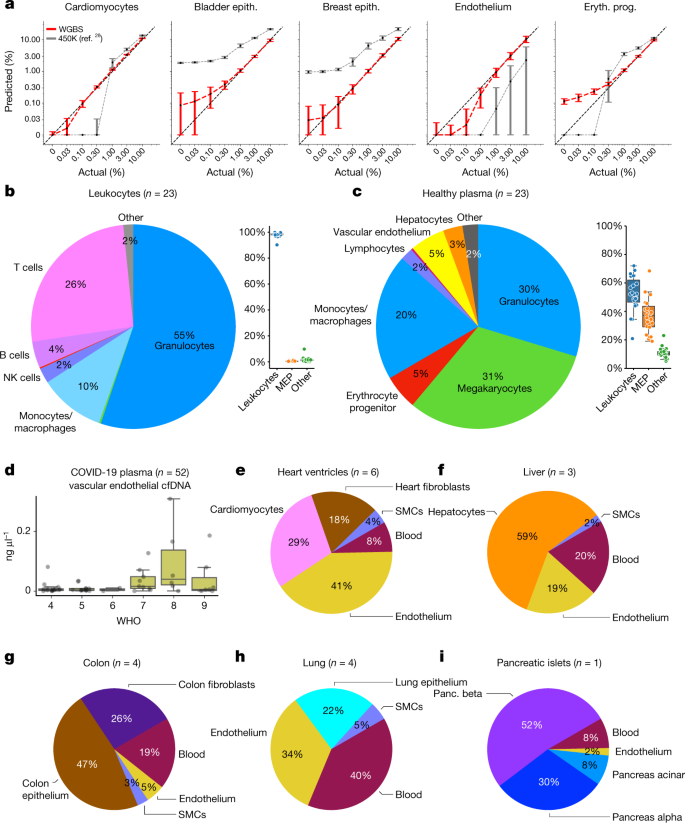
一个gydF4y2Ba,细胞类型特异性标记的分辨率低于0.1%。在五种细胞类型的硅模拟中,将保留的样本计算混合在白细胞内,然后使用1246个图谱标记加上25个额外的巨核细胞标记(红色)或对这些混合进行基于阵列的反褶积分析gydF4y2Ba28gydF4y2Ba(灰色)。箱形图显示了10次模拟的平均贡献,误差条表示1 s.d。gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba,白细胞的细胞类型组成(gydF4y2BabgydF4y2Ba)及血浆样本(gydF4y2BacgydF4y2Ba)来自健康的捐赠者。箱形图显示了白细胞、巨核细胞和红细胞(MEP)和其他细胞类型的总体比例。gydF4y2BadgydF4y2Ba52例SARS-CoV-2患者低覆盖率血浆样本分析(参考文献;gydF4y2Ba44gydF4y2Ba)在WHO序数量表7级或更高(需要进入重症监护室)的患者中发现了内皮来源的cfDNA。gydF4y2BaegydF4y2Ba- - - - - -gydF4y2Ba我gydF4y2Ba, Roadmap/ENCODE样本的片段级反褶积gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba显示特定于单元格类型的贡献。gydF4y2BaegydF4y2Ba心脏心室样本包含心肌细胞、内皮细胞、成纤维细胞和血液的混合物。gydF4y2BafgydF4y2Ba肝脏样本含有约60%的肝细胞DNA,外加血液和内皮细胞。gydF4y2BaggydF4y2Ba结肠样本含有大约50%的上皮细胞,以及成纤维细胞和血液。gydF4y2BahgydF4y2Ba肺标本中肺上皮细胞含量低于30%。gydF4y2Ba我gydF4y2Ba胰岛样本包含β、α、导管和腺泡细胞。箱形图为中位数和IQR,须为1.5× IQR。gydF4y2Ba
然后,我们利用23名健康供体的WGBS数据估计白细胞和cfDNA的细胞组成;99.5%的白细胞来源DNA来自于粒细胞、单核细胞、巨噬细胞和NK细胞、T细胞和B细胞,与典型的血液计数一致(图2)。gydF4y2Ba6 bgydF4y2Ba及补充表gydF4y2Ba8gydF4y2Ba).健康人的cfDNA主要来源于白细胞:粒细胞(29.7%)、单核/巨噬细胞(20%)和淋巴细胞(3%)。对cfDNA有贡献的实体组织包括血管内皮细胞(6%)和肝细胞(3.1%)。gydF4y2Ba6摄氏度gydF4y2Ba),与先前的结果一致gydF4y2Ba28gydF4y2Ba.目前的图谱还显示了巨核细胞(31%)和红细胞祖细胞(5%)对cfDNA的显著贡献,这在以前使用范围更有限的参考甲基组的研究中没有观察到。gydF4y2Ba
COVID-19患者内皮细胞cfDNA的研究gydF4y2Ba
基于DNA甲基化模式的分析为确定cfDNA的组织起源提供了机会。COVID-19会对多个组织造成损伤,其中一些组织没有生物标志物。我们使用该图谱对52例因COVID-19住院患者的浅WGBS数据进行反卷积(参考文献)。gydF4y2Ba44gydF4y2Ba).我们从粒细胞、红细胞祖细胞、肺和肝脏中鉴定出过量的无细胞DNA片段,与已发表的这些样本分析一致(gydF4y2Ba补充信息gydF4y2Ba).引人注目的是,我们还发现了血管内皮细胞对这些患者cfDNA的显著贡献,在没有内皮细胞甲基化组参考的情况下,这在已发表的分析中无法检测到。gydF4y2Ba6 dgydF4y2Ba).有趣的是,重症患者(WHO评分≥7)内皮细胞来源的cfDNA浓度高于轻症患者(WHO评分≤6;gydF4y2BaPgydF4y2Ba≤6 × 10gydF4y2Ba5gydF4y2BaMann-Whitney)。这些结果表明,血管内皮细胞死亡在COVID-19的发病机制中起着重要作用,可能与凝血疾病有关,并强调了使用全面的细胞类型特异性图谱进行cfDNA甲基化组分析的好处。gydF4y2Ba
复合组织的细胞类型反褶积gydF4y2Ba
最后,我们分析了ENCODE的全基因组甲基组gydF4y2Ba5gydF4y2Ba以及表观基因组图谱路线图gydF4y2Ba4gydF4y2Ba使用我们的图谱(基于每种细胞类型的25个标记)。一些甲基组的反褶积显示了预期的同质组成,例如,路线图T细胞样本中97-99%的T细胞DNA(补充表)gydF4y2Ba9gydF4y2Ba).然而,其他样本的分析显示出高度异质性的组成,如先前基于阵列的散装组织反褶积算法(如EpiDISH和EpiScore)报道的那样gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba45gydF4y2Ba.例如,心脏心室样本包含29%的心肌细胞,41%的内皮细胞和18%的心脏成纤维细胞(图2)。gydF4y2Ba6 egydF4y2Ba);肝脏甲基化组由约60%的肝细胞、21%的血液和20%的内皮细胞组成;结肠甲基组由大约50%的结肠上皮细胞,26%的结肠成纤维细胞和19%的血液组成。最引人注目的是,路线图肺样本主要由血液(40%)、内皮(34%)和平滑肌(5%)组成,只有22%的DNA来自肺上皮细胞(图2)。gydF4y2Ba外:我6gydF4y2Ba及补充表gydF4y2Ba9gydF4y2Ba).重要的是,这里提出的205个样本的类似反褶积对每个样本的预期细胞类型的平均贡献为94%(中位数为95%,补充表gydF4y2Ba10gydF4y2Ba),或91%(中位数92%),在更严格的漏掉交叉验证分析中(补充表gydF4y2Ba11gydF4y2Ba),以显示所收集样本的纯度。gydF4y2Ba
当然,片段级分析仅限于可获得全基因组测序数据的细胞类型,一些细胞类型只能通过基于阵列的算法进行分析gydF4y2Ba15gydF4y2Ba,gydF4y2Ba28gydF4y2Ba.尽管如此,这里提出的标记和算法允许分析复合散装组织和血浆样本,跨多种细胞类型,并具有较高的准确性。gydF4y2Ba
讨论gydF4y2Ba
这里描述的人类细胞类型甲基化的综合图谱阐明了DNA甲基化的原理,并为多条研究线以及翻译应用提供了有价值的资源。gydF4y2Ba
我们的分析使用全基因组测序数据显示,来自不同个体的相同细胞类型的健康复制之间的甲基化模式惊人地相似。至少就健康组织而言,个体之间的相似性反映了细胞分化和维持回路的稳健性。涉及表观基因组不稳定的病理明显破坏了这些回路,导致来自特定正常细胞类型的细胞之间的甲基化模式更加多样化。我们预测,即使在癌症(具有相同的主要解剖部位和组织学类型)中,在甲基化块水平上对纯化的上皮细胞进行比较甲基组分析,也将显示出比通常假设的更小的个体间差异。gydF4y2Ba
正如图谱所示,每种细胞类型都有一组基因组区域,与其他细胞类型相比,该细胞类型中唯一的未甲基化区域,以及与相关细胞类型共享甲基化模式的其他基因组区域。使用细胞类型特异性甲基组的无监督聚类,我们发现细胞类型的聚类方式反映了它们的发育起源,而不是表达模式。这提供了一个迷人的观点,DNA甲基化作为祖细胞甲基组的记录,通过戏剧性的发育转变和此后几十年的生命保留在基因组中。我们提出,比较甲基组分析将允许重建胎儿结构或细胞类型的部分甲基组,类似于在进化生物学中重建最后的共同祖先。gydF4y2Ba
绝大多数细胞类型特异性差异甲基化区域在一种细胞类型中特异性去甲基化。这些区域的染色质通常是高度可及的,并具有与活性基因调控相关的组蛋白标记,如在增强子和启动子中发现的那样。此外,这些位点在该细胞类型中操作的TF结合位点基序丰富。我们设计了一种基于距离和基因表达谱的综合方法,使我们能够突出这些假定增强子区域的潜在靶基因。许多增强子区域与附近广泛表达的基因相关,可能反映了多个组织特异性增强子的基因调控。我们的发现与先前的研究一致,表明组织特异性低甲基化发生在基因增强子上gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba.我们的标记识别数据驱动方法是对最近的基因中心方法的补充gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba使用组织特异性单细胞RNA测序数据来定义标记基因,并识别目标细胞类型中特异性未甲基化的邻近CpGs。最后,我们设计了一个片段级基因组分析,以识别每种细胞类型的数万个未甲基化区域,这些区域用基因组特征、DNA可及性、染色质标记和TF结合基序进行注释,以产生一个假定增强子的细胞类型特异性目录。对该图谱的进一步分析将显示并验证每种细胞类型中完整的人类增强子集。gydF4y2Ba
相反,我们在一种或两种细胞类型中确定了特异性甲基化的基因组区域,约占细胞类型特异性差异甲基化区域的3%。它们通常位于CpG岛,特征是H3K27me3和Polycomb结合在位点未甲基化的组织中gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba.这种表观遗传抑制转换以前在癌症和早期发展中被描述过gydF4y2Ba41gydF4y2Ba,gydF4y2Ba46gydF4y2Ba但其在特定细胞类型分化过程中的作用尚不清楚。这些区域富含CTCF结合位点,这表明DNA甲基化在减弱CTCF结合中的作用,从而调节相邻DNA的细胞类型特异性的三维组织gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba47gydF4y2Ba.gydF4y2Ba
对于DNA甲基化测序数据,据我们所知,这里描述的图集是迄今为止最全面的概要。我们确定了超过1000个细胞类型独特的DNA甲基化区域,这些区域可以作为片段级分析和通过监测cfDNA识别细胞死亡事件的准确和特异的生物标志物。值得注意的是,这些标记区域中的大多数没有被450K/EPIC BeadChip DNA甲基化阵列覆盖,并且以前没有被认识到。为了允许阵列数据的解释,我们提供了一组细胞类型特定的标记,这些标记仅限于BeadChip 450K阵列中包含的CpG位点。类似地,我们在RRBS和混合捕获面板靶向的区域识别了细胞类型特异性标记(扩展数据图)。gydF4y2Ba9gydF4y2Ba及补充表格gydF4y2Ba12gydF4y2Ba- - - - - -gydF4y2Ba17gydF4y2Ba).如图扩展数据图所示。gydF4y2Ba10gydF4y2Ba,这种阵列适配的图谱可以对胰岛、肺和乳腺活检的阵列甲基组进行高分辨率的解释,突出了以前没有描述过的细胞类型的存在gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.gydF4y2Ba
许多细胞类型从图谱中缺失,通常是因为材料的可用性有限。例如成骨细胞、胆管细胞、肾上腺细胞、尿道上皮细胞和造血干细胞。此外,我们没有分离许多感兴趣的亚群——例如,不同类型的神经元或淋巴细胞。地图集被视为一个活的、公开可用的数据库,将来可以更新。图谱的分辨率产生了对复合组织的定量理解,并允许人们识别尚未被表征的其他细胞类型的缺失甲基组。我们也承认,由于用于FACS的抗体质量的差异以及它们允许分离细胞类型的程度,分选细胞群的纯度有所不同。尽管如此,即使是图谱中纯度最低的细胞类型(例如,一些血管内皮细胞、成纤维细胞、SMC和脂肪细胞的制剂显示纯度为70-80%),在重复平均后,也可用于鉴定差异甲基化区域和推断混合物中的细胞组成。gydF4y2Ba
总之,我们提出了一个全面的初级人类细胞类型甲基化图谱,以及一套广泛的细胞类型特异性标记和计算工具,用于混合细胞类型样本的片段级分析。这些补充了大量的阵列甲基组和反褶积工具,可用于阵列数据的分析。总之,这些数据阐明了DNA甲基化在细胞生物学和基因调控中的作用,并促进了每种细胞类型中活性增强子的鉴定。也许我们的图谱最有前途的用途是混合细胞类型样本的片段级反褶积的潜力,允许敏感地识别患有癌症和其他疾病的个体血浆中cfDNA的起源组织gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba.gydF4y2Ba
方法gydF4y2Ba
人体组织样本gydF4y2Ba
人体组织来源多种多样,详见补充表gydF4y2Ba1gydF4y2Ba.分析的205个样本中,大多数(148个)是从哈大沙医疗中心常规临床外科手术时获得的组织残余物中分选出来的。在所有病例中,都使用了远离任何已知病理的正常组织。在切除组织前咨询外科医生和/或病理学家,以确认其切除不会以任何方式影响最终的病理诊断。例如,在盲肠癌行右结肠切除术的患者中,取升结肠最远端和回肠末端最近端进行细胞分离。无已知血液学病理的患者在关节置换术时获得正常骨髓。患者人群包括135人(gydF4y2BangydF4y2Ba= 60名男性,gydF4y2BangydF4y2Ba= 74名女性)年龄3-83岁。大多数捐赠者是白人。以色列耶路撒冷哈大沙医疗中心机构审查委员会(赫尔辛基委员会)批准收集正常组织残余物。术前从每个捐赠者或法定监护人处获得书面知情同意。gydF4y2Ba
详见补充表gydF4y2Ba1gydF4y2Ba,通过合作安排获得一些细胞和组织:胰腺外分泌和肝脏样本(尸体器官捐赠者,gydF4y2BangydF4y2Ba= 5)来自俄勒冈健康与科学大学M. Grompe;脂肪细胞(减肥后整容手术时的皮下脂肪细胞,gydF4y2BangydF4y2Ba= 3),少突胶质细胞和神经元(脑解剖,gydF4y2BangydF4y2Ba= 14)来自K. L.斯伯丁和H.德鲁伊,斯德哥尔摩卡罗林斯卡学院;阿尔伯塔大学J. Shapiro的研究级尸体胰岛(gydF4y2BangydF4y2Ba= 16)。在所有病例中,组织的获取和转移都符合当地法律,并经过了当地人体实验伦理委员会的批准。从商业来源获得16种细胞类型,其中15种来自Lonza, 1种来自Sigma-Aldrich。三种胰岛制剂从综合胰岛分配计划(gydF4y2Bahttps://iidp.coh.orggydF4y2Ba).gydF4y2Ba
纯化细胞群的组织分离和FACS分选gydF4y2Ba
手术时获得的新鲜组织被修剪以去除多余组织。使用针对每种组织类型优化的基于酶的方案分散细胞。将得到的单细胞悬浮液与相关抗体孵育,并对FACS进行分类,以获得所需的细胞类型(扩展数据图。gydF4y2Ba2gydF4y2Ba而且gydF4y2Ba补充信息gydF4y2Ba).gydF4y2Ba
活分选细胞的纯度是通过对关键已知细胞类型特异性基因的信使RNA分析来确定的,而分选前固定的细胞纯度是通过先前验证的细胞类型特异性甲基化信号来确定的(扩展数据图。gydF4y2Ba2摄氏度gydF4y2Ba而且gydF4y2Ba补充信息gydF4y2Ba).DNA提取使用DNeasy血液和组织试剂盒(no。69504, Qiagen),并保存在- 20°C用于亚硫酸氢盐转换和全基因组测序。gydF4y2Ba
WGBSgydF4y2Ba
使用EZ-96 DNA甲基化试剂盒(Zymo Research)对高达75 ng的剪切基因组DNA进行亚硫酸氢盐转化,并在MicroLab STAR (Hamilton)上进行液体处理。双索引测序文库使用Accel-NGS甲基- seq DNA文库制备试剂盒(Swift BioSciences)制备,并在Hamilton MicroLab STAR上执行定制的液体处理脚本。使用KAPA文库量化试剂盒(KAPA Biosystems)对Illumina平台的文库进行量化。4个独特的双索引文库,以及10% PhiX v.3文库(Illumina),汇集并聚集在Illumina NovaSeq 6000 S2流式细胞上,然后进行150 bp的配对端测序。在1×和10×时,每个样品的总读计数和平均测序深度(以读对为单位)以及cpg百分比见补充表gydF4y2Ba1gydF4y2Ba.还列出了平均甲基化水平,每个样品,在CpG,非CpG和CC二核苷酸。有趣的是,与其他样本(约1%)相比,排序神经元样本显示出更高的CpA甲基化(约10%)。gydF4y2Ba
WGBS计算处理gydF4y2Ba
使用bwa-meth (v.0.2.0)将配对的FASTQ文件映射到人类(hg19, hg38), lambda, pUC19和病毒基因组gydF4y2Ba51gydF4y2Ba然后使用SAMtools转换为BAM文件(v.1.9)gydF4y2Ba52gydF4y2Ba.Sambamba (v.0.6.5)使用参数' -l 1 -t 16——sort-buffer-size 16000——overflow-list-size 10000000 '标记重复读取。gydF4y2Ba53gydF4y2Ba).使用SAMtools视图(参数为' -F 1796 -q 10 ')排除映射质量低、重复或未映射到正确的对中的读。从非cpg核苷酸中剥离读取,使用wgbstools转换为PAT文件(v.0.1.0)gydF4y2Ba54gydF4y2Ba.gydF4y2Ba
基因组分割成多样本同质块gydF4y2Ba
我们开发并实现了一种多通道动态p规划分割算法,将基因组划分为连续的基因组区域(块),在每个样本的多个cpg中显示均匀的甲基化水平gydF4y2Ba54gydF4y2Ba.使用生成概率模型,每个块诱导伯努利分布与一些gydF4y2Ba\({\θ}_{我}^ {k} \)gydF4y2Ba,在那里gydF4y2Ba我gydF4y2Ba是块索引和gydF4y2BakgydF4y2Ba样本索引(gydF4y2BakgydF4y2Ba= 1,…,gydF4y2BaKgydF4y2Ba),每个观测值(在一个测序片段上出现一个CpG)由一个从相同beta值Ber采样的随机变量i.i.d(独立且同分布)表示gydF4y2Ba\({\θ}_{我}^ {k} \)gydF4y2Ba.所有测序数据的对数似然是所有块的对数似然之和,每个块分解为所有样本的对数似然之和。的对数可能性gydF4y2Ba我gydF4y2Ba因此,Th块可以形式化为:gydF4y2Ba
在哪里gydF4y2Ba\ ({{N} _ {C})} _{我}^ {k} \,,, \ {({N} _ {T})} _{我}^ {k} \)gydF4y2Ba中甲基化和非甲基化观察的数量分别是多少gydF4y2Ba我gydF4y2Ba第Th块gydF4y2BakgydF4y2BaTh样本,而gydF4y2Ba\({{\帽子{\θ}}_{我}}^ {k} \)gydF4y2Ba标记伯努利分布参数的贝叶斯估计量,用gydF4y2BaC \({} _{},{一}_ {T} \)gydF4y2Ba每个块/样本的伪计数:gydF4y2Ba
这些超参数用于正则化,以控制过拟合(较短的块)和泛化(较长的块)之间的权衡。然后使用动态规划来寻找整个基因组的最佳分割。简单地说,我们保持1 ×gydF4y2BaNgydF4y2Ba表gydF4y2BaTgydF4y2Ba(gydF4y2BaNgydF4y2Ba= 28,217,448 CpGs)为所有前缀的最佳分割分数。具体地说,gydF4y2BaTgydF4y2Ba[gydF4y2Ba我gydF4y2Ba]表示从1到i的所有CpG站点的最优分割得分gydF4y2BaTgydF4y2Ba[gydF4y2BaNgydF4y2Ba]在整个基因组中拥有最终的、最优的分数。表本身从1到依次更新gydF4y2BaNgydF4y2Ba,其中最优分割到位置gydF4y2Ba我gydF4y2Ba通过在较短的最佳分段中添加一个新的块来实现(例如,到位置gydF4y2Ba我gydF4y2Ba”):gydF4y2Ba
为此,考虑所有先前的最优分割,并从位置(gydF4y2Ba我gydF4y2Baʹ+ 1)定位gydF4y2Ba我gydF4y2Ba(最大区块大小为5,000 bp)。选择log-likelihood最大的组合作为从1到的最优分割gydF4y2Ba我gydF4y2Ba,最后一个块的起始索引记录在回溯表中。计算最优分割的分值gydF4y2BaTgydF4y2Ba[gydF4y2BaNgydF4y2Ba], traceback表用于检索完整的分段。设置块长度的上限(5000个碱基)以提高运行时间,并且每个染色体分别运行。该模型忽略了连续cpg之间的线性距离。文中进一步描述了该模型和分割算法gydF4y2Ba补充信息gydF4y2Ba.gydF4y2Ba
分割和聚类分析gydF4y2Ba
我们使用wgbstools(参数为segment—max_bp 5000),以所有205个样本为参考,将基因组分割为7,104,162个区块,保留了2,099,681个区块,覆盖至少4个cpg。对于分层聚类(图。gydF4y2Ba2gydF4y2Ba),我们选择了所有样本中平均甲基化变异性最高的前1%(20,997)块。在三分之二的样本中,有足够覆盖至少10个观测值(按排序的CpG位点计算)的块被进一步保留。然后,我们计算每个块的平均甲基化,使用wgbstools(——beta_to_table -c 10)计算样本,标记少于10个观察值的块为缺失值,并使用sklearn KNNImputer (v.0.24.2)估算其甲基化值。gydF4y2Ba55gydF4y2Ba.采用无监督聚类算法对205个样本进行聚类gydF4y2Ba23gydF4y2Ba使用scipy (v.1.6.3)gydF4y2Ba56gydF4y2Ba和L1范数。扇形图使用ggtree (v.2.2.4)绘制gydF4y2Ba57gydF4y2Ba.gydF4y2Ba
Cell-type-specific标记gydF4y2Ba
205份图谱样本按细胞类型分为51组,产生39个基本群和12个复合超群(补充表)gydF4y2Ba3.gydF4y2Ba).然后,我们进行了一个单一与所有的比较,以确定每种细胞类型唯一的差异甲基化块。为此,我们使用wgbstools的“find_markers”函数首先识别至少覆盖5个cpg(长度10-1,500 bp)的区块,计算每个区块/样本的平均甲基化,并根据目标样本与所有其他样本之间平均甲基化的差异对区块进行排序。为了具有一定的灵活性,这一差异(对于未甲基化标记)被计算为目标样本中第75百分位(通常允许一个异常值)与背景组中第2.5百分位(通常允许大约五个异常值样本)之间的差异。对于甲基化标记,计算为第25百分位和97.5百分位之间的差值(gydF4y2Ba补充信息gydF4y2Ba).低覆盖率区块(少于25个观测值),其中平均甲基化的估计误差约为10%,被默认值0.5所取代,该默认值既非甲基化也非甲基化,从而减少了区块的甲基化差异并降低了其等级。对于细胞类型特异性标记,我们选择每种细胞类型的前25个标记,共1246个标记(补充表)gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
450K/EPIC、RRBS和混合捕获面板的地图集在检查基因组区域子集、重叠各种探针集或基因组区域(-b选项)时被相似地识别出来。对每种细胞类型的前250个标记进行染色质分析(共11713个标记;补充表gydF4y2Ba4 bgydF4y2Ba).对每种细胞类型的前1000个标记进行Motif分析(共50,286个标记;补充表gydF4y2Ba4 bgydF4y2Ba)利用第25百分位和第75百分位之间的差异,允许假定的增强子在其他细胞类型中未甲基化。gydF4y2Ba
基因集注释的富集gydF4y2Ba
利用GREAT进行基因集富集分析gydF4y2Ba31gydF4y2Ba.对于每种细胞类型,我们选择了前250个差异未甲基化区域,并使用默认参数通过批处理web界面运行GREAT。忽略“ensemble Genes”的富集,采用二项错误发现率≤0.05的显著性阈值。gydF4y2Ba
染色质标记富集gydF4y2Ba
对于每一种细胞类型,我们分析了前250个不同的未甲基化区域与发表的ChIP-seq (H3K27ac和H3K4me1)和路线图表观基因组学项目的DNase测序(从ftp.ncbi.nlm.nih.gov/pub/geo/DATA/roadmapepigenomics/by_experiment下载)gydF4y2Bahttp://egg2.wustl.edu/roadmap/data/byDataType/dnase/BED_files_enhgydF4y2Ba)的bigWig和bed格式。其中包括用于B细胞标志物的E032,用于T细胞标志物的E034,用于单核/巨噬细胞标志物的E029,用于肝细胞的E066,用于心肌细胞和成纤维细胞的E104,用于胃/小肠/结肠的E109和E110gydF4y2Ba4gydF4y2Ba.下载了chromHMM的注释(15个州版本)gydF4y2Bahttps://egg2.wustl.edu/roadmap/data/byFileType/chromhmmSegmentations/ChmmModels/coreMarks/jointModel/finalgydF4y2Ba3.gydF4y2Ba,提取标注为增强子(7_Enh)的基因组区域,以bigWig格式重新格式化。原始单细胞ATAC-seq数据从GEO GSE165659下载。gydF4y2Ba32gydF4y2Ba)为70个样本的“特征”和“矩阵”文件。对于每个样本,将相同类型的单元格合并以输出一个床图文件,该文件使用UCSC liftOver从hg38映射到hg19gydF4y2Ba58gydF4y2Ba.使用床工具删除重叠区域(v.2.26.0)gydF4y2Ba59gydF4y2Ba.最后,使用bedGraphToBigWig创建bigWig文件(v.4)gydF4y2Ba60gydF4y2Ba.使用deepTools (v.3.4.1)制备热图和平均图gydF4y2Ba61gydF4y2Ba,功能为“computeMatrix”、“plotHeatmap”及“plotProfile”。除了' referencePoint=center ',对于ChIP-seq, DNaseI和chromHMM数据,我们使用默认参数,15 kb margin和' binSize=200 ',对于ATAC-seq数据,我们使用75 kb margin和' binSize=1000 '。gydF4y2Ba
主题分析gydF4y2Ba
对于每种细胞类型,我们分析了已知基序的前1000个差异未甲基化区域(补充表gydF4y2Ba6gydF4y2Ba)使用HOMER函数' findMotifsGenome.pl ',参数' -bits '和' -size 250 'gydF4y2Ba39gydF4y2Ba.对每种细胞类型的未甲基化区域进行了类似的分析(补充表)gydF4y2Ba6 bgydF4y2Ba),以及与H3K27ac重叠而不与H3K4me3重叠的非甲基化区域(补充表gydF4y2Ba6摄氏度gydF4y2Ba).gydF4y2Ba
甲基化标记-基因关联gydF4y2Ba
对于每个细胞类型特异性标记,我们鉴定了所有相距500 kb的邻近基因。然后,我们在涵盖50种组织和细胞类型的GTEx数据集中检测了这些基因的表达水平gydF4y2Ba62gydF4y2Ba.然后,我们通过将表达值替换为标准差(gydF4y2BazgydF4y2Ba-scores)高于/低于该基因在样本中的平均表达。接下来是按列标准化,其中一个基因在给定条件下的相对富集被其他基因在该条件下的富集标准化。这突出了每个组织中过度表达最多的基因。然后,我们将每个“标记-基因-条件”组合分类为第1层:距离≤5 kb,表达≥10 TPM和gydF4y2BazgydF4y2Ba分数≥1.5;Tier 2:与Tier 1相同,但距离≤50kb;tier 3:最大可达750kb,表达式≥25tpm和gydF4y2BazgydF4y2Ba分数≥5;第4层:和第3层一样,但是gydF4y2BazgydF4y2Ba分数≥3.5。gydF4y2Ba
每种细胞类型的未甲基化位点和假定增强子目录gydF4y2Ba
对于每个基因组区域(至少四个cpg的区块),以及39个细胞类型组中的任何一个,来自所有重复的至少四个cpg的片段被合并并分类为U(片段级甲基化15%或更低),M(至少85%)或X(超过15%但低于85%)。然后使用' wgbstools homog -threshold .15,计算U片段的百分比。85 ',以及至少保留85%未甲基化片段的块。这些区块与基于UCSC hg19注释的基因组特征重叠,包括CpG岛和转录起始位点区域(距离基因起始位点最远1 kb)。我们还使用了从Roadmap下载的窄峰注释gydF4y2Ba4gydF4y2Ba和ENCODE项目gydF4y2Ba5gydF4y2Ba(附表所列之资格gydF4y2Ba6 dgydF4y2Ba).使用liftOver将hg38床文件转换为hg19gydF4y2Ba58gydF4y2Ba.对于假定的增强子,非启动子活性调控区域定义为在匹配条件下重叠的H3K27ac峰,而不是H3K4me3峰。TF绑定位点从JASPAR 2022(参考。gydF4y2Ba63gydF4y2Ba).gydF4y2Ba
细胞甲基化的个体间变异gydF4y2Ba
我们将两个样本之间的相似分数定义为包含至少3个CpG和至少10个二进制观测值(测序CpG位点)的块的比例,其中两个样本的平均甲基化差异至少0.5。只有细胞类型gydF4y2BangydF4y2Ba考虑来自不同供体的≥3个facs分类重复(共136个样本)。gydF4y2Ba
ChIP-seq分析gydF4y2Ba
CTCF ChIP-seq数据从ENCODE项目下载gydF4y2Ba5gydF4y2Ba为168个bigWig文件,涵盖61种组织/细胞类型(hg19)。使用multiBigwigSummary对相同细胞类型的样本进行平均(v.3.4.1)gydF4y2Ba61gydF4y2Ba.gydF4y2Ba
内胚层标记物分析gydF4y2Ba
使用wgbstools函数' find_markers ' (v.0.2.0)找到所有892个内胚层低甲基化标记,参数为'——delta_quants 0.4——tg_quant 0.1——bg_quant 0.1 '(参考。gydF4y2Ba54gydF4y2Ba).对于内胚层来源的上皮细胞,51个样本与来自中胚层或外胚层的103个非上皮细胞样本进行了比较。如果上皮样本的第90百分位的平均甲基化比非上皮样本的第10百分位的平均甲基化至少低0.4,则选择块作为标记。gydF4y2Ba
UXM片段级反褶积算法gydF4y2Ba
我们开发了一种片段级反褶积算法:根据甲基化和非甲基化cpg的数量,每个片段被注释为U(大部分未甲基化)、M(大部分甲基化)或X(混合)gydF4y2Ba64gydF4y2Ba.然后,我们计算了每个基因组区域(标记)和所有细胞类型的U/X/M片段的比例gydF4y2BakgydF4y2Ba论文认定。这里我们用了gydF4y2BakgydF4y2Ba= 4, U读数的甲基化CpGs阈值小于或等于25%,M读数的甲基化CpGs阈值大于或等于75%。然后构建参考地图集gydF4y2Ba一个gydF4y2Ba共有1232个区域(每种细胞类型的前25个标记),其中gydF4y2Ba一个gydF4y2Ba我,我gydF4y2Ba单元持有U的比例gydF4y2Ba我gydF4y2Ba的Th标记gydF4y2BajgydF4y2BaTh细胞类型。给定一个输入样本,计算每个标记处的U比例,形成一个1232 × 1的向量gydF4y2BabgydF4y2Ba.然后,应用NNLS算法对系数向量进行推断gydF4y2BaxgydF4y2Ba通过最小化gydF4y2Ba\({| A\乘以x-b|}_{2}\)gydF4y2Ba以非负性为准gydF4y2BaxgydF4y2Ba,归一化为gydF4y2Ba\({\σ}_ {j} {x} _ {j} = 1 \)gydF4y2Ba.或者,每个标记可以根据输入样本中的片段覆盖率进行不同的加权。为此,gydF4y2BabgydF4y2Ba可以定义为每个区域的U片段数,gydF4y2Ba一个gydF4y2Ba类似地乘以gydF4y2BaCigydF4y2Ba,则每个区域的片段总数为最小gydF4y2Ba\({| {\rm{diag}}(C)\times A\times x-b|}_{2}\)gydF4y2Ba.详情见gydF4y2Ba补充信息gydF4y2Ba.gydF4y2Ba
WGBS反褶积的计算机模拟gydF4y2Ba
对心肌细胞进行模拟混合(gydF4y2BangydF4y2Ba= 4)、膀胱上皮(gydF4y2BangydF4y2Ba= 5)、乳腺上皮细胞(gydF4y2BangydF4y2Ba= 7),内皮细胞(gydF4y2BangydF4y2Ba= 19)和红细胞祖细胞(gydF4y2BangydF4y2Ba(3)用省略的方式。为此,保留一个样本,并使用剩余的204个样本重新进行分割和标记选择(每种细胞类型25个)。然后,我们通过采样和将10、3、1、0.3、0.1、0.03和0%的保留样本的读数混合到白细胞样本的背景中来模拟混合物。这样重复了十次。最后,使用UXM片段级算法对混合样本进行分析,标记来自缩减的(204)图谱,使用至少三个cpg的片段。使用wgbstools (v.0.1.0)进行合并、拆分和混合读取gydF4y2Ba54gydF4y2Ba.gydF4y2Ba
基于阵列的分析通过计算进行,对于每个混合片段集,在450K阵列(' wgbstools beta_to_450k ')中存在的约48万个CpG位点的平均甲基化水平。然后我们根据Moss等人的方法对这些数据进行反卷积。gydF4y2Ba28gydF4y2Ba(gydF4y2Bahttps://github.com/nloyfer/meth_atlasgydF4y2Ba).gydF4y2Ba
我们还模拟了四向混合物,其中背景血浆甲基组被模拟为来自白细胞的90%片段、来自血管内皮样本的7.5%片段和来自肝细胞样本的2.5%片段的组合。如上所述,这是通过保留三个样本(例如,心肌细胞、内皮细胞和肝细胞),然后对剩余的样本(202 = 205 - 3)重新进行分割和标记选择来完成的,以获得一组标记,然后用于片段级的混合物反褶积。gydF4y2Ba
WGBS反褶积gydF4y2Ba
白细胞及相应的血浆样本(gydF4y2BangydF4y2Ba= 23)进行如上处理,并使用WGBS甲基化图谱进行分析,包括1246个标记加上(对于血浆样本)额外的25个巨核细胞标记。来自28例SARS-CoV-2患者的52份血浆样本(参考文献)。gydF4y2Ba44gydF4y2Ba)下载为FASTQ文件,处理如上所述。由于这些样本的覆盖率较低(1-2×),我们将标记集从每种细胞类型的前25个标记扩展到前250个标记(补充表)gydF4y2Ba4 bgydF4y2Ba),还包括250个巨核细胞标记物gydF4y2Ba65gydF4y2Ba.路线图gydF4y2Ba4gydF4y2Ba和编码gydF4y2Ba5gydF4y2Ba样品按上述方法处理,并使用UXM算法进行分析。gydF4y2Ba
450K阵列数据的反褶积gydF4y2Ba
先前发表的450K阵列数据从癌症基因组图谱(肺和乳腺活检)下载gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba或GEO登记编号。GSE62640 (ref。gydF4y2Ba48gydF4y2Ba),并使用meth_atlas NNLS软件(gydF4y2Bahttps://github.com/nloyfer/meth_atlasgydF4y2Ba)使用我们的阵列适配图集(补充表gydF4y2Ba12gydF4y2Ba).乳房活检采用PAM50分类进行分组gydF4y2Ba66gydF4y2Ba.gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然组合报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
DNA甲基化数据的格式为bigWig (28,217,448 CpGs的位置和平均甲基化)和beta(类似的wgbstools兼容二进制格式)。GSE186458)。BigWig和hg38的beta文件也可用。片段级信息(pat格式,包括CpG起始指数、所有覆盖CpG的甲基化模式和具有精确的multiCpG模式的片段数)也可获得。fastq原始文件已存放在欧洲基因组-表型组档案馆(EGA),研究登录号:EGAS00001006791,可根据要求向EGA下载(通过地图集数据访问委员会)。gydF4y2Ba
代码的可用性gydF4y2Ba
代码可在gydF4y2Bagithub.com/nloyfer/wgbs_toolsgydF4y2Ba而且gydF4y2Bagithub.com/nloyfer/UXM_deconv。gydF4y2Ba
参考文献gydF4y2Ba
多,Y. &雪松,H. DNA甲基化原理及其对生物学和医学的影响。gydF4y2Ba《柳叶刀》gydF4y2Ba392gydF4y2Ba, 777-786(2018)。gydF4y2Ba
齐勒,m.j.等。绘制人类基因组的动态DNA甲基化图景。gydF4y2Ba自然gydF4y2Ba500gydF4y2Ba, 477-481(2013)。gydF4y2Ba
癌症基因组图谱研究网络。全面的基因组特征定义了人类胶质母细胞瘤基因和核心通路。gydF4y2Ba自然gydF4y2Ba455gydF4y2Ba, 1061-1068(2008)。gydF4y2Ba
昆达杰等人。111个参考人类表观基因组的综合分析。gydF4y2Ba自然gydF4y2Ba518gydF4y2Ba, 317-330(2015)。gydF4y2Ba
ENCODE项目联盟扩充了人类和小鼠基因组中DNA元素的百科全书。gydF4y2Ba自然gydF4y2Ba583gydF4y2Ba, 699-710(2020)。gydF4y2Ba
雷纽斯,L. E.等。纯化人血细胞中的差异DNA甲基化:对细胞谱系和疾病易感性研究的影响。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba7gydF4y2Ba, e41361(2012)。gydF4y2Ba
史密斯,Z. D.等。早期哺乳动物胚胎中DNA甲基化的独特调节阶段。gydF4y2Ba自然gydF4y2Ba484gydF4y2Ba, 339-344(2012)。gydF4y2Ba
汉纳姆,G.等人。全基因组甲基化谱揭示了人类衰老速度的定量观点。gydF4y2Ba摩尔。细胞gydF4y2Ba49gydF4y2Ba, 359-367(2013)。gydF4y2Ba
提图斯,a . J.,加利莫尔,R. M.,萨拉斯,L. a . &克里斯滕森,B. C. DNA甲基化的细胞型反褶积:最近应用综述。gydF4y2Ba嗡嗡声。摩尔,麝猫。gydF4y2Ba26gydF4y2Ba, r216-r224(2017)。gydF4y2Ba
Bibikova, M.等人。单CpG位点分辨率的高密度DNA甲基化阵列。gydF4y2Ba基因组学gydF4y2Ba98gydF4y2Ba, 288-295(2011)。gydF4y2Ba
莱曼-沃曼,R.等。利用循环DNA的甲基化模式鉴定组织特异性细胞死亡。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, e1826-e1834(2016)。gydF4y2Ba
郭,S.等。甲基化单倍型块的鉴定有助于异构组织样本的反褶积和来自血浆DNA的肿瘤组织起源映射。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba, 635-642(2017)。gydF4y2Ba
弗兰岑,J.等。DNA甲基化变化在长期体外细胞培养是由表观遗传漂变引起的。gydF4y2BaCommun。医学杂志。gydF4y2Ba4gydF4y2Ba, 598(2021)。gydF4y2Ba
Teschendorff, A. E., Zhu, T., Breeze, C. E. & Beck, S. EPISCORE:单细胞RNA-Seq数据中大块组织DNA甲基化的细胞类型反褶褶。gydF4y2Ba基因组医学杂志。gydF4y2Ba21gydF4y2Ba, 221(2020)。gydF4y2Ba
朱,T.等。泛组织DNA甲基化图谱能够在细胞型分辨率下硅分解人体组织甲基化组。gydF4y2BaNat方法。gydF4y2Ba19gydF4y2Ba, 296-306(2022)。gydF4y2Ba
万建民等。液体活检成熟:实现循环肿瘤DNA。gydF4y2BaNat. Rev. CancergydF4y2Ba17gydF4y2Ba, 223-238(2017)。gydF4y2Ba
Aravanis, a.m., Lee, M. & Klausner, R. D.用于早期癌症检测的新一代循环肿瘤DNA测序。gydF4y2Ba细胞gydF4y2Ba168gydF4y2Ba, 571-574(2017)。gydF4y2Ba
孙,K.等。通过全基因组甲基化测序进行血浆DNA组织定位,用于无创产前、癌症和移植评估。gydF4y2Ba国家科学院学报美国gydF4y2Ba112gydF4y2Ba, e5503-e5512(2015)。gydF4y2Ba
Kang, S.等。CancerLocator:使用无细胞DNA甲基化谱进行非侵入性癌症诊断和组织起源预测。gydF4y2Ba基因组医学杂志。gydF4y2Ba18gydF4y2Ba, 53(2017)。gydF4y2Ba
李,W.等。CancerDetector:使用无细胞DNA甲基化测序数据,在个人读取分辨率下进行超灵敏和非侵入性癌症检测。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e89(2018)。gydF4y2Ba
克莱因,E. A.等。使用独立验证集的靶向甲基化多癌早期检测试验的临床验证。gydF4y2Ba安。肿瘤防治杂志。gydF4y2Ba32gydF4y2Ba, 1167-1177(2021)。gydF4y2Ba
1000个基因组计划联盟。人类遗传变异的全球参考。gydF4y2Ba自然gydF4y2Ba526gydF4y2Ba, 68-74(2015)。gydF4y2Ba
米切纳,C. D. &索卡尔,R. R.分类问题的定量方法。gydF4y2Ba进化gydF4y2Ba11gydF4y2Ba, 130-162(1957)。gydF4y2Ba
Gu, G., Dubauskaite, J. & Melton, D. A.胰腺谱系的直接证据:NGN3gydF4y2Ba+gydF4y2Ba细胞是胰岛祖细胞,不同于导管祖细胞。gydF4y2Ba发展gydF4y2Ba129gydF4y2Ba, 2447-2457(2002)。gydF4y2Ba
Le Douarin, n.m.关于胰腺内分泌细胞的起源。gydF4y2Ba细胞gydF4y2Ba53gydF4y2Ba, 169-171(1988)。gydF4y2Ba
van Arensbergen等人。在胰腺器官发生过程中,Polycomb靶点的解除抑制使胰岛素生产β细胞采用神经基因活动程序。gydF4y2Ba基因组Res。gydF4y2Ba20.gydF4y2Ba, 722-732(2010)。gydF4y2Ba
弗兰克,D. B.等。早期谱系规范定义了小鼠肺中肺泡上皮的个体发育。gydF4y2Ba国家科学院学报美国gydF4y2Ba116gydF4y2Ba, 4362-4371(2019)。gydF4y2Ba
莫斯,J.等。全面的人类细胞型甲基化图谱揭示了循环无细胞DNA在健康和疾病中的起源。gydF4y2BaCommun Nat。gydF4y2Ba9gydF4y2Ba, 5068(2018)。gydF4y2Ba
Liu, m.c.等。利用游离细胞DNA中的甲基化特征进行敏感和特异的多种癌症检测和定位。gydF4y2Ba安。肿瘤防治杂志。gydF4y2Ba31gydF4y2Ba, 745-759(2020)。gydF4y2Ba
Gai, W.等。血浆DNA遗传-表观遗传组织作图在产前检测、移植和肿瘤学中的应用。gydF4y2BaeLifegydF4y2Ba10gydF4y2Ba, e64356(2021)。gydF4y2Ba
麦克林,C. Y.等。GREAT改善了顺式调控区域的功能解释。gydF4y2Ba生物科技Nat。》。gydF4y2Ba28gydF4y2Ba, 495-501(2010)。gydF4y2Ba
张,K.等。人类基因组中染色质可及性的单细胞图谱。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 5985-6001(2021)。gydF4y2Ba
恩斯特,J. &凯利斯,M. ChromHMM:自动色谱状态发现和表征。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 215-216(2012)。gydF4y2Ba
基里洛夫等人。核NF-kappaB在b细胞特异性Igkappa位点去甲基化中的作用。gydF4y2BaNat,麝猫。gydF4y2Ba13gydF4y2Ba, 435-441(1996)。gydF4y2Ba
斯塔德勒,m.b.等人。dna结合因子在远端调控区塑造小鼠甲基组。gydF4y2Ba自然gydF4y2Ba480gydF4y2Ba, 490-495(2011)。gydF4y2Ba
Schübeler, D. DNA甲基化的功能和信息含量。gydF4y2Ba自然gydF4y2Ba517gydF4y2Ba, 321-326(2015)。gydF4y2Ba
Yin, Y.等。胞嘧啶甲基化对人类转录因子DNA结合特异性的影响。gydF4y2Ba科学gydF4y2Ba356gydF4y2Ba, eaaj2239(2017)。gydF4y2Ba
费尔德曼,A.等。转录因子占用可介导调控区DNA甲基化的主动翻转。gydF4y2Ba公共科学图书馆麝猫。gydF4y2Ba9gydF4y2Ba, e1003994(2013)。gydF4y2Ba
海因茨,S.等。谱系决定转录因子的简单组合是巨噬细胞和B细胞鉴定所需的主要顺式调节元件。gydF4y2Ba摩尔。细胞gydF4y2Ba38gydF4y2Ba, 576-589(2010)。gydF4y2Ba
施特劳斯曼等人。人类基因组CpG岛甲基化谱的发育规划。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba16gydF4y2Ba, 564-571(2009)。gydF4y2Ba
Gal-Yam, E. N.等。PC3前列腺癌细胞系Polycomb抑制标记频繁切换和DNA高甲基化。gydF4y2Ba国家科学院学报美国gydF4y2Ba105gydF4y2Ba, 12979-12984(2008)。gydF4y2Ba
王,H.等。CTCF占用的广泛可塑性与DNA甲基化有关。gydF4y2Ba基因组Res。gydF4y2Ba22gydF4y2Ba, 1680-1688(2012)。gydF4y2Ba
Wiehle, L.等。胚胎干细胞中的DNA(去)甲基化控制ctcf依赖的染色质边界。gydF4y2Ba基因组Res。gydF4y2Ba29gydF4y2Ba, 750-761(2019)。gydF4y2Ba
程,A. P.等。通过甲基化分析,来源的无细胞DNA组织揭示了与COVID-19严重程度相关的显著细胞、组织和器官特异性损伤。gydF4y2Ba医生(北及Y)gydF4y2Ba2gydF4y2Ba, 411-422(2021)。gydF4y2Ba
郑,S. C.等。一种新的细胞型反褶积算法揭示了唾液、口腔和宫颈中免疫细胞的大量污染。gydF4y2Ba表观基因组学gydF4y2Ba10gydF4y2Ba, 925-940(2018)。gydF4y2Ba
李,Y.等。全基因组分析揭示了Polycomb在促进DNA甲基化谷低甲基化中的作用。gydF4y2Ba基因组医学杂志。gydF4y2Ba19gydF4y2Ba, 18(2018)。gydF4y2Ba
Nordin, M., Bergman, D., Halje, M., Engström, W. & Ward, A. Igf2/H19基因簇的表观遗传调控。gydF4y2Ba细胞Prolif。gydF4y2Ba47gydF4y2Ba, 189-199(2014)。gydF4y2Ba
霍尔,E.等。人类胰岛全基因组DNA甲基化模式的性别差异及其对基因表达、microRNA水平和胰岛素分泌的影响gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 522(2014)。gydF4y2Ba
Weisenberger, d.j.从癌症基因组图谱中描述DNA甲基化改变。gydF4y2Baj .中国。投资。gydF4y2Ba124gydF4y2Ba, 17-23(2014)。gydF4y2Ba
癌症基因组图谱网络。人类乳腺肿瘤的综合分子图谱。gydF4y2Ba自然gydF4y2Ba490gydF4y2Ba, 61-70(2012)。gydF4y2Ba
Pedersen, b.s., Eyring, K., De, S., Yang, i.v. & Schwartz, d.a.。预印在gydF4y2Bahttps://doi.org/10.48550/arXiv.1401.1129gydF4y2Ba(2014)。gydF4y2Ba
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 2078-2079(2009)。gydF4y2Ba
Tarasov, A., Vilella, A. J., Cuppen, E., Nijman, I. J. & Prins, P. Sambamba: NGS对齐格式的快速处理。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 2032-2034(2015)。gydF4y2Ba
Loyfer, N. & Kaplan, T. wgbstools:用于DNA甲基化测序数据表示、可视化和分析的计算套件。gydF4y2Bahttps://github.com/nloyfer/wgbs_toolsgydF4y2Ba(2022年11月28日访问)。gydF4y2Ba
Pedregosa, F.等人。Scikit-learn: Python中的机器学习。gydF4y2Baj·马赫。学习。Res。gydF4y2Ba12gydF4y2Ba, 2825-2830(2011)。gydF4y2Ba
维尔塔宁等人。SciPy 1.0: Python科学计算的基本算法。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 261-272(2020)。gydF4y2Ba
使用ggtree在树状结构上可视化数据。gydF4y2Ba咕咕叫。Protoc。生物信息学gydF4y2Ba69gydF4y2Ba, e96(2020)。gydF4y2Ba
肯特,W. J.等。UCSC的人类基因组浏览器。gydF4y2Ba基因组Res。gydF4y2Ba12gydF4y2Ba, 996-1006(2002)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
Kent, W. J., Zweig, A. S., Barber, G., Hinrichs, A. S. & Karolchik, D. BigWig和BigBed:允许浏览大型分布式数据集。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 2204-2207(2010)。gydF4y2Ba
Ramírez, F. et al. deepTools2:用于深度测序数据分析的下一代web服务器。gydF4y2Ba核酸测定。gydF4y2Ba44gydF4y2Ba, w160-w165(2016)。gydF4y2Ba
GTEx财团。人类组织中基因表达的遗传效应。gydF4y2Ba自然gydF4y2Ba550gydF4y2Ba, 204-213(2017)。gydF4y2Ba
卡斯特罗蒙德拉贡,J. A.等。JASPAR 2022:转录因子结合谱开放获取数据库的第9个版本。gydF4y2Ba核酸测定。gydF4y2Ba50gydF4y2Ba, d165-d173(2022)。gydF4y2Ba
Loyfer, N. & Kaplan, T. UXM:使用DNA甲基化测序数据对整个组织和血浆样本进行计算反褶积的片段级算法。gydF4y2Bahttps://github.com/nloyfer/UXM_deconvgydF4y2Ba(2022年11月28日访问)。gydF4y2Ba
莫斯,J.等。血浆和血小板中的巨核细胞和红细胞DNA。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/2022.10.03.510502gydF4y2Ba(2022)。gydF4y2Ba
帕克,J. S.等人。基于内在亚型的乳腺癌监督风险预测因子。gydF4y2Baj .中国。肿瘤防治杂志。gydF4y2Ba27gydF4y2Ba, 1160-1167(2009)。gydF4y2Ba
确认gydF4y2Ba
我们感谢H. Cedar和N. Friedman的富有洞察力的讨论。我们还要感谢Dor、Kaplan和Rosenfeld实验室的成员。这项工作得到了GRAIL、阿尔茨海默病药物发现基金会、人类胰岛研究网络(编号:HIRN UC4DK116274和UC4DK104216)、Ernest和Bonnie Beutler基因组医学卓越研究计划、Alex U Soyka胰腺癌基金、以色列科学基金会、Waldholtz/Pakula家族、Robert M.和Marilyn Sternberg家族慈善基金会、Helmsley慈善信托基金和DON基金会(to Y.D.)、以色列科学基金会(编号:104216)的资助。1250/18至T.K.)和跨学科数据科学研究中心(T.K., Y.D.和B.G.)。nl得到了CIDR数据科学和莱布尼茨奖学金的支持。Y.D.拥有沃尔特和格里塔·斯蒂尔主席和心脏研究基金。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
a.a., g.c., r.s., b.g., T.K.和Y.D.构想并发起了这个项目,并设计了实验。焦燕雄D.B个终身制。,整理,广义相对论,有限责任,新罕布什尔州,漆包线,a . Pikarsky a . Khalaileh G.Z, rg, M.A.G,坜,n ., a . Korach O.W, U.I,电子工程,V.Y,廖曜生,D.R.G, K.L.S, H.D。每年,A.M.J.S.和M.G.提供材料。J. Magenheim, A. Peretz, A. Klochendler, i.f.f。, m.h., T.P.和Z.D.进行了实验。g.c., j.b., h.a., p.m., s.n., O.V.和A.J.进行了测序和分析。n.l. S.S.-P。,J. Moss and T.K. developed and performed computational analyses. N.L., Y.D. and T.K. wrote the manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
这项工作得到GRAIL, Inc.的支持。g.c., j.b., a.a., O.V.和A.J.是GRAIL, Inc.的员工,股东和/或创始人。j。m。j。f。,R.S., Y.D., B.G. and T.K. have filed patents on cfDNA analysis technology. The remaining authors declare no competing interests.
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Matteo Pellegrini和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1健康细胞类型的人类全基因组DNA甲基化图谱。gydF4y2Ba
从成人中获得205份健康样本,分离并深度测序(WGBS,平均深度≥30倍),形成全面的人类细胞类型特异性甲基化图谱。gydF4y2Ba
扩展数据图2样品制备和纯度。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba手术时获得新鲜组织并分离(每种组织类型优化),然后用抗体孵育,并进行流式细胞仪分类。用qRT-PCR分析分选细胞的关键细胞类型特异性基因,或用靶向PCR分析细胞类型特异性DNA甲基化标记。DNA甲基化也使用全基因组亚硫酸氢盐测序进行分析。(gydF4y2BaBgydF4y2Ba胰腺内分泌细胞类型的FACS分选示例。左图:β细胞标记物c肽(x轴)与α细胞标记物胰高血糖素(y轴)的染色。注意未观察到双阳性细胞。中图:c-肽(x轴)与δ细胞标记物生长抑素(y轴)染色。右图:未染色对照(仅添加荧光二抗,未添加一抗)。(gydF4y2BaCgydF4y2Ba)利用靶向PCR对样品纯度进行片段级验证。细胞类型特异性标记使用预先存在的450K数据设计,覆盖4-7个相邻的cpg。所示为每种细胞类型(包括内皮细胞和白细胞)中未甲基化分子的百分比。颜色渐变从完全未甲基化的分子(allT)到除了一个CpG (allT-1)以外的所有未甲基化分子(allT-1),等等。报道了hg19中腺泡细胞、α、β、δ、导管和内皮标记物的扩增子位置(从左到右)。(gydF4y2BaDgydF4y2Ba)使用地图集WGBS数据对相同位置进行分段级验证。y轴为未甲基化片段(≥4 CpGs)的百分比。正如这些标记所示,在该目标细胞类型中,大约90%的分子是未甲基化的,而在其他细胞类型中,这一比例不到5%,因此,使用一组独立选择的DMRs强调了DNA甲基化图谱的纯度。gydF4y2Ba
扩展数据图3使用图谱标记对胰腺、肺、心脏、肝脏、乳房和胃肠道的纯度估计。gydF4y2Ba
从所选的差异甲基化标记中,≥4 CpGs的片段中未甲基化片段的百分比(y轴)可以作为地图集纯度的(低)估计。在这里,我们展示了每种细胞类型的一个这样的标记,从前25个标记中选择,并使用片段级分析来证明与来自相同组织或环境的其他细胞类型相比,目标细胞类型的纯度。(gydF4y2Ba一个gydF4y2Ba)胰腺。(gydF4y2BaBgydF4y2Ba肺)。(gydF4y2BaCgydF4y2Ba)的心。(gydF4y2BaDgydF4y2Ba)肝脏。(gydF4y2BaEgydF4y2Ba乳房)。(gydF4y2BaFgydF4y2Ba)胃肠道。对于大多数细胞类型,目标细胞类型中90%的分子是未甲基化的,相比之下,其他类型的分子只有不到5%。这是一种低估,因为每种细胞类型都可能出现一些异质性,反映了随机噪声、细胞状态、年龄或环境变化。gydF4y2Ba
扩展数据图4来自不同个体的相同细胞类型的生物复制显示出惊人的低甲基化差异块率。gydF4y2Ba
我们重点研究了37种重复次数≥3次的细胞亚型(例如来自特定组织的内皮细胞),并测量了不同重复次数中甲基化差异达50%(绝对δ β)的甲基化块(≥3 CpGs)的平均百分比(如图y轴所示)。几乎所有细胞亚型(36/37)的差异都≤0.5%,这表明重复之间的保守程度非常高。红色虚线标记了两个不同细胞类型的随机样本之间的平均差异块数(4.9%)。箱形图标记中位数和四分位范围(IQR),有1.5*IQR须。gydF4y2Ba
图5人类甲基组的无监督凝聚聚类。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)同图。gydF4y2Ba2gydF4y2Ba,按胚层发育谱系着色,包括内胚层(绿色)、中胚层(蓝色)和外胚层(红色)。(gydF4y2BaBgydF4y2Ba)同图。gydF4y2Ba2gydF4y2Ba,为路线图表观基因组DNA甲基化图谱。gydF4y2Ba
扩展数据图6 953种细胞类型特异性标记的标记特异性。gydF4y2Ba
对于每个单元格类型(行),我们绘制出前25个标记(显示为方框)。对于每个标记物,我们比较了目标样本(蓝点)中未甲基化片段(≥3 CpGs)的百分比与背景样本(金点)中的百分比。蓝色和金色条形图分别表示所有目标和背景样本的平均比例。gydF4y2Ba
扩展数据图7其他atlas细胞类型中假定增强子的标记。gydF4y2Ba
包括B细胞(左上)、肝细胞(右上)、胃/小肠/结肠上皮(左下)和心肌细胞/心脏成纤维细胞(右下)的前250个未甲基化标记。灰线标记相同的ChIP-seq/ATAC/DNase/chromHMM信号,在所有11,371个未甲基化标记中平均(每种细胞类型的前250个)。gydF4y2Ba
扩展数据图8片段级分析gydF4y2Ba在网上gydF4y2Ba混合。gydF4y2Ba
显示的是gydF4y2Ba在网上gydF4y2Ba四种细胞类型的模拟,计算中以不同比例混合90%白细胞,7.5%血管内皮细胞和2.5%肝细胞的血浆样混合物。使用我们的图谱(红色)分析每种混合物,并与Moss等人(灰色)进行比较。箱形图显示了10次模拟的平均贡献,带有1个SD误差条。gydF4y2Ba
扩展数据图10先前发表的450K DNA甲基化阵列数据的反褶积。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)胰岛甲基组的反褶积。甲基化阵列来自53名男性和34名女性非糖尿病捐赠者gydF4y2Ba48gydF4y2Ba分析了atlas甲基组,揭示了详细的细胞组成,包括以前未鉴定的α和δ细胞。细胞组成的性别差异无统计学意义。(gydF4y2BaBgydF4y2Ba) TCGA中865个肺甲基组的分析gydF4y2Ba49gydF4y2Ba.基于wgbs的肺泡上皮和肺支气管上皮细胞标记揭示了443个LUAD、11个SCLC、337个LUSC、32个正常相邻(LUAD)和42个正常相邻(LUSC)肺甲基组的不同细胞群。请注意,在肺腺癌中只鉴定出肺泡细胞DNA,而小细胞肺癌和鳞状细胞癌也含有支气管DNA,这与每种类型肺癌的假定细胞起源一致。注意上皮细胞在肺腺癌和正常肺中都是少数。这可能是由于在正常肺或肺癌的大量制备中有丰富的间质细胞。(gydF4y2BaCgydF4y2Ba)来自TCGA的721例癌变和97例正常乳腺活检的DNA甲基化。利用wgbs为基础的乳腺管腔和基底上皮细胞标记物研究TCGA中的细胞组成gydF4y2Ba50gydF4y2Ba使用PAM50(一种基于50个基因表达的分类)将其分为5个亚型gydF4y2Ba66gydF4y2Ba.与健康乳腺活检相比,观察到正常样、基底样、管腔A、管腔B和her2富集PAM50亚型的不同细胞组成。乳腺癌中乳腺基底细胞的低比例可能是由于正常乳腺和乳腺癌中非上皮细胞的丰富所致。箱形图标记中位数和四分位范围(IQR),有1.5*IQR须。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充表图例;细胞分选:FACS图和RNA/甲基化富集;甲基化片段的全基因组分割;类型特异性标记的选择;UXM片段级反褶积算法;纯度估计:各样品中未甲基化片段的比例;COVID-19患者cfDNA组成分析;离解协议;附录:RBC裂解缓冲液(X10);补充表18:流式细胞仪抗体; and Supplementary Table 19: reagents used.
补充表gydF4y2Ba
该文件包含补充表1-17(图例请参阅主要补充信息PDF)。gydF4y2Ba
补充数据集1gydF4y2Ba
全基因组的每一种细胞类型的未甲基化区域,注释。Zip文件包含39个床文件,每个床文件包含所有基因组区域(至少4个CpGs的块,其中至少85%的测序片段在至少85%的覆盖CpGs中未甲基化)。床文件信息包括位置(chr, from, to, CpG_from, CpG_to, hg19), HOMER和邻近基因的区域注释,以及当一个区域距离转录起始位点1 kb或更小,重叠CpG岛的注释,该细胞类型或类似细胞中的chromHMM注释,H3K4me3(启动子),H3K27ac(基因调控),H3K4me1(增强子),H3K27me3的ChIP-seq峰注释,重叠ATAC-seq峰,CTCF结合位点和CTCF ChIP-seq峰的存在。gydF4y2Ba
补充数据集2gydF4y2Ba
每一种细胞类型的全基因组未甲基化区域集合,重叠H3K27ac,但不重叠J3K4me3, ChIP-seq峰值,已注释。包含床文件的Zip文件,为每种细胞类型列出一组假定的增强子,定义为低甲基化区域的子集(扩展数据集1),标记为活性基因调控染色质标记(H3K27ac峰值),但缺乏启动子标记(H3K4me3峰值)。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
罗佛,N.,马根海姆,J.,佩雷兹,A.。gydF4y2Baet al。gydF4y2Ba正常人类细胞类型的DNA甲基化图谱。gydF4y2Ba自然gydF4y2Ba613gydF4y2Ba, 355-364(2023)。https://doi.org/10.1038/s41586-022-05580-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05580-6gydF4y2Ba
这篇文章被引用gydF4y2Ba
绘制人类甲基组的细节gydF4y2Ba
自然评论遗传学gydF4y2Ba(2023)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba