摘要
通过对生物体的遗传密码进行工程设计,可以防止病毒感染和基因转移,从而为自然生态系统提供防火墙1,2,3.,4,5,6.然而,许多病毒和可移动的遗传元件编码翻译装置的部分7,8,9这可能会导致基于基因代码的防火墙失效。在这里,我们表明这种移动转移rna (tRNAs)使基因转移和允许病毒复制大肠杆菌尽管全基因组删除了64个密码子中的3个,以及之前必不可少的同源tRNA和释放因子基因。然后,我们通过发现病毒trna来建立遗传防火墙,这些病毒trna提供了非常有效的密码子重新分配,使我们能够开发出具有氨基酸交换遗传密码的细胞,在翻译过程中将六个丝氨酸密码子中的两个重新分配给亮氨酸。这种氨基酸交换的遗传密码使细胞通过错误翻译病毒蛋白质组而抵抗病毒感染,并通过工程依赖丝氨酸密码子来产生需要亮氨酸的蛋白质来防止合成遗传信息的逃逸。由于这些细胞可能比野生生物具有病毒抗性的选择优势,我们也重新利用第三个密码子,通过依赖自然界中不存在的氨基酸来生物包含这种抗病毒宿主10.我们的结果可能为一种通用策略提供基础,使任何生物都能安全地抵抗所有天然病毒,并防止遗传信息流入和流出转基因生物。
这是订阅内容的预览,通过你所在的机构访问
访问选项
访问《自然》和其他54种《自然》杂志
获取Nature+,我们最超值的在线订阅
每月29.99美元
随时取消
订阅这本杂志
收到51个印刷问题和在线访问
199.00美元一年
每期仅需3.90美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算


数据可用性
来自全基因组测序、转录组和tRNA-seq实验的原始数据已以BioProject ID保存在Sequence Read Archive中PRJNA856259.本研究的tRNA数据和生成的序列包含在补充数据文件中。质谱和蛋白质组测量结果已存入MassIVE (MSV000089854;https://doi.org/10.25345/C5FF3M41W)65.Syn61Δ3(ev5) ΔrecA(ev1)菌株来源于Addgene(菌株no.;189857)。我们无法沉积Ec_Syn61∆3- sl和Ec_Syn61∆3 adk。d6一个t Addgene owing to the incompatibility of Addgene’s methods of strain distribution and growth medium requirements, but all materials used in this study are freely available from the corresponding authors for academic research use upon request. The PHROG HMM database is available athttps://phrogs.lmge.uca.fr和裁判。51.组装的注释基因组大肠杆菌Syn61亚株Syn61Δ3(ev5)可在补充数据中获得4, REP噬菌体注释基因组已按登录号存入NCBI GenBankOQ174500,OQ174501,OQ174502,OQ174503,OQ174504,OQ174505,OQ174506,OQ174507,OQ174508,OQ174509,OQ174510而且OQ174511.源数据提供了这篇论文。
参考文献
丘奇,g.m. &里吉斯,E。再生:合成生物学如何重塑自然和人类(Basic Books, 2014)。
Lajoie, M. J.等。基因组编码的生物体扩展了生物功能。科学342, 357-360(2013)。
马,N. J.和艾萨克斯,F. J.基因组编码广泛地阻碍水平转移遗传元件的传播。细胞系统。3., 199-207(2016)。
罗伯逊,W. E.等人。意义密码子重配使病毒抗性和编码聚合物合成。科学372, 1057-1062(2021)。
Ostrov, N.等人。针对57个密码子基因组的设计、合成和测试。科学353, 819-822(2016)。
藤野,T., Tozaki, M. &村上,H.氨基酸交换遗传密码。ACS合成器。医学杂志。9, 2703-2713(2020)。
Abrahão, J.等。尾巨图潘病毒拥有已知病毒圈中最完整的转译器官。Commun Nat。9, 749(2018)。
莫加多,S. & Vicente, a.c . tRNA基因及其在病毒基因组中的组织的全球硅内情景。病毒11, 180(2019)。
al - shayeb, B.等。来自地球生态系统的巨大噬菌体。自然578, 425-431(2020)。
曼德尔,d.j.等人。通过合成蛋白质设计对转基因生物的生物遏制。自然518, 55-60(2015)。
邹,等。开发噬菌体抗性的系统策略大肠杆菌菌株。Commun Nat。13, 4491(2022)。
巴隆,P. W.等。生物制造中的病毒污染及其对新兴疗法的影响。生物科技Nat。》。https://doi.org/10.1038/s41587-020-0507-2(2020)。
抗噬菌体工业发酵菌株:从摇篮到CRISPR/Cas9。微生物学。Biotechnol。45, 1003-1006(2018)。
Ostrov, N.等人。改变遗传密码的合成基因组。咕咕叫。当今。系统。医学杂志。https://doi.org/10.1016/j.coisb.2020.09.007(2020)。
弗雷登斯,J.等人。全合成大肠杆菌用一个重新编码的基因组。自然https://doi.org/10.1038/s41586-019-1192-5(2019)。
杨,J. Y.等。宿主翻译机制的退化驱动病毒中tRNA的获取。细胞系统。12, 771-779(2021)。
彼得斯,s.l.等人。人类微生物噬菌体使用替代遗传编码的实验验证。Commun Nat。13, 5710(2022)。
博尔赫斯,a.l.等人。噬菌体中广泛的停止密码子重编码可能调节裂解基因的翻译。Microbiol Nat。7, 918-927(2022)。
Abe, T.等人tRNADB-CE 2011: tRNA基因数据库由专家手工策划。核酸测定。39, d210-d213(2011)。
Alamos, P.等人。嗜酸细菌的可移动遗传元件中tRNAs编码的功能。RNA杂志。15, 518-527(2018)。
Santamaría-Gómez, J.等。一个隐藏的tRNA基因操纵子在翻译应激下存活的作用。核酸测定。49, 8757-8776(2021)。
布斯塔曼特等人。冰Afe1,从生物采矿细菌中主动切除遗传元素Acidithiobacillus ferrooxidans.J. Mol.微生物。Biotechnol。22, 399-407(2012)。
鲍登,R. J., Simas, J. P, Davis, A. J. & Efstathiou, S.鼠γ疱疹病毒68编码trna样序列,在潜伏期表达。维罗尔将军。78, 1675-1687(1997)。
Maffei, E.等。系统探索大肠杆菌噬菌体-宿主与BASEL噬菌体集合的相互作用。公共科学图书馆杂志。19, e3001424(2021)。
Brok-Volchanskaya, V. S.等。噬菌体T4 SegB蛋白是与相关噬菌体联合感染时发生的T4 tRNA基因区域优先遗传所需的归巢内切酶。核酸测定。36, 2094-2105(2008)。
Miles, Z. D., McCarty, R. M., Molnar, G. & Bandarian, V.环氧队列苷(oQ)还原酶的发现揭示了halorespiration和tRNA修饰之间的相似性。国家科学院学报美国108, 7368-7372(2011)。
刘,R.-J。,Long, T., Zhou, M., Zhou, X.-L. & Wang, E.-D. tRNA recognition by a bacterial tRNA Xm32 modification enzyme from the SPOUT methyltransferase superfamily.核酸测定。43, 7489-7503(2015)。
施密特,M. &库比什金,V.如何量化基因防火墙?基于极性的遗传密码工程度量。ChemBioChem22, 1268-1284(2021)。
杨,X.-L。et al。水晶人tRNA合成- tRNA复合物的两种构象:蛋白质合成的意义。EMBO J。25, 2919-2929(2006)。
小林,T.等人。用于遗传密码扩展的酪氨酸-tRNA合成酶的正交tRNA特异性的结构基础。Nat。结构。摩尔。杂志。10, 425-432(2003)。
Giege, R., Sissler, M. & Florentz, C. tRNA身份的普遍规则和特质特征。核酸测定。26, 5017-5035(1998)。
丘奇,G.贝恩斯,B.和投手,E.基因组工程的层次组装方法。PCT/US2006/001427专利申请(2007)。
Zürcher, J. F.等。重构的遗传密码使双向遗传隔离成为可能。科学https://doi.org/10.1126/science.add8943(2022)。
Łoś, J. M., Golec, P., wichgrzyn, G., wichgrzyn, A. & Łoś, M.简单的电镀方法大肠杆菌噬菌体在标准条件下形成非常小的斑块或没有斑块。达成。Env。Microbiol。74, 5113-5120(2008)。
艾伯敦,殷志强,殷志强噬菌体:方法和方案卷1(编Clokie, M. R. J. & Kropinski, a.m.) 161-174https://doi.org/10.1007/978-1-60327-164-6_17(胡玛纳,2009)。
Serwer, P., Hayes, S. J., Thomas, J. a . & Hardies, S. C.传播缺失的噬菌体:一个新类的大型噬菌体。病毒学J。4, 21(2007)。
王杰,Yashiro, Y., Sakaguchi, Y., Suzuki, T. & Tomita, K.接触依赖生长抑制蛋白和翻译因子对tRNA切割的机制研究。核酸测定。50, 4713-4731(2022)。
Tomita, K., Ogawa, T., Uozumi, T., Watanabe, K. & Masaki, H.一种细胞毒性核糖核酸酶,特异性地在其反密码子环上切割四个异接受精氨酸trna。国家科学院学报美国97, 8278-8283(2000)。
Takai, K., Takaku, H. & Yokoyama, S.不同反密码子的未修饰tRNA分子在序列背景下的体外密码子阅读特异性大肠杆菌tRNASer1。物化学。Biophys。Commun >,257, 662-667(1999)。
Takai, K., Okumura, S., Hosono, K., Yokoyama, S. & Takaku, H.在人工tRNA的摆动位置上进行单个尿苷修饰,可以增强an的摆动大肠杆菌无细胞翻译系统。2月。https://doi.org/10.1016/s0014 - 5793 (99) 00255 - 0(1999)。
Kunjapur, a.m.等。经过不断进化和与哺乳动物细胞共培养后,合成的营养不良仍然保持稳定。科学。睡觉。7, eabf5851(2021)。
罗夫纳,A. J.等人。编码生物体,使其依赖合成氨基酸。自然518, 89-93(2015)。
Nyerges,。et al。一种高度精确和便携的基因组工程方法允许比较细菌物种的突变效应。国家科学院学报美国https://doi.org/10.1073/pnas.1520040113(2016)。
万尼尔,t.m.等人。基因组编码的适应性进化大肠杆菌.国家科学院学报美国https://doi.org/10.1073/pnas.1715530115(2018)。
柯奇伯格,P. C. & Ochman, H.一种全局宏基因组定义的gokushovirus的复活。eLife9, e51599(2020)。
陈,Y.等。多重碱基编辑将人类基因组中的TAG转化为TAA密码子。Commun Nat。13, 4482(2022)。
Boeke, J. D.等人。基因组计划——写作。科学https://doi.org/10.1126/science.aaf6850(2016)。
戴俊杰,鲍凯,罗志强,蒋淑娟,蔡勇。Sc3.0:酵母基因组的改进与最小化。基因组医学杂志。21, 205(2020)。
博尼拉,N.等。噬菌体的水龙头-一个快速和有效的方案制备噬菌体实验室储备。PeerJ4, e2261(2016)。
Seemann, T. Prokka:快速原核基因组注释。生物信息学30., 2068-2069(2014)。
Terzian, P.等人。原核病毒蛋白家族利用远程同源性聚类。NAR Genomics Bioinform。3., lqab067(2021)。
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。Nat方法。9, 357-359(2012)。
Deatherage, D. E. & Barrick, J. E.使用breseq从下一代测序数据识别实验室进化微生物的突变。方法分子生物学。1151, 165-188(2014)。
古道尔,e.c.a.等。基本基因组大肠杆菌k - 12。mBio9, e02096-17(2018)。
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组医学杂志。15, 550(2014)。
Jiang, W., Bikard, D., Cox, D., Zhang, F. & Marraffini, L. A.使用CRISPR-Cas系统对细菌基因组进行rna引导编辑。Nat,生物技术。31, 233-239(2013)。
乌门霍夫,K.等人。利用基因组洗牌和CRISPR/ cas辅助的MAGE在全基因组范围内消除移动遗传元件,可以有效稳定细菌底盘。ACS合成器。医学杂志。6, 1471-1483(2017)。
Szili, P.等。通过垫脚石突变降低对平衡双靶向抗生素的敏感性的快速进化。Antimicrob。代理商和Chemother。63, e00207-19(2019)。
Kunjapur, a.m.等。区分非标准氨基酸的工程翻译后校对。国家科学院学报美国115, 619-624(2018)。
陈鹏鹏,林伯勇,Mak, A. J. & Lowe, t.m. tRNAscan-SE 2.0:改进的转移RNA基因检测和功能分类。核酸测定。49, 9077-9096(2021)。
侯赛因,A.等。为工程稳定的遗传系统自动化设计数千个非重复部件。生物科技Nat。》。38, 1466-1475(2020)。
莫勒,K.等人。MS-READ:氨基酸掺入量的定量测定。Biochim。Biophys。学报。1861, 3081-3088(2017)。
wi涅夫斯基,J. R., Zougman, A., Nagaraj, N. & Mann, M.蛋白质组分析的通用样品制备方法。Nat方法。6, 359-362(2009)。
Käll, L., Storey, J. D. & Noble, W. S.与串联质谱鉴定的多肽相关的后测误差概率的非参数估计。生物信息学24, i42-i48(2008)。
Nyerges, A.等。交换的遗传密码阻止病毒感染和基因转移(MSV000089854)。巨大的https://doi.org/10.25345/C5FF3M41W(2023)。
确认
我们感谢G. Pósfai(匈牙利生物研究中心)分享MDS42和J. W.。Chin团队(英国医学研究理事会分子生物学实验室)通过Addgene共享Syn61∆3。这项研究的资金支持由美国能源部提供,资助号为DE-FG02-02ER63445,国家科学基金会资助号为2123243(均授予G.M.C.)。M.B.感谢美国国立卫生研究院NIGMS (R35GM133700)、大卫和露西尔·帕卡德基金会、皮尤慈善信托基金会和阿尔弗雷德·p·斯隆基金会的支持。A.N.得到了EMBO LTF 160-2019长期奖学金的支持。我们感谢A. Millard的实验室为噬菌体注释提供PHROG HMM数据库,感谢GenScript美国公司对DNA合成的支持,感谢D. Snyder, K. Harris和宾夕法尼亚州匹兹堡米格公司的所有成员对DNA和RNA测序的支持。感谢吴涛对我们的支持,感谢沈勇和严胜(北京基因组研究所生物化学研究所)在基因组编码方面的合作,感谢B. Hajian的图形设计和插图的帮助。
作者信息
作者及隶属关系
贡献
A.N.开发了该项目,领导分析,并根据所有作者的输入撰写了手稿。A.N.和G.M.C.监督研究。S.V.进行tRNA低浓缩铀抑制筛选和sfGFP表达分析,并协助构建pLS质粒和生物遏制实验。R.F.协助进行实验,并进行自适应实验室进化和生长速率测量。S.V.O、E.A.R.和M.B.为噬菌体分离提供了环境样本,进行了复制分析,并为噬菌体实验和基因组分析提供了支持。m.l., K.C.和F.H.进行了DNA合成;M.B.-T。,A.C.-P. and E.K. supported the project. B.B. carried out MS/MS analyses. K.N. and J.A.M. provided reagents for tRNA-seq experiments.
相应的作者
道德声明
相互竞争的利益
哈佛医学院已经提交了与这项工作相关的临时专利申请,a.n., S.V.和G.M.C.被列为发明人。m.l., K.C.和F.H.受雇于GenScript USA Inc.,但该公司在设计或执行实验中没有任何作用。G.M.C.是以下公司的创始人,他拥有相关的财务利益:GRO Biosciences, EnEvolv (Ginkgo Bioworks)和64x Bio。通用汽车其他潜在相关的财务利益列于http://arep.med.harvard.edu/gmc/tech.html.
同行评审
同行评审信息
自然感谢Benjamin Blount和其他匿名审稿人对本工作的同行评审所作的贡献。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展的数据图形和表格
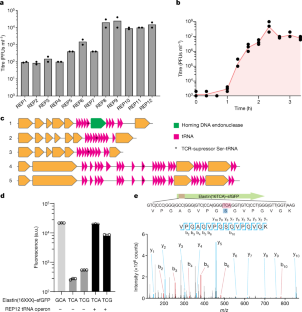
扩展数据图1病毒丝氨酸tRNAs将TCA密码子解码为丝氨酸,但Syn61∆3阻碍含有基因组tRNA-Ser的病毒的复制佐治亚大学.
一个)病毒TCR抑制tRNAs将TCA密码子解码为丝氨酸。翻译后的TCR密码子在弹性蛋白中的氨基酸同一性16柠檬酸-sfGFP-His6经串联质谱证实为表达tRNA-Ser的Syn61∆3佐治亚大学的埃希氏杆菌属噬菌体IrisVonRoten (GenBank ID MZ501075)24.图中为所分析的弹性蛋白的氨基酸序列和MS/MS谱16柠檬酸肽。MS/MS数据收集1次。b) Syn61∆3阻断含有基因组tRNA-Ser的病毒的复制佐治亚大学.图为12种含tRNA基因的噬菌体滴度24,在MDS42和Syn61上生长24 h后。除MZ501058外,所有被分析的噬菌体都含有基因组tRNA-Ser佐治亚大学根据我们的筛选提供TCR抑制活性的tRNA(图。1 b、补充数据1).MDS42和Syn61∆3的早期指数期培养以MOI为0.001感染相应的噬菌体,孵育24 h后测定游离噬菌体滴度。测量在n = 3个独立实验中进行(即.、mds42 + mz501058、mz501065、mz501075、mz501105、mz501106;Syn61∆3 + MZ501046, MZ501067, MZ501066, MZ501096, MZ501074, MZ501098)或n = 2个独立实验(即.、mds42 + mz501046、mz501066、mz501067、mz501074、mz501089、mz501096、mz501098;Syn61∆3 + MZ501058、MZ501065、MZ501075、MZ501089、MZ501105、MZ501106);虚线表示不含细菌细胞的输入噬菌体滴度(即,滴度为420 PFU/ml);点表示来自独立实验的数据;柱状图代表平均值;误差条表示基于n = 3个独立实验的扫描电镜。
扩展数据图2病毒通过用病毒编码的tRNA快速补充细胞tRNA池来克服基于遗传代码的耐药性。
使用tRNAseq(方法)定量了REP12噬菌体感染后Syn61∆3细胞中宿主和病毒tRNA表达的时间过程动力学。内生服务公司而且serW主机Syn61∆3的trna分别以绿色和蓝色突出显示,而tRNA-Ser . 3为佐治亚大学以红色突出显示的是REP12病毒。REP12病毒trna用浅红色表示;灰色为Syn61∆3的内源性trna。数据表示TPM平均值(transcript/million)。本文提供了原始数据。
图3基于trnaseq的CCA tRNA尾加REP12 tRNA- ser检测佐治亚大学.
即使噬菌体编码tRNA- ser, tRNA尾部也被修改为CCA佐治亚大学不对CCA端进行编码。基因组噬菌体编码的tRNA-Ser序列佐治亚大学用洋红色突出显示。黑色字母表示单次实验在噬菌体附着后直接从REP12感染的Syn61∆3细胞中读取tRNAseq测序。
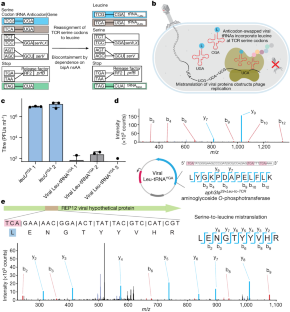
图6基于trnaseq的病毒Leu-tRNA定量佐治亚大学和Leu-tRNA注册会计师在Ec_Syn61的tRNAome中∆3-SL。
使用tRNAseq(方法)定量Ec_Syn61∆3-SL的tRNA水平。病毒Leu-tRNA佐治亚大学和Leu-tRNA注册会计师用红色标出的是宿主的内源性大肠杆菌血清tRNA用橙色突出显示。tRNAseq数据收集一次。数据表示TPM (transcript/million)。本文提供了原始数据。
图7 Ec_Syn61∆3-SL细胞中TCT密码子丝氨酸-亮氨酸误译
一个丝氨酸-亮氨酸误译TufA肽的MS/MS谱。b)野生型TufA肽的MS/MS谱。图中为Ec_Syn61∆3- sl表达的TufA蛋白片段的氨基酸序列和MS/MS谱及其基因组序列,其中丝氨酸编码的TCT密码子(如面板所示)b)被部分误译为亮氨酸(如面板所示)一个).实验通过分析表达Leu9-tRNA的Ec_Syn61∆3-SL细胞的总蛋白质组进行YGA从埃希氏杆菌属通过串联质谱分析噬菌体OSYSP (GenBank ID MF402939.1)。MS/MS数据收集1次。
扩展数据图8 Syn61Δ3(ev5)、Syn61Δ3(ev5)Δ的加倍时间和增长曲线recA3, Syn61ΔΔ(ev5)recA(ev1), Ec_Syn61∆3-SL。
一个)倍增次数Syn61Δ3(ev5),倍增次数Syn61Δ3(ev5)ΔrecA3, Syn61ΔΔ(ev5)recA(ev1)和Ec_Syn61∆3-SL,根据增长曲线计算(如图所示)b,c,d)在标准实验室条件下的富细菌培养基中。b) Syn61Δ3(ev5)、Syn61Δ3(ev5)Δ的生长曲线recA、Syn61Δ3(ev5)ΔrecA(ev1)在LBL肉汤中。c) Syn61Δ3(ev5)和Syn61Δ3(ev5)Δ的生长曲线recA(ev1)在2×YT肉汤。d在含50 μg/ml卡那霉素的2×YT肉汤中,Ec_Syn61∆3-SL的生长曲线。在37°C和OD条件下,在通风摇瓶中进行有氧培养600在指数增长期间进行测量(方法)。数据曲线和条形表示平均值。误差条表示基于n = 3个独立实验的标准偏差。
扩展数据图9 Ec_Syn61∆3-SL在环境样品中能够抵抗病毒。
(一个)噬菌体富集实验采用Ec_Syn61∆3-SL,表达kp86910.1 tRNA24 LeuYGA和MF402939.1 tRNA9 LeuYGA作为东道主。(b)噬菌体富集实验采用Ec_Syn61∆3-SL,表达kp86910.1 tRNA24 LeuYGA和MF402939.1 tRNA21 LeuYGA作为东道主。噬菌体富集实验是通过将Ec_Syn61∆3-SL的早期指数培养物与10 ml环境样品混合物混合进行的,其中含有我们研究中的样品2-13的混合物(扩展数据表)1).经过两个富集周期(方法:补充说明),过滤灭菌的培养上清与噬菌体敏感混合大肠杆菌将MDS42细胞置于上端琼脂上,并在LBL琼脂板上进行病毒滴度测定。在n = 2个独立重复中进行富集实验,结果相同。(c)溶解性大肠杆菌10后MDS42噬菌体斑块3.-倍稀释的环境样品混合物。d)在Ec_Syn61上富集前后环境样品混合的裂解噬菌体滴度∆3-SL。点表示基于三个独立实验的未富集样本的病毒滴度大肠杆菌MDS42细胞。ND表示未检测到斑块。柱表示均值。误差条表示n = 3次独立实验的标准差。
权利和权限
根据与作者或其他权利持有人签订的出版协议,自然或其许可方(例如,社会或其他合作伙伴)对本文拥有排他性权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。
关于本文
引用本文
Nyerges, A., Vinke, S., Flynn, R.。et al。交换的遗传密码可以防止病毒感染和基因转移。自然(2023)。https://doi.org/10.1038/s41586-023-05824-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41586-023-05824-z
