文摘
DNA病毒产生重大影响细胞生物的生态和进化1,2,3,4,但他们的整体多样性和进化轨迹仍然是难以捉摸的5。这里我们进行了phylogeny-guided genome-resolved宏基因组阳光海洋调查,发现plankton-infecting亲戚的疱疹病毒形成一个公认的新门被称为Mirusviricota。这个大的病毒粒子形态发生模块单元进化枝是典型的病毒的领域Duplodnaviria6,多个组件与animal-infecting强烈表明共同祖先Herpesvirales。mirusvirus相当大一部分基因,包括转录机械标志基因缺失在疱疹病毒,是密切相关的同系物巨大的真核生物DNA病毒病毒从另一个领域,Varidnaviria。这些令人瞩目的空想的属性连接Mirusviricota疱疹病毒和巨大的真核病毒是由超过100个环保mirusvirus基因组,包括432碱基的几乎完全连续的基因组。此外,mirusviruses最丰富和活跃的真核病毒特征在阳光照射的海洋,编码多样化功能微生物的感染期间使用真核生物从南极到北极。流行、功能活动,多样化和非典型的空想的属性mirusviruses指向一个持久的作用Mirusviricota在海洋生态系统的生态和真核生物DNA病毒的进化。
主要
大多数双链DNA病毒分为两个主要领域:Duplodnaviria和Varidnaviria。Duplodnaviria包括跟踪噬菌体及相关热点病毒类的Caudoviricetes以及真核病毒的秩序Herpesvirales。Varidnaviria包括大型和巨型门的真核生物DNA病毒Nucleocytoviricota以及更小的与无尾的二十面体病毒衣壳6。这两个领域的基础上,建立了异源的病毒粒子形态发生基因(病毒粒子模块),包括编码结构无关的主要衣壳蛋白(兆赫)“双果冻卷”和HK97折叠Varidnaviria和Duplodnaviria分别6。这两个领域都代表了所有的生活领域,与各自的祖先认为追溯到最后的万能细胞祖先7。
在Duplodnaviria、细菌和古细菌的成员Caudoviricetes基因组大小的展览一个连续范围,从大约10个碱基(kb) > 700 kb,而疱疹病毒,局限于动物宿主,更均匀的基因组范围100 - 300 kb。从噬菌体疱疹病毒可能演变,但缺乏相关的病毒在动物王国关于其确切的进化轨迹提出了疑问5。的成员Varidnaviria也表现出广泛的基因组大小,从大约10 kb > 2 Mb,但是有一个不连续的大型和巨型病毒之间的复杂性Nucleocytoviricota门和其他与基因组varidnaviruses < 50 kb。有人建议Nucleocytoviricota从一个小varidnavirus的祖先进化而来8,9,10,但多个信息的复杂性必然要收购基因(信息模块)还有待充分理解。
病毒在Caudoviricetes和Nucleocytoviricota普遍存在在阳光海洋发挥重要作用在调节浮游生物的群落组成和盛开的活动吗11,12,13,14,15,16,17。这里我们进行了genome-resolved浮游DNA病毒宏基因组的调查指导下一个标志基因的发展史。调查涵盖了近3000亿元基因组读取从海洋表面的样品塔拉海洋探险18,19,20.。我们和手动策划数百人口基因组特征,扩大已知的多样性Nucleocytoviricota。然而,最值得注意的是,我们的调查发现了plankton-infecting亲戚的疱疹病毒形成一个公认的新门我们称为Mirusviricota。mirusviruses分享复杂功能特征和普遍存在在阳光海洋积极感染真核生物,填补一个关键空白浮游生物在生态的理解。尽管一个清晰的疱疹病毒的进化关系,mirusviruses编码更加同系物密切相关的基因Nucleocytoviricota。这些显著的空想的属性Mirusviricota连接两个远亲病毒领域,提供关键的见解真核生物DNA病毒的进化。
海洋真核基因组的病毒
子单元(RNApolA)和RNA聚合酶B (RNApolB)进化的基因标记出现在大多数的已知的海洋微生物真核生物DNA病毒感染9,21直到现在,它只包括Nucleocytoviricota。这里我们进行一个全面的搜索RNApolB基因从极地的透光层,温带和热带海洋使用大型co-assemblies从798基因组(共有2800亿读取产生约1200万重叠群超过2500个核苷酸)19,20.来自于塔拉海洋探险18。这些基因组包含八个浮游生物大小分数从0.8µm 2000µm(补充表1),所有富含微生物真核生物22,23。我们确定了RNApolB基因在这些重叠群使用广谱隐马尔科夫模型(HMM)配置文件,随后建立了2500多个冗余的数据库环境RNApolB蛋白质序列(相似度< 90%;补充表2)。这些序列的系统发育信号不仅重现了相当大的多样性的海洋Nucleocytoviricota24但还透露之前从未描述deep-branching血统明显脱离生活的三个领域和其他已知的病毒(扩展数据图。1)。我们认为,这些新的演化支代表未知的血统的双链DNA病毒。
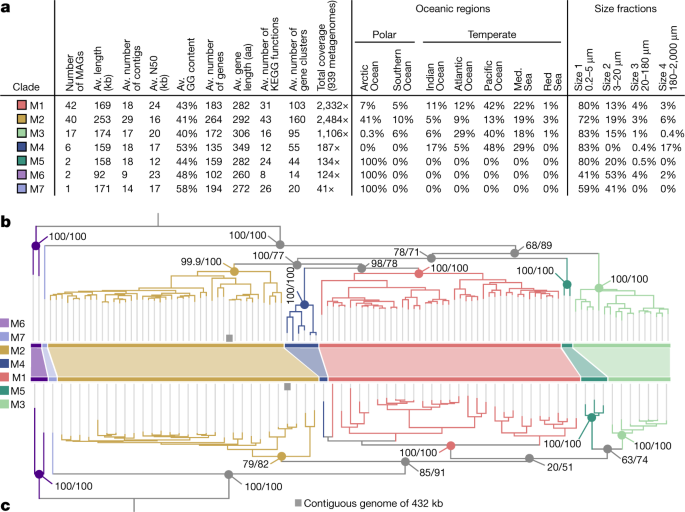
我们进行了一次phylogeny-guided genome-resolved metagenomic RNApolB调查关注的Nucleocytoviricota和新演化支描绘他们的基因组上下文(补充表3)。我们和手动策划581冗余特征Nucleocytoviricotametagenome-assembled基因组(杂志)到1.45 Mb长(平均大约270 kb)和117冗余杂志438 kb的长度(平均约200 kb)的演化支。我们注册的海洋Nucleocytoviricota从先前的宏基因组调查杂志11,12从文化和参考基因组和细胞分类构建一个全面的数据库丰富大型和巨型海洋真核双链DNA病毒(此后称为“全球海洋真核病毒(GOEV)数据库;补充表4)。GOEV数据库包含约060万个基因,并提供上下文信息来识别主要生态和进化的属性包含新杂志RNApolB演化支。
发现的第三个Duplodnaviria门
新组装的Nucleocytoviricota杂志包含大部分的标志基因的病毒门,对应于病毒粒子和信息模块4,5(补充表4)。他们扩大已知的多样性Imitervirales,Pandoravirales,Pimascovirales和Algavirales订单内的类Megaviricetes。此外,一个新的RNApolB演化支暴露一个假定的新Nucleocytoviricota类级组我们称为Proculviricetes,它是由六个杂志专门检测在北极和南部海洋(图。1)。剩下的111杂志新RNApolB演化支也含有相关关键基因进化Nucleocytoviricota信息模块,包括RNApolA和RNApolB、家庭B DNA聚合酶(DNApolB)和转录因子II-S (TFIIS)。这些杂志在一个单基因的发展史的地方(DNApolB)或多个演化支(RNApolA和RNApolB),总是在已知的Nucleocytoviricota订单(扩展数据图。2)。信号TFIIS弱由于其较短的长度。健壮phylogenomic推论连接四个信息基因的标记表明他们代表一个单元病毒进化枝与几个标志基因密切相关,然而,不同于已知的Nucleocytoviricota类(无花果。1)。我们被称为病毒进化枝mirusviruses (mirus是令人惊讶的拉丁词或奇怪的)。
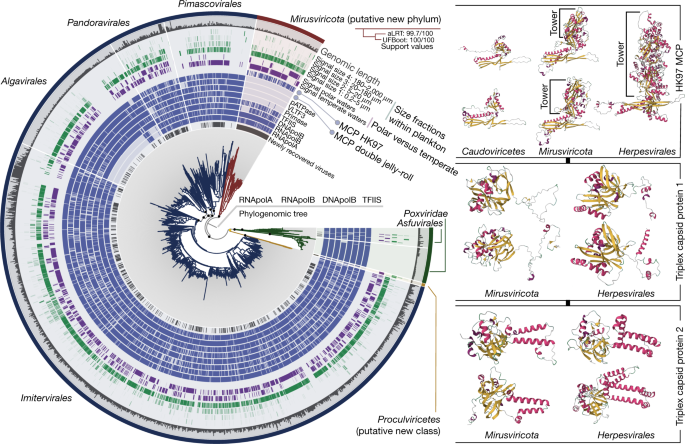
左:最大似然系统发育树构建从GOEV数据库(1722)基因组的基础上策划RNApolA手动连接,RNApolB, DNApolB TFIIS基因(3715氨基酸位置)使用后意味着网站频率混合模型(LG + C30 + F + R10)和扎根mirusviruses和休息。强调了系统发育树中(点)被认为是支持高(近似似然比(aLRT)≥80,超快的引导近似(UFBoot)≥95,黑色)或介质(aLRT≥80或UFBoot≥95,黄色;看到方法)。树上点缀着各色的环互补信息的可视化和anvi传闻。右:3 d结构的预测HK97 MCP的Caudoviricetes,使用AlphaFold2 mirusvirus和疱疹病毒代表获得。蛋白质的二级结构的基础上的属性。专家组还显示三衣壳蛋白的三维结构预测mirusvirus和疱疹病毒代表使用相同的方法。
mirusvirus杂志被组织成七个不同的subclades, M1, M7(从最密集),是M1和M7由41杂志和一个杂志,分别(无花果。2和补充表4)。然而,值得注意的是,他们没有可识别的同系物Nucleocytoviricota病毒粒子模块,包括果冻卷MCP的两倍。相反,注释mirusvirus基因集群使用敏感的序列和结构相似性搜索(方法)发现了一个遥远的同系物HK97-fold兆赫发生在大多数这些杂志(图。1和扩展数据图。3)。这MCP褶皱的存在,只有共享Caudoviricetes和Herpesvirales,表明mirusviruses属于领域Duplodnaviria。与MCP的标识一致,进一步比较嗯概要文件和预测三维(3 d)结构发现关键的剩余组件Duplodnaviria病毒粒子模块,包括terminase (ATPase-nuclease DNA包装机)的关键组件,门户蛋白质衣壳成熟蛋白酶和三层衣壳蛋白1和2(无花果。1,扩展数据图。4和补充表5)。基因编码这些蛋白质的存在mirusviruses建立DNA病毒,他们是真正的大能形成病毒颗粒类似先前已知病毒的领域Duplodnaviria。值得注意的是,系统发育推断的mirusvirus HK97-fold MCP重现了七subclades最初确定的基础上DNApolB, RNApolA, RNApolB TFIIS(无花果。2 b),表明病毒粒子的进化和信息模块。
一个七、基因和环境统计数据Mirusviricotasubclades。Av,平均;aa、氨基酸;KEGG京都基因和基因组的百科全书;将军,所需的最短长度重叠群捕获50%的总装配尺寸;地中海,地中海。b最大似然的系统发育树的构建Mirusviricota杂志的基础上连接四个标志信息基因(RNApolB,这些编码RNApolA DNApolB TFIIS;3715个氨基酸职位)使用LG + F + R7模型。c最大似然的系统发育树的构建Mirusviricota杂志的基础上使用LG MCP(701氨基酸位置)+ R6模型。两树之间的进化枝M6和其他演化支。值在节点代表分支支持(100)计算Shimodaira-Hasegawa-like aLRT(1000复制;左分数)和UFBoot(1000复制;正确的分数)。
广泛的序列差异和长度差异蛋白的病毒粒子mirusviruses之间的模块,疱疹病毒Caudoviricetes(补充表5)阻止有意义的系统发育推断为新领域扩展Duplodnaviria。然而,多个组件的模块提供了重要的见解澄清mirusviruses的进化轨迹。首先,两个三层衣壳蛋白,形成一个heterotrimeric复杂和稳定衣壳壳通过与相邻MCP子单元的交互25在疱疹病毒,但失踪,是守恒的Caudoviricetes。其次,在疱疹病毒兆赫,HK97-fold域,称为层域和负责衣壳壳的形成,是美化的“塔”域项目远离表面的衣壳组装26。塔内域是一个插入的子域核心HK97褶皱26,27。在mirusviruses, MCP蛋白质中还包含一个插入一个子域,虽然大大规模较小(无花果。1无花果和扩展数据。3和4)。这座塔领域迄今为止还没有被描述的任何成员Caudoviricetes,包括所谓的巨型噬菌体(即噬菌体与一个非常大的基因组28)。总的来说,三层衣壳蛋白和MCP塔代表标志特征指向密切mirusviruses之间进化关系和疱疹病毒细菌和古细菌相比,他们的亲戚。
DNApolB基因的系统发育推断使用GOEV数据库和多种真核和额外的病毒血统29日支持的进化距离mirusviruses相对于双链DNA病毒的所有其他已知的演化支(扩展数据图。5)。的单元mirusvirus DNApolB被定位为妹妹进化枝Herpesviridae,两个真核生物的演化支Duplodnaviria大多数真核歹字型构造密切相关,三角洲类型DNApolB序列,在一起形成一个强烈支持进化枝有别于其他病毒的DNApolB。综上所述,病毒粒子之间的相当大的遗传距离mirusviruses模块,Caudoviricetes和Herpesvirales的独特的三维结构mirusvirus MCP(见扩展数据图预测三维结构比较。4)和DNApolB系统发育推断坚定立场mirusviruses在领域内Duplodnaviria外,但前面两个类群的特征Uroviricota(Caudoviricetes),Peploviricota(疱疹病毒),在一个单独的门我们称为Mirusviricota。
Mirusviruses功能复杂
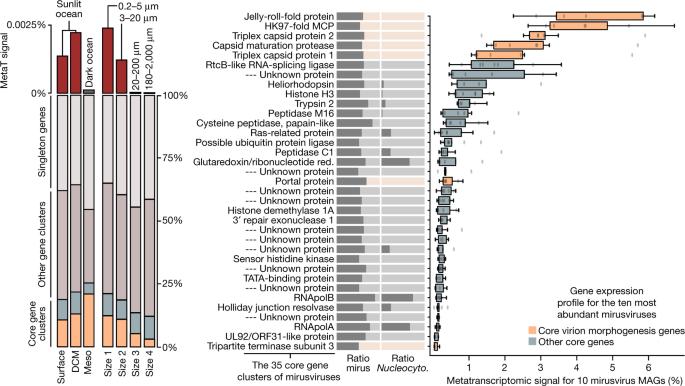
111年Mirusviricota杂志总共包含22242个基因组织成35核心基因簇存在于至少50%的杂志,1825年非核心基因簇,最后9018单例没有近亲在GOEV数据库(补充表6和7)。核心基因集群提供了窗口的关键功能跨subclades共享mirusviruses(补充表8)。除了上述病毒粒子的核心组件和信息模块,它们对应于函数相关的DNA稳定性(H3组蛋白),DNA复制(DNA复制许可因素,glutaredoxin /核苷酸还原酶,霍利迪结游离酶和3′修复核酸外切酶1),转录(tata结合蛋白),基因表达调控(赖氨酸特定组蛋白demethylase 1) RNA的转录后修饰(RtcB-like RNA拼接连接酶)和蛋白质(假定的泛素蛋白连接酶),蛋白质降解(trypsin-like, C1和M16-family肽酶),细胞生长控制(Ras-related蛋白质),外部信号检测传感器组氨酸激酶和感光受体蛋白(heliorhodopsins)。因此,mirusviruses编码一个精心设计的工具包,可以微调的宿主细胞生物学和充满活力的潜在最佳的病毒复制。最后,十个核心基因簇不能分配任何函数序列或蛋白质结构比较的基础上,在参考数据库并等待实验功能描述。
聚类的Mirusviricota从文化(包括杂志和参考病毒基因组Nucleocytoviricota,Herpesvirales和Caudoviricetes基于定量发生基因簇凸显了强大功能区分mirusviruses和疱疹病毒,相反,一个强大的功能相似性mirusviruses和Nucleocytoviricota(扩展数据图。6和补充表9)。因此,function-wise mirusviruses更相似Nucleocytoviricota病毒(其中许多也普遍在海洋的表面;见图。1)相比Herpesvirales。进一步探索的功能景观eukaryote-infecting海洋病毒,我们聚集他们的基因组的基础上定量发生基因集群使用整个GOEV数据库(补充表6和7)。mirusviruses聚集在一起,进一步组织成subclades符合phylogenomic信号(扩展数据图。7)。相比之下,这一分析强调了复杂的功能组成Nucleocytoviricota血统,一些演化支(例如,Imitervirales和Algavirales)分成多个组。除了信息模块的核心组件,基因簇连接的很大一部分Mirusviricota和Nucleocytoviricota参与DNA复制的基因组是由功能:glutaredoxin /核苷酸还原酶,霍利迪结解离,增殖细胞核抗原,dUTPase和DNA拓扑异构酶ⅱ。通常共享功能还包括Ras蛋白,patatin-like磷脂酶(脂质降解),肽酶C1,泛素carboxy-terminal水解酶(蛋白质活动监管)和Evr1 /规律的家庭(胞质成熟的Fe / S蛋白)。因此,两者之间的功能连通性门远远超出信息模块。另一方面,数百个基因簇和功能明显丰富mirusviruses或Nucleocytoviricota(补充表6- - - - - -8两个演化支),暴露出不同的生活方式。核心基因簇中代表的mirusviruses大大减少Nucleocytoviricota基因组包括trypsin-like (mirusviruses基因组的73%和9%Nucleocytoviricota)和M16-family(60%和2%)肽酶,tata结合蛋白质(59%比0%),heliorhodopsin(64%和5%)和组蛋白(54%和2%)。组蛋白的系统发育推断和视紫红质指向一个复杂的这些基因的进化历史Mirusviricota和Nucleocytoviricota,与多个水平转移事件之间的病毒演化支和海洋浮游真核生物(扩展数据图。8)。此外,一个Micromonasheliorhodopsin可能源于一种mirusvirus(扩展数据图。8),这表明Mirusviricota贡献,还有Nucleocytoviricota3,4的进化浮游真核生物的基因流。
Mirusviruses丰富和活跃
据我们所知,Mirusviricota代表第一eukaryote-infecting血统Duplodnaviria发现是广泛和丰富的阳光海洋中的浮游生物。事实上,mirusviruses发现131年的143塔拉海洋站,从南极到北极。他们大多发生在0.2 - 5µm(76.3%的整个mirusvirus宏基因组信号)和3 -µm(15.4%)大小分数覆盖高单细胞浮游真核生物的多样性22(无花果。1和2和补充表10)。在塔拉海洋基因组被认为是在我们的研究中,总的意思是覆盖的海洋Nucleocytoviricota杂志和文化基因在GOEV高出15倍的mirusvirus杂志相比,反映出目前基因组单位这两个门之间的不平衡(1706和111)。然而,平均累积意味着覆盖mirusviruses高于病毒在所有Nucleocytoviricota订单,值得注意的例外Algavirales(扩展数据图。9和补充表10)。因此,mirusviruses是迄今为止最丰富的真核病毒特征在阳光照射的海洋。
mirusviruses不仅丰富,而且是高度活跃的浮游生物。事实上,mirusvirus杂志,仅含有3.8%的基因GOEV,代表13%的塔拉海洋metatranscriptomic信号这个基因组数据库(补充表11)。这大量的原位转录组信号压力的相关性Mirusviricota在海洋系统真核宿主动力学。Mirusviruses在阳光最活跃的海洋(特别是在透光地下层富含叶绿素)比海洋中层的区域(> 200米深度),和细胞内一系列0.2 -20µm(无花果。3),与宏基因组信号。35核心基因簇Mirusviricota代表20%的metatranscriptomic信号(包括七衣壳蛋白为12%),剩余的信号与非核心基因集群(43%)和单例(37%)。因此,高度多样化的基因(近10000确定了单例)似乎发挥重要作用的功能活动Mirusviricota在海洋微生物感染的真核生物。
左:总结整个metatranscriptomic信号的不同的基因类别mirusvirus杂志中塔拉海洋metatranscriptomes。DCM、深叶绿素最大层;内消旋,海洋中层的(上黑暗的海洋层低于200米)。总结35的发生Mirusviricota核心基因簇的比率mirusvirus杂志(mirus)和Nucleocytoviricota(Nucleocyto)。块对应的面板显示框整体metatranscriptomic信号基因对应35核心基因簇中最丰富的mirusviruses和发生在10塔拉海洋基因组。genome-centric百分比值和对应的百分比意味着覆盖(和所有metatranscriptomes)的一个基因在考虑所有基因的累积平均覆盖率(和所有metatranscriptomes)中找到相应的基因组。中心在箱形图显示中位数;框限制表明第25和第75百分位数;胡须延长1.5倍四分位范围从第25和第75百分位数;离群值由点(n= 10分)。红色的。,reductase.
Mirusviruses有不同的生物地理的分布(例如,一些被发现只有在北冰洋),然而他们35核心基因表达水平与印度相似与metatranscriptomic信号样本,表明相对同质的功能性的生活方式无论纬度或subclade(无花果。3和补充表11)。表达的最高水平是在衣壳蛋白基因编码,与比率关键技术的比例相应蛋白质衣壳的疱疹病毒(例如,更多HK97兆赫比三缸或门户蛋白质)。基因编码的新型heliorhodopsin和组蛋白也表达了在高水平,在感染过程中指向一个重要的功能角色。集体、生物地理的和原位转录组mirusviruses模式表明,他们积极地感染丰富海洋单细胞真核生物在温带和极地水域。
Mirusviruses连接两个病毒领域
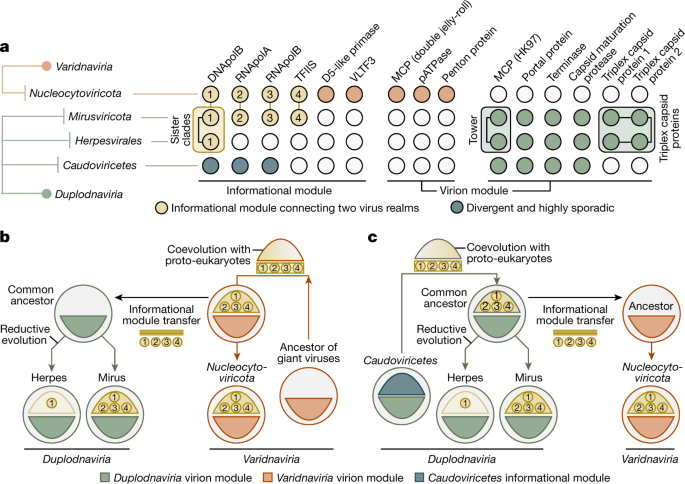
进一步验证mirusviruses的基因组内容和排除人工chimaerism的可能性,我们创建了一个新发现的嗯MirusviricotaMCP和使用它作为诱饵来搜索完整的基因组在额外的数据库中。首先,我们发现只有两个Mirusviricota兆赫全面病毒基因组资源从海洋表面的< 0.2µm大小分数(全球海洋Virome 2)16,这表明大多数病毒粒子进化枝大于0.2µm大小。我们随后筛查MirusviricotaMCP在数据库包含数百元基因组组装的0.2 - 3µm大小比例的海洋表面30.。我们找到了一个连续的Mirusviricota基因组(355个基因)在地中海附属的进化枝平方米431.5 kb的长度,只是比最长的短6 kbMirusviricota杂志(无花果。2 b, c)。其基因概括mirusviruses的核心功能(例如,拓扑异构酶II, tata结合蛋白,组蛋白,多个heliorhodopsins Ras-related gtpase,细胞表面受体,泛素和胰蛋白酶),和80年的这些基因有一个明确的相比Nucleocytoviricota摘要(见方法和扩展数据图。10)。最关键的是,不仅是所有标志基因的信息(DNApolB、RNApolA RNApolB和TFIIS)和病毒粒子(HK97-fold MCP, terminase,门户蛋白质衣壳成熟蛋白酶和两个三层衣壳蛋白)模块Mirusviricota现在也出现相对均匀的基因组(扩展数据图。10)。因此,这几乎完全连续的病毒基因组完全概括特点模块特征只有mirusviruses和疱疹病毒之间的共享,以及模块之间共享信息Mirusviricota和Nucleocytoviricota(无花果。4)。
一方面,mirusviruses属于领域Duplodnaviria病毒粒子的基础上他们的模块。另一方面,他们的标志信息基因同系物盛行的门Nucleocytoviricota意外与高水平的序列相似性。这些结果强烈表明,该信息模块起源于巨型病毒(巨型病毒起源假说;无花果。4 b)或mirusviruses (mirusvirus起源假说;无花果。4摄氏度之间传输),那么他们的两个领域,最有可能的长期共同进化后的相应基因在病毒和proto-eukaryotic主机之间9。因此,mirusviruses不仅是生态系统的组成部分真核浮游生物,但他们也填补关键差距对我们理解的进化轨迹两双链DNA病毒的主要领域。
讨论
我们phylogeny-guided genome-resolved宏基因组的浮游生物调查五大洋和两个海洋表面暴露的主要进化枝大真核生物DNA病毒,与基因组可以达到超过400 kb的长度,这是多样化,流行和积极阳光的海洋。这种进化枝,被称为Mirusviricota,对应于一个领域内公认的新门Duplodnaviria直到现在只包括细菌和archaea-infectingCaudoviricetes和animal-infectingHerpesvirales。的Mirusviricota门被组织成至少7 subclades可能对应于不同的家庭。尽管mirusviruses和Herpesvirales是eukaryote-infecting duplodnaviruses,他们表现出非常不同的基因特性。最值得注意的是,mirusviruses大幅偏离之前所有其他特征组DNA病毒,病毒粒子的形态发生模块(今年双链DNA病毒的定义特征分类)附属领域Duplodnaviria和信息模块密切相关领域内的大型和巨型病毒Varidnaviria。这些明显的空想的属性戒律几乎完全连续的基因组的431.5 kb。的发现Mirusviricota提醒我们,我们尚未掌握完整的生态和进化的复杂性甚至最丰富的双链DNA病毒等关键生态系统在我们的海洋和海洋的表面。
Mirusviruses相对丰富的不同部位的阳光照射的海洋,他们积极感染真核浮游生物小于20µm大小和表达多种功能。Mirusviricota有凝聚力和复杂的生活方式,包括独特的特性(许多核心基因被发现,只有在本门)也大幅重叠与大型和巨型真核varidnaviruses吗11,12。这些共享功能远远超出信息模块,包括生态系统——和寄主专一性的基因,这可能是两组之间的横向转移从共享主机获得的病毒或收敛在进化过程中在不同的时间点。例如,patatin-like磷脂酶之间共享的两个门已经建议促进运输Nucleocytoviricota基因组的细胞质和细胞核31日。功能丰富的mirusviruses相比Nucleocytoviricota包括系统不同的H3组蛋白(蛋白参与染色质在真核细胞内形成32)和heliorhodopsins(感光受体蛋白质可以用作质子通道由巨型病毒在感染33)。在一起,生物地理的模式、功能基因体验和metatranscriptomic信号表明mirusviruses影响关键的生态海洋真核生物使用以前被忽视的生活方式。
病毒的Herpesvirales和Nucleocytoviricota属于两个古老的病毒血统,Duplodnaviria和Varidnaviria分别与相应的祖先可能前发过去万能细胞祖先6,7。然而,确切的进化轨迹和各自的身份最近共同祖先的著名eukaryote-infecting双链DNA病毒演化支仍然难以捉摸,部分是由于缺乏中间状态。尤其令人费解的差距无处不在Caudoviricetes,其中一些竞争对手Nucleocytoviricota功能的复杂性和丰富性体验他们的基因34,35,36,Herpesvirales局限于动物的主机和均匀核复制转录机械与实践缺乏。的识别Mirusviricota扩大duplodnaviruses超越动物真核浮游生物的存在主机,强烈建议他们古老的真核生物。塔的存在和位置域结合的保护两个三层衣壳蛋白(这些存在于已知Caudoviricetes)在两个Mirusviricota和Herpesvirales(见图。1)强烈建议这些真核病毒的共同祖先,而不是从截然不同的独立进化Caudoviricetes演化支。deep-branching定位mirusvirus信息基因证明一个或多个古老的转移(图。1和扩展数据图。2)和关闭之间的相似性DNApolB两个真核生物Duplodnaviria演化支相比于其他DNA病毒(扩展数据图。5)提供补充信息。与塔的短尺寸域和考虑到后来出现的动物和单细胞真核生物相比,Mirusviricota病毒可能更像是真核duplodnaviruses的原始状态。因此,mirusviruses浮游祖先为疱疹病毒,这将发生了还原进化,尤其是失去转录机械,和专门的动物细胞的感染37。
同样神秘的是大型和巨型之间进化海沟Nucleocytoviricota基因组和相对简单的varidnaviruses温和病毒粒子的形成和基因组复制基因体验(这些感染细菌和古菌以及virophageAdenoviridae,否则yaraviruses polintoviruses38,39)。推测,这些噬菌体之间的简单varidnaviruses可能代表进化中间体和真核巨型病毒从门Nucleocytoviricota5。内mirusviruses浮游生物基因组的复杂性,他们的核心功能与共享Nucleocytoviricota提供进一步的见解。信息模块,可能还有其他功能,可能被转移Nucleocytoviricota的祖先mirusviruses(巨型病毒起源假说),导致真核duplodnaviruses的复杂性。在这种情况下,一个Nucleocytoviricota病毒可能交换其病毒粒子模块与一个无特征duplodnavirus合并相同的主机,同时保留详细信息模块。然而,我们的数据不排除转会的同样发人深省的可能性的信息模块从mirusvirus更简单的祖先Nucleocytoviricota(mirusvirus起源假说)。这种情况可能有助于解释进化飞跃从“小”varidnaviruses绝大多数复杂Nucleocytoviricota。无论假设考虑,mirusviruses阐明真核双链DNA病毒的进化轨迹从两个领域。
总的来说,流行、功能复杂性和转录活性验证Mirusviricota指向一个突出的角色mirusviruses在海洋生态系统的生态。这种假定的门不仅扩大我们对浮游生物生态学的理解,但它也为病毒进化提供了新的见解。尽管mirusviruses可能先于疱疹病毒的出现,时间表Mirusviricota在浮游生物起源(之前或之后的巨大的真核病毒)尚未阐明。前进,附加功能和基因组特征加上栽培和环境主机识别细胞排序将进一步促进我们的生活方式和评估mirusviruses在海洋的重要性。
方法
塔拉海洋基因组和metatranscriptomes
我们分析了937个基因组,1149 metatranscriptomes塔拉海洋在EBI下项目PRJEB402。补充表1和11报告一般信息(包括读取和环境的元数据)的数量为每个metagenome metatranscriptome。
限制自动装箱和编造
相对应的798基因组大小分数从0.8µm 2毫米以前组织成11“宏基因组集”的基础上他们的地理坐标19,20.。这0.28万亿读取被用作输入11 metagenomic co-assemblies使用热卖40v1.1.1和重叠群头名称简化在生成的汇编输出使用anvi传闻41,42v6.1。Co-assemblies产生7800万重叠群超过1000核苷酸总量的150.7 Gb(参考文献。19,20.)。限制每个co-assembly输出进行自动装箱,只关注1190万重叠群超过2500个核苷酸。简要:传闻异形anvi叠连群使用浪子43v2.6.3使用默认参数来确定一组初始的基因;我们从宏基因组将映射短读叠连群使用BWA v0.7.15 (ref。44;最低的95%)和存储使用samtools BAM的招募读取文件45;传闻异形anvi每个BAM文件估计每个重叠群的覆盖率和检测数据,并结合映射配置文件合并成一个文件数据库为每个宏基因组集。然后我们集群重叠群的自动装箱算法编造46通过约束集群/宏基因组的数量设置为从50到400人根据一组(共2550宏基因组从大约1200万块重叠群)19,20.。
依赖dna的RNApolB基因的多样性
我们使用HMMER47v3.1b2检测基因匹配2550元基因组之间的依赖dna的RNApolB块单一HMM模型的基础上。我们使用CD-HIT48v4.8.1创建非冗余数据库RNApolB基因的氨基酸水平与序列相似性小于90%(最长达到被选为每个集群)。短序列被排除在外。最后,我们包括参考RNApolB氨基酸序列从细菌、古细菌、真核生物和巨型病毒9:序列与MAFFT对齐49v7.464和FFT-NS-i算法使用默认参数和修剪> 50%差距Goalign v0.3.5 (https://www.github.com/evolbioinfo/goalign)。我们使用最佳拟合模型进行了系统发育重建ModelFinder根据贝叶斯信息准则50IQ-TREE +选项51v1.6.2。我们可视化,扎根发展史使用anvi传闻。这棵树使我们能够识别RNApolB对应于已知的类Nucleocytoviricota以及新RNApolB演化支。
Phylogeny-guided genome-resolved宏基因组
RNApolB的每个宏基因组小块包含至少一个感兴趣的基因(见前一节)手动箱使用anvi专门寻找传闻互动界面Nucleocytoviricota和mirusvirus杂志。首先,我们使用HMMER47v3.1b2识别8标志基因(8不同嗯内运行anvi传闻)以及149个额外的同源组织经常发现在参考Nucleocytoviricota病毒9(一个嗯运行在anvi传闻)。接口考虑序列组成,微分覆盖,GC每个重叠群的内容和分类的信号,并显示八个标志作为单独的层以及149额外的直系同源基因组织经常发现在参考Nucleocytoviricota病毒9作为一个额外的层进行指导。在装箱过程中,应用于术语的数量没有限制Nucleocytoviricota核心基因标记,只要信号暗示的出现一个假定的杂志。注意,而一些宏基因组块包含有限数量的杂志,其他包含几十个。最后,我们分别精制的所有Nucleocytoviricota长度和mirusvirus杂志> 50 kb ref的概述。52,并改名为叠连群他们包含根据杂志ID。
创建GOEV数据库
除了Nucleocytoviricota和mirusvirus杂志在我们的研究中,我们包含了海洋Nucleocytoviricota杂志的特点在裁判中使用自动装箱。11(n= 743)和裁判。12(n= 444),部分使用塔拉海洋基因组。我们还包含235参考Nucleocytoviricota基因组特征主要通过培养还在浮游生物细胞分类53。我们决定每组的平均核苷酸的身份Nucleocytoviricota或从伶人mirusvirus杂志使用dnadiff工具包54v4.0b2。杂志被认为是冗余的,当他们平均核苷酸的身份是> 98%(最低对齐> 25%的小杂志每个比较)。手动策划杂志被选来代表一组冗余的杂志。对组织缺乏手动策划杂志,最长的杂志被选中。这种分析提供了非冗余的1593个海洋基因组数据库杂志+ 224参考基因组,名叫GOEV数据库。我们创建了一个叠连群数据库GOEV数据库使用anvi传闻。浪子43是用来识别基因。
内容管理的标志基因
的氨基酸序列数据集RNApolA、RNApolB DNApolB和TFIIS手动策划通过BLASTp对齐(爆炸55v2.10.1)和系统发育重建,如前所述,真核基因标志20.。简单,多个序列的一个标志基因在同一杂志被检查的基础上,他们的立场在相应的单一蛋白系统树生成使用相同的协议如前所述(部分题为依赖dna的RNApolB基因多样性)。基因组的多个序列然后与BLASTp他们最亲密的参考序列,和彼此。重叠的重要身份> 95%(可能对应于最近的重复事件),只有最长序列是守恒的;清晰的分割,相应序列融合和标签进行进一步的检查。最后,RNApolA和RNApolB序列短于200个氨基酸也删除,以及DNApolB序列短于100个氨基酸,和TFIIS序列短于25个氨基酸。这个步骤创建了一个组策划标志基因。
比对,修剪和单一蛋白的进化分析
对于每一个策划的四个标志基因,序列与MAFFT对齐49v7.464和FFT-NS-i算法使用默认参数。超过50%的网站差距减少使用Goalign v0.3.5 (https://www.github.com/evolbioinfo/goalign)。MAFFT L-INS-i算法和阈值70%削减有缺口的网站用于MCP mirusviruses序列,heliorhodopsin和组蛋白序列(ref heliorhodopsin和组蛋白的序列。20.从裁判和额外的组蛋白参考序列。56添加)。IQ-TREE51v1.6.2用于系统发育重建,ModelFinder50+选项来确定最佳拟合模型根据贝叶斯信息准则。支持从1000复制计算Shimodaira-Hasegawa (SH)——如aLRT57和UFBoot58。按照IQ-TREE手册,支持被认为好当SH-like aLRT≥80%, UFBoot≥95%。传闻v7.1 Anvi用于可视化和根的系统发育树。扩展数据图的树。2不包括模糊迭代识别基因组单和连接蛋白的发展史(见描述supermatrix系统发育分析的部分)。对于大型DNApolB分析,Duplodnaviria和Baculoviridae序列的国家生物技术信息中心(NCBI)病毒基因组数据库(https://www.ncbi.nlm.nih.gov/labs/virus/vssi/ /;2022年4月访问),以及真核生物和病毒序列从裁判。29日、收集、对齐和修剪,树重建,用同样的方法描述,除了FFT-NS-i算法用于MAFFT差距Goalign阈值设置为50%。遥远的演化支被反复删除,以及长树枝,系统不提供信息的序列与Treemmer估计59树的基础上,相对长度为0.95。
解决标志基因发生很多次了
我们手动检查所有的重复序列(标志基因检测多次在同一基因组)后仍然管理步骤,在个人的系统发育树(参见前面章节)。首先,重复的基础上被视为公认的污染主要个人(也就是说,不保存在一个进化枝)不一致的位置对应的基因组在其他单一蛋白的树木。假定的污染物很容易识别和移除。第二,我们识别标志基因paralogues封装整个演化支和/或subclades,表明重复事件发生之前的多元化有关病毒演化支。这是明显的Imitervirales,有两个paralogues RNApolB。这些paralogues首次单一蛋白守恒的系统发育推断,但是只有paralogue演化支与最短的分支是守恒的后续分析,从单一蛋白树木一致性检查和连接。最后,我们还发现一个小的进化枝Algavirales病毒包含一个同系物的TFIIS分支冷淡地普通TFIIS类型,表明基因的收购。这些序列并不包括在后续分析。这个步骤创建了一个组策划和duplicate-free标志基因。
Supermatrix GOEV数据库的系统发育分析
串连的四个一致并修剪策划和duplicated-free标志基因(方法如上所述)进行了提高分辨率的系统发育树。四个标志基因的基因组包含只有TFIIS被排除在外。剩余的杂志和参考基因组,失踪序列替换为空白。模棱两可的基因组的基础上,确定主要和孤立的存在(也就是说,不是一个进化枝模式)在单一和连接蛋白树不一致,以及频繁长树枝和不稳定的职位分类抽样推断,被移除。使用IQ-TREE连接重建系统发育树51v1.6.2最佳拟合模型从ModelFinder根据贝叶斯信息准则50+选项。为分析包括整个GOEV数据库,生成的树被用来指导树基于特定频率的系统发育重建后意味着网站频率混合模型60(LG + C30 + F + R10)。连接树的支持从1000复制计算SH-like aLRT57和UFBoot58。按照IQ-TREE手册,支持被认为好当SH-like aLRT≥80%, UFBoot≥95%。传闻v7.1 Anvi用于可视化和根的系统发育树。
分类推理GOEV数据库
我们确定的分类Nucleocytoviricota杂志的系统发育分析结果的基础上,使用的指导参考基因组内GOEV数据库以及以前的文献分类的推论。11,12,21。
生物地理学的GOEV数据库
我们进行了映射的所有基因组计算的平均覆盖率和检测GOEV数据库。短暂,我们使用BWA v0.7.15(至少95%)的身份和FASTA包含杂志1593和224的文件参考基因组招募短期读取所有937个基因组。我们认为杂志被发现在一个给定的过滤器> 25%的长度被读取时尽量减少非特异性阅读招聘61年。招募读取的数量低于这个截止之前设置为0的比例确定垂直覆盖和招募。
Metatranscriptomics GOEV的数据库
我们做了一个映射塔拉海洋metatranscriptomes计算的平均覆盖率和检测基因中发现GOEV数据库。短暂,我们使用BWA v0.7.15(至少95%)的身份和FASTA包含060万个基因招募短期读取的文件从所有937个基因组。
从Orthofinder同源组
直向同源组(og) mirusvirus杂志(n= 111),mirusvirus几乎完全连续的基因组数据库和参考基因组的病毒-宿主(VHDB;其中包括1754Duplodnaviria,184年Varidnaviria和11个非保密基因组)生成的。我们使用Orthofinder62年v2.5.2 (- s diamond_ultra_sens)生成噩。共有26045个噩是生成和og (n= 9631)至少有5个基因组观测被用于集群的基因组。
AGNOSTOS功能聚合推理
AGNOSTOS v。1partitioned protein-coding genes from the GOEV database in groups connected by remote homologies and categorized those groups as members of the known or unknown coding sequence space on the basis of the workflow described previously63年。AGNOSTOS熵产生较低的基因功能组参所示。20.,63年让我们提供功能注释(包含域架构)的一些基因集群使用远程同源性的方法。
识别和建模的mirusvirus MCP
mirusvirus和其他形态形成模块的假定的MCP蛋白质被确定为指导AGNOSTOS结果,使用公开包含v35 HHsearch, PDB70和UniProt / Swiss-Prot病毒蛋白质数据库64年,65年。候选人MCP当时模仿使用AlphaFold2(参考文献。66年,67年)(使用Cobafold v1.4)和RoseTTAFold68年v.1.1.0。生成的3 d模型然后MCP噬菌体结构相比HK97并使用ChimeraX人类巨细胞病毒和可视化69年v.1.4。
功能的推论Nucleocytoviricota基因组
基因从GOEV数据库对VHDB BLASTp-searched70年,RefSeq71年UniRef90 (ref。72年),NCVOGs73年(所有数据库更新到2021年11月版)和NCBI nr数据库(2020年8月)使用钻石74年v2.0.6与截止E值1×10−5。一个最近发表GVOG数据库21也用于注释使用hmmer47v3.2.1搜索的E1×10的价值−3作为一个重要的门槛。此外,KEGG Orthology和功能类别和Eggnog-Mapper分配75年v2.1.5。最后,tRNAscan-SE76年v2.0.7预测7734年图示。
3 d结构的预测Mirusviricota核心基因
相对应的蛋白质Mirusviricota核心基因簇和缺乏功能注释基于序列相似性建模使用如果AlphaFold2 2.3.0(参考文献。66年,67年;- c full_dbs - t 2022-03-12)。大理服务器77年是用来预测其功能的基础上,蛋白质结构比较。
3 d结构的预测Duplodnaviria标志基因病毒粒子模块
病毒粒子模块的基因Duplodnaviria收集从NCBI蛋白质数据库的基础上,他们最初提交的注释。病毒的基因组基因模块代表了病毒的家庭Herpesviridiae,Alloherpesviridae,Ackermannviridae,Autographiviridae,Chaseviridae,Demerecviridae,Drexlerviridae,Herelleviridae,Myoviridae,Podoviridae,Schitoviridae,Siphoviridae,Zobellviridae,Guelinviridae,Rountreeviridae,Salasmaviridae和非保密caudovirus lilyvirus。基因簇Mirusviricota对应于这些病毒粒子模块收集七mirusvirus subclades。如果3 d模型预测使用AlphaFold2 2.3.0(参考文献。66年,67年)(- c full_dbs - t 2022-03-12)和排名第一的结构模型被用于以下分析。
三维结构比较
Foldseek v4.645 (ref。78年)被用来使多个预测蛋白质结构与程序简便的搜索。对齐的TM分数被排列长度计算和规范化。集群的3 d结构DuplodnaviriaMCP传闻程序进行了使用anvi anvi-matrix-to-newick anvi-interactive和手动模式。
从几乎完全基因组领域分配的基因
两个内部嗯数据库创建如下。首先,所有编码序列(CDSs)贴上Nucleocytoviricota被移除的Varidnaviria光盘的数据集(nVHDB = 53776)70年(2022年5月)。这个数据集,塔拉海洋Nucleocytoviricota杂志(都是手动策划)和235参考Nucleocytoviricota基因组是集成。最后一个Nucleocytoviricota蛋白质数据库包含269523信用违约掉期。同样,我们更换了所有Herpesvirales信用违约互换在VHDBDuplodnaviriacd数据集与Herpesvirales2022年4月蛋白质序列从NCBI下载。此外,海洋Caudovirales数据库包括巨型噬菌体环境基因组34,35被集成到Duplodnaviria蛋白质。最后一个Duplodnaviria蛋白质数据库包含748546蛋白质。蛋白质在两个数据库独立集群在30%序列身份(- c 0.4——cov-mode 5),使用Linclust MMseqs79年v13 - 45111。少于三个基因的基因簇被移除,并使用MAFFT其余的基因簇是一致的49v7.487。嗯文件(n= 16689年和57259年Varidnaviria和Duplodnaviria分别使用hmmbuild HMMER3 (ref)创建。80年)v3.2.1。几乎所有的蛋白质Mirusviricota基因组是对两个自定义搜索嗯数据库使用hmmsearch截止E1×10的价值−6。
统计分析
片面的确切概率法(更大的)是用来确定KEGG Orthology功能以及与远程同源基因簇,111年明显富集Mirusviricota杂志与其他相比Nucleocytoviricota在GOEV数据库中,的基础上发生的这些功能和基因集群。P值是纠正在R使用Benjamini-Hochberg过程,和值< 0.05被认为是重要的。
命名的mirus和procul
拉丁文的形容词mirus(奇怪,奇怪)被选中来描述假定的新Duplodnaviria门:Mirusviricota。拉丁文的副词procul(,在距离遥远)被选中来描述的公认的新类Nucleocytoviricota发现从北极和南部海洋:Proculviricetes。
报告总结
进一步研究信息设计是可用的自然投资组合报告总结与这篇文章有关。
数据可用性
我们的研究包括:使用数据库塔拉海洋基因组和metatranscriptomes (https://www.ebi.ac.uk/ena/browser/view/PRJEB402);从门公开海洋杂志Nucleocytoviricota11,12;VHDB (https://www.genome.jp/virushostdb/);RefSeq (https://ftp.ncbi.nlm.nih.gov/refseq/);UniRef90 (https://ftp.ebi.ac.uk/pub/databases/uniprot/uniref/uniref90/);NCVOG (https://ftp.ncbi.nih.gov/pub/wolf/COGs/NCVOG/);与NCBI nr数据库(https://ftp.ncbi.nih.gov/blast/db/)。生成的数据在我们的研究中已经公开https://doi.org/10.6084/m9.figshare.20284713这链接提供:RNApolB基因的重建塔拉海洋总成(连同参考);个人FASTA文件1593冗余的海洋Nucleocytoviricota手动和mirusvirus杂志(包括697年策划杂志从我们调查)和224年的参考Nucleocytoviricota基因组中包含GOEV数据库;传闻重叠群的GOEV anvi数据库;基因和蛋白质中发现GOEV数据库;手动策划标志基因;预测的3 d结构Duplodnaviria病毒粒子模块(包括蛋白质及其排列);的发展史和相关anvi传闻与元数据配置文件数据库;摘要为标志的基因;FASTA文件几乎连续的基因组(SAMEA2619782_METAG_scaffold_2);和补充表1- - - - - -11。源数据本文提供的。
引用
文森特,F。Sheyn U。波拉特,Z。,Schatz, D. & Vardi, A. Visualizing active viral infection reveals diverse cell fates in synchronized algal bloom demise.Proc。《科学。美国118年e2021586118 (2021)。
净重,c . a .海洋病毒——全球生态系统的主要参与者。启Microbiol Nat。https://doi.org/10.1038/nrmicro1750(2007)。
欧文:a . T。彼蒂,A。,Richards, T. A. & Keeling, P. J. Systematic evaluation of horizontal gene transfer between eukaryotes and viruses.Microbiol Nat。7,327 - 336 (2022)。
Moniruzzaman, M。,Weinheimer, A. R., Martinez-Gutierrez, C. A. & Aylward, F. O. Widespread endogenization of giant viruses shapes genomes of green algae.自然https://doi.org/10.1038/s41586 - 020 - 2924 - 2(2020)。
Koonin, e . V。,Dolja, V. V. & Krupovic, M. Origins and evolution of viruses of eukaryotes: the ultimate modularity.病毒学479 - 4802-25 (2015)。
Koonin,大肠诉et al。全球组织和提出megataxonomy病毒的世界。Microbiol。摩尔。杂志。牧师。84年e00061-19 (2020)。
Krupovic, M。,Dolja, V. V. & Koonin, E. V. The LUCA and its complex virome.启Microbiol Nat。18,661 - 670 (2020)。
Krupovic, m & Koonin大肠诉Polintons:真核病毒的温床,转座子和质粒的进化。启Microbiol Nat。13,105 - 115 (2015)。
Guglielmini, J。,哇,a . C。,Krupovic, M。,Forterre, P. & Gaia, M. Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes.Proc。《科学。美国116年,19585 - 19592 (2019)。
哇,a . C。盖亚,M。,Guglielmini, J。,da Cunha, V. & Forterre, P. Phylogeny of theVaridnaviria形态发生模块:一致和不一致与生命之树和病毒分类。前面。Microbiol。121708 (2021)。
舒尔茨,f . et al .巨型病毒通过全球宏基因组多样性和主机的交互。自然https://doi.org/10.1038/s41586 - 020 - 1957 - x(2020)。
Moniruzzaman, M。,Martinez-Gutierrez, C. A., Weinheimer, A. R. & Aylward, F. O. Dynamic genome evolution and complex virocell metabolism of globally-distributed giant viruses.Commun Nat。111710 (2020)。
Endo, h . et al .生物地理学的海洋巨人病毒显示与真核生物和生态功能的相互作用。Nat,生态。另一个星球。4,1639 - 1649 (2020)。
曼:h .噬菌体蓝藻海洋的picophytoplankton。《。牧师。27华裔(2003)。
金子,h . et al .真核病毒成分可以预测在全球海洋碳出口的效率。iScience24102002 (2021)。
Gregory a . c . et al .海洋病毒DNA宏观和microdiversity从南极到北极。细胞177年,1109 - 1123 (2019)。
标签,c . p . et al . Coccolithovirus便利化的碳出口在北大西洋。Microbiol Nat。3,537 - 547 (2018)。
Sunagawa, s . et al。塔拉海洋:对全球海洋生态系统生物学。启Microbiol Nat。https://doi.org/10.1038/s41579 - 020 - 0364 - 5(2020)。
铁锹戳,t . o . et al .异养细菌diazotrophs蓝藻更丰富的比他们的同行在海洋基因组覆盖大部分的阳光。ISME J。https://doi.org/10.1038/s41396 - 021 - 01135 - 1(2021)。
铁锹戳,t . o . et al .功能性曲目收敛的远亲真核浮游生物谱系海洋丰富的阳光。细胞基因组学https://doi.org/10.1016/J.XGEN.2022.100123(2022)。
Aylward, f . O。,Moniruzzaman, M。,Ha, A. D. & Koonin, E. V. A phylogenomic framework for charting the diversity and evolution of giant viruses.公共科学图书馆杂志。19e3001430 (2021)。
•德•巴尔加斯c . et al .真核浮游生物多样性在阳光照射的海洋。科学348年,1261605 (2015)。
Carradec,问:et al。全球海洋真核基因的图谱。Commun Nat。9373 (2018)。
历经甲级,t . et al。丰富的分类单元的Megaviridae超过这些海洋中的细菌和古生菌。微生物环境。33,162 - 171 (2018)。
Okoye m E。,Sexton, G. L., Huang, E., McCaffery, J. M. & Desai, P. Functional analysis of the triplex proteins (VP19C and VP23) of herpes simplex virus type 1.j .性研究。80年,929 - 940 (2006)。
张y . et al .原子结构人类疱疹病毒6 b的衣壳和capsid-associated外皮复合物。Commun Nat。105346 (2019)。
杜达,r . l . & Teschke c . m .神奇的HK97褶皱:多才多艺的适度的差异的结果。咕咕叫。当今。性研究。369 - 16,(2019)。
华,j . et al .衣壳和硕大的噬菌体的基因组的进化达到HK97褶皱。mBio8e01579-17 (2017)。
外籍教练,D。Krupovic, M。,Guglielmini, J。,Forterre, P. & Venclovas, C. S. Diversity and evolution of B-family DNA polymerases.核酸Res。4810142 (2020)。
Paoli l . et al。全球海洋微生物的生物合成的潜力。自然https://doi.org/10.1038/s41586 - 022 - 04862 - 3(2022)。
勒让德,m . et al .多样性和进化的新兴Pandoraviridae家庭。Commun Nat。92285 (2018)。
Talbert, p . B。,Armache, K. J. & Henikoff, S. Viral histones: pickpocket’s prize or primordial progenitor?表观遗传学染色质1521岁(2022年)。
Hososhima, s . et al . Proton-transporting heliorhodopsins从海洋巨大的病毒。Elife11e78416 (2022)。
Weinheimer a r & Aylward f . o .感染策略和生物地理学区分世界性的海洋大型噬菌体组。ISME J。https://doi.org/10.1038/s41396 - 022 - 01214 - x(2022)。
Al-Shayeb, b . et al .演化支巨大的噬菌体来自地球的生态系统。自然578年,425 - 431 (2020)。
Weinheimer A r & Aylward f . o .不同血统的Caudovirales编码一个深深分支multi-subunit RNA聚合酶。Commun Nat。114506 (2020)。
阿德勒B。,Sattler, C. & Adler, H. Herpesviruses and their host cells: a successful liaison.Microbiol趋势。25,229 - 241 (2017)。
Yutin, N。,Shevchenko, S., Kapitonov, V., Krupovic, M. & Koonin, E. V. A novel group of diverse Polinton-like viruses discovered by metagenome analysis.BMC医学杂志。1395 (2015)。
Boratto p v . m . et al . Yaravirus:病毒感染Acanthamoeba castellanii小说80 nm。Proc。《科学。美国117年,16579 - 16586 (2020)。
李,D。,Liu, C. M., Luo, R., Sadakane, K. & Lam, T. W. MEGAHIT: an ultra-fast single-node solution for large and complex metagenomics assembly via succinct de Bruijn graph.生物信息学31日,1674 - 1676 (2014)。
、a . m . et al . Anvi传闻:一种先进的组学数据分析和可视化平台。PeerJ3e1319 (2015)。
社区领导、a . m . et al .,集成的、可再生的和anvi multi-omics传闻。Microbiol Nat。6,3 - 6 (2021)。
凯悦,d . et al .浪子:原核基因识别和翻译起始位点的识别。BMC Bioinform。11119 (2010)。
李,h &杜宾,r .快速和准确的短阅读符合burrows - wheeler变换。生物信息学25,1754 - 1760 (2009)。
李,h . et al .序列比对/地图格式和SAMtools。生物信息学25,2078 - 2079 (2009)。
Alneberg, j . et al .装箱宏基因组叠连群覆盖率和组成。Nat方法。11,1144 - 1146 (2014)。
艾迪,s . r .加速概要嗯搜索。公共科学图书馆第一版。医学杂志。7e1002195 (2011)。
李,w . & Godzik Cd-hit:快速聚类和比较大的组蛋白或核苷酸序列。生物信息学22,1658 - 1659 (2006)。
Katoh, k &史坦利·d·m·MAFFT多重序列比对软件版本7:改善性能和可用性。摩尔。杂志。另一个星球。30.,772 - 780 (2013)。
Kalyaanamoorthy, S。明,问。,Wong, T. K. F., von Haeseler, A. & Jermiin, L. S. ModelFinder: fast model selection for accurate phylogenetic estimates.Nat方法。14,587 - 589 (2017)。
阮,l . T。,Schmidt, H. A., von Haeseler, A. & Minh, B. Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies.摩尔。杂志。另一个星球。32,268 - 274 (2015)。
铁锹戳,t . o . &、a . m .识别污染与先进的可视化和分析实践:宏基因组方法真核基因组装配。PeerJ4e1839 (2016)。
李约瑟,d . m . et al .目标宏基因组复苏的四种不同病毒显示共享和巨大的海洋真核生物病毒的独特特征。费罗斯。反式。r . Soc。B374年,20190086 (2019)。
Delcher, a . L。,Phillippy, A., Carlton, J. & Salzberg, S. L. Fast algorithms for large-scale genome alignment and comparison.核酸Res。30.,2478 - 2483 (2002)。
Altschul, s F。、《W。,Miller, W., Myers, E. W. & Lipman, D. J. Basic local alignment search tool.j·摩尔,杂志。215年,403 - 410 (1990)。
Yoshikawa, g . et al . Medusavirus小说大DNA病毒发现温泉的水。j .性研究。93年e02130-18 (2019)。
Guindon, s . et al。新算法和最大似然估计方法的发展史:评估PhyML 3.0的性能。系统。医学杂志。59,307 - 321 (2010)。
黄平君,d . T。,Chernomor, O., von Haeseler, A., Minh, B. Q. & Vinh, L. S. UFBoot2: improving the ultrafast bootstrap approximation.摩尔。杂志。另一个星球。35,518 - 522 (2018)。
Menardo f . et al . Treemmer:一个工具来减少多样性的大系统数据集以最小的损失。BMC Bioinform。19164 (2018)。
王,h . C。,Minh, B. Q., Susko, E. & Roger, A. J. Modeling site heterogeneity with posterior mean site frequency profiles accelerates accurate phylogenomic estimation.系统。医学杂志。67年,216 - 235 (2018)。
铁锹戳,t . o . et al .固氮Planctomycetes种群和变形菌门是在海洋表面丰富的基因组。Microbiol Nat。3,804 - 813 (2018)。
Emms, d . m . &凯利,s . OrthoFinder:解决基本的偏见在整个基因组的比较可显著改善orthogroup推理精度。基因组医学杂志。16157 (2015)。
万尼,c . et al .统一已知和未知微生物编码序列空间。Elife11e67667 (2022)。
加布勒,f . et al .蛋白质序列分析使用MPI生物信息学工具。咕咕叫。Protoc。Bioinform。72年e108 (2020)。
Steinegger m . et al . HH-suite3快速远程同源性检测和蛋白质注释。BMC Bioinform。20.473 (2019)。
跳投,j . et al .高度精确和AlphaFold蛋白质结构预测。自然596年,583 - 589 (2021)。
米尔迪塔族人,m . et al . ColabFold:使蛋白质折叠。Nat方法。19,679 - 682 (2022)。
门敏,m . et al .准确预测蛋白质结构和交互使用three-track神经网络。科学373年,871 - 876 (2021)。
佩特森工作室内由手工制作完成,e . f . et al . UCSF ChimeraX:结构可视化研究人员、教育工作者,和开发人员。蛋白质科学。30.,70 - 82 (2021)。
历经甲级,t . et al .病毒基因组与主机分类法。病毒866 (2016)。
电台k·D。,Tatusova, T. & Maglott, D. R. NCBI reference sequences (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins.核酸Res。35D61-D65 (2007)。
Suzek b E。王,Y。,Huang, H., McGarvey, P. B. & Wu, C. H. UniRef clusters: a comprehensive and scalable alternative for improving sequence similarity searches.生物信息学31日,926 - 932 (2015)。
Yutin, N。,Wolf, Y. I., Raoult, D. & Koonin, E. V. Eukaryotic large nucleo-cytoplasmic DNA viruses: clusters of orthologous genes and reconstruction of viral genome evolution.性研究。J。6223 (2009)。
Buchfink B。,Xie, C. & Huson, D. H. Fast and sensitive protein alignment using DIAMOND.Nat方法。1259-60 (2015)。
Huerta-Cepas, j .等人蛋5.0:层次结构、功能和系统注释orthology资源基于5090生物和2502病毒。核酸Res。47D309-D314 (2019)。
劳,t . m . &艾迪,s . r . tRNAscan-SE:一个程序来提高检测转移核糖核酸基因在基因组序列。核酸Res。25,955 - 964 (1997)。
河中沙洲,l . & Rosenstrom, p .大理服务器:保护映射在3 d。核酸Res。38W545 (2010)。
van Kempen m . et al .快速和精确的蛋白质结构与Foldseek搜索。预印在bioRxivhttps://doi.org/10.1101/2022.02.07.479398(2022)。
豪泽,M。,Steinegger, M. & Söding, J. MMseqs software suite for fast and deep clustering and searching of large protein sequence sets.生物信息学32,1323 - 1330 (2016)。
芬恩,r D。,Clements, J. & Eddy, S. R. HMMER web server: interactive sequence similarity searching.核酸Res。39W29-W37 (2011)。
确认
我们的调查是通过两个科学努力:抽样和测序的努力塔拉海洋项目,提供的生物信息学和可视化功能anvi魄(https://anvio.org/)。我们感谢所有谁促成了这些努力,以及其他开源的生物信息学工具的透明度和开放的承诺。塔拉海洋(包括塔拉海洋和塔拉海洋极地圈探险)不存在没有的领导塔拉海洋基金会和持续支持23个学院(https://oceans.taraexpeditions.org/)。我们也承认国家的承诺和Genoscope / CEA。的一些计算进行了使用装饰用锌铜合金,titane和居里高性能计算机通过GENCI提供资助(t2011076389、t2012076389 t2013036389, t2014036389, t2015036389和t2016036389)。本研究支持部分由法国GENOMIQUE (ANR-10-INBS-09),日本社会科学促进KAKENHI (18 h02279 h00384 22日),全球可持续性发展研究中心,京都大学研究协调联盟的国际合作研究项目化学研究所,京都大学(2022 - 26日2021 - 29和2020 - 28)。表示抗议的赠款支持跟国家de la矫揉造作的(ANR - 20 - ce20 - 0009 - 02年和ANR - 21 - ce11 - 0001 - 01), M.G.支持ANR ALGALVIRUS ANR - 17 - ce02 - 0012,和T.O.D.支持ANR氢ANR - 14 - ce23 - 0001。计算工作的一部分是由超级计算机系统,化学研究所,京都大学。本文的贡献141号塔拉海洋。
作者信息
作者和联系
贡献
T.O.D.进行这项研究,这是与M.G.和功率因数M.G.发起,L.M.纳瑞雅南,简历,E。P。和T.O.D. carried out the primary data analysis. T.O.D. completed the genome-resolved metagenomic analysis. M.G. and T.O.D. curated the marker genes and identified the biological duplicates. M.G. carried out phylogenetic and phylogenomic analyses. L.M. carried out functional analyses, gene comparisons and protein structure predictions with the supervision of H.O. C.V. produced gene clusters with remote homologies with the supervision of A.F.-G. M.K. identified the MCP ofMirusviricota和其他病毒粒子模块的关键基因。密纹唱片进行比较基因组,生物地理的和metatranscriptomic分析。所有作者的贡献来解释数据和写作手稿。
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
同行评审
同行审查的信息
自然谢谢弗兰克•Aylward k . Eric Wommack和其他匿名的,审稿人(s)为他们的贡献的同行评审工作。同行审查报告是可用的。
额外的信息
出版商的注意施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
扩展数据图1小说B RNA聚合酶的识别(RNApolB)演化支在阳光照射的海洋。
最大似然系谱树(LG + F + R10模型,906网站)是基于2728年RNApolB长度超过800个氨基酸序列相似度< 90%(内圈)灰色识别来自11个大型海洋metagenomic co-assemblies。这种分析还包括262年参考RNApolB序列内圈(红色)对应于古细菌,细菌、真核和巨型病毒血统为视角。中间环显示RNApolB序列的数量从11元基因组co-assemblies选中的氨基酸序列与身份相匹配(log10) > 90%。外环显示选择的不同的演化支。最后,RNApolB贴上新的血统的红点mirusviruses (subclades特征在后续分析)和蓝色的Proculviricetes。
扩展数据图3三维结构的主要衣壳蛋白(MCP)。
图显示了MCP的3 d结构埃希氏杆菌属噬菌体HK97 (Caudoviricetesmirusviruses),一个代表基因组(估计使用Alphafold),和人类巨细胞病毒(Herpesvirales)。PDB加入数字HK97和巨细胞病毒兆赫在括号中表示。
扩展数据图4蛋白质序列和建立三维结构比较。
面板显示蛋白质序列和三维结构比较(Blastp和Foldseek) HK97 MCP从三个主要覆盖各种家庭的代表Duplodnaviria演化支。中心行箱线图显示,中位数;框限制表明第25和第75百分位数;胡须延长1.5倍四分位范围从第25和第75百分位数;离群值由点(从上到下,n = 22日,50岁,38岁,25岁,35岁,16岁,40岁,23和117个独立的比较)。校准值的范围从最低9氨基酸最多1437个氨基酸。至少bitscore值的范围从19.6到2577。的Foldseek TMscore值范围从最低为0.09至0.997最多。生成系统树图使用欧式距离和病房内anvi传闻,是基于Foldseek TMscore值。面板B描述了建立三维结构的选择HK97 MCP和三层蛋白质的三个主要的代表Duplodnaviria演化支(Caudoviricetes病毒缺乏三衣壳蛋白)。蛋白质是彩色基于二级结构属性。
扩展数据图5 DNApolB标志基因的发展史。
图显示一个最大似然系谱树(847个网站,1475序列)的dna聚合酶b族序列使用LG + F + R10模型(由ModelFinder +选择)从所述数据库,Duplodnaviria和Baculoviridae从NCBI病毒基因组序列数据库,并从外籍教练等真核生物和病毒序列。29日(见方法)。真核山字形和相关演化支作为外群。系统支持被认为是高(aLRT UFBoot > = 95 > = 80和黑色),中等(aLRT > = 80或UFBoot > = 95,在黄色)或低(aLRT < 80和UFBoot < 95年红色)(见方法)。Baculo: Baculoviridae;Caudo: Caudoviricetes;Nudi: Nudiviridae。
扩展数据图6 mirusviruses和参考病毒基因组的功能集群文化。
内部的树是一个聚类的Mirusviricota”和其他基因组的基础上发生的所有基因集群(OrthoFinder方法,Bray-Curtis距离)。
扩展数据图7功能丰富的集群和海洋病毒在mirusviruses和广泛Nucleocytoviricota。
在面板,内树Mirusviricota和聚类Nucleocytoviricota基因组> 100 kbp长度的基础上发生的所有单体基因集群(欧氏距离),扎根的Chordopoxvirinae亚科的痘病毒基因组。戒指的信息显示的主要分类Nucleocytoviricota以及发生的60基因簇中发现至少50%的Mirusviricota”或Nucleocytoviricota。60基因集群是集群基于他们的整个基因组发生(没有/存在)。面板B显示了发生的基因簇的已知函数包含了检测到至少50%的“Mirusviricota”或Nucleocytoviricota基因组。
扩展数据图8 Mirusviruses包含新的组蛋白和heliorhodopsins系统发育演化支。
图显示两个面板。左面板显示最大似然系谱树的组蛋白发生GOEV数据库在真核杂志,根植与H4(遥远的真核进化枝)(266序列;180网站)和基于LG + R8模型。各种真核生物的演化支远离H2-H3-H4被关注更为克制病毒信号。一圈提供了额外的分类信息。底部面板总结的比例从不同的病毒基因组演化支含有组蛋白。系统支持被认为是高(aLRT UFBoot > = 95 > = 80和黑色),中等(aLRT > = 80或UFBoot > = 95,在黄色)或低(aLRT < 80和UFBoot < 95年红色)(见方法)。右面板显示最大似然heliorhodopsins发生的种系发生树GOEV数据库和真核杂志(280序列;313网站),扎根大进化枝浓缩在真核生物和基于v + F + R8模型。一圈提供了额外的分类信息。 Bottom panel summarizes the proportion of genomes from different viral clades containing heliorhodopsins. Phylogenetic supports were considered high (aLRT>=80 and UFBoot>=95, in black), medium (aLRT>=80 or UFBoot>=95, in yellow) or low (aLRT<80 and UFBoot<95, in red) (see Methods).
扩展数据图9环境信号的病毒真核生物的演化支在阳光照射的海洋。
对于每个海洋真核病毒演化支,箱形图显示累计平均覆盖率GOEV基因组937人之上塔拉海洋基因组。只有基因组中发现至少一个metagenome被认为是。中心行箱线图显示,中位数;框限制表明第25和第75百分位数;胡须延长1.5倍四分位范围从第25和第75百分位数;离群值表示为点。的平均覆盖率值范围从最低0.35倍上限6273.1倍。认为是基因组的数量每进化枝及其累积覆盖值也进行了描述。
扩展数据图10几乎完全基因组的Mirusviricota”。
355个基因的同线性mirusvirus几乎完全连续的基因组信息突出标志基因的发生,病毒粒子模块,以及heliorhodopsins和组蛋白。基因与摘要的打击Duplodnaviria或Varidnaviria分别标记为绿色和红色(内部树)。
补充信息
补充表1
937年的描述塔拉海洋基因组。
补充表2
从阳光海洋依赖dna的RNApolB基因特征。
补充表3
Metabins包含依赖dna的RNApolB感兴趣的基因,用于genome-resolved宏基因组。
补充表4
基因和环境统计数据GOEV数据库。
补充表5
蛋白质序列和三维结构比较标志基因的病毒粒子模块Caudoviricetes,mirusviruses和疱疹病毒。表包括BLASTp和Foldseek结果总结为每个标志基因。
补充表6
发生与远程同源基因簇的GOEV数据库。包括一个核心基因簇的总结。基因簇的浓缩Mirusviricota评估使用确切概率法(片面的,更大的)和使用Benjamini-Hochberg方法调整。
补充表7
基因之间的联系,与远程同源的基因组和基因簇GOEV数据库。
补充表8
基因的功能注释GOEV数据库。的浓缩KEGG直接同源Mirusviricota评估使用确切概率法(片面的,更大的)和使用Benjamini-Hochberg方法调整。
补充表9
发生与远程同源基因簇mirusviruses和参考基因组的文化,包括疱疹病毒。基因mirusviruses之间共享和疱疹病毒进行了总结。
补充表10
生物地理的信号GOEV数据库。表包括宏基因组的元数据和平均覆盖整个基因组的基因组,
补充表11
原位GOEV数据库基于1140的基因表达塔拉海洋metatranscriptomes。表包括metatranscriptomics元数据,意味着所有metatranscriptomes mirusvirus基因的报道,和个人基因和核基因表达水平时,考虑到10个最丰富的mirusviruses。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
盖亚,M。,Meng, L., Pelletier, E.et al。Mirusviruses链接巨型病毒疱疹病毒。自然616年,783 - 789 (2023)。https://doi.org/10.1038/s41586 - 023 - 05962 - 4
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586 - 023 - 05962 - 4