摘要
胞嘧啶碱基编辑器(CBEs)能有效地产生精确的C·g到t·A碱基转换,但激活诱导的胞嘧啶脱氨酶/载脂蛋白B mrna编辑酶催化多肽样蛋白(AID/APOBEC)家族脱氨酶组分会诱导相当大的脱靶效应和吲哚。为了探索非自然胞嘧啶脱氨酶,我们重新利用腺嘌呤脱氨酶TadA-8e进行胞嘧啶转化。在TadA-8e中引入N46L变体消除了其腺嘌呤脱氨酶活性,从而产生了TadA-8e衍生的C- g碱基编辑器(Td-CGBE),能够高效和精确地进行C·g - g·C编辑。通过与尿嘧啶糖基化酶抑制剂的融合和进一步引入其他变体,我们获得了一系列td - cbe,它们要么具有与BE4max相似的高活性,要么与其他报道的精确cbe相比具有更高的精密度。Td-CGBE/ td - cbe显示非常低的indel效应和背景水平的cas9依赖或cas9独立的DNA/RNA脱靶编辑。此外,Td-CGBE/ td - cbe在细胞或小鼠胚胎中均聚胞嘧啶位点产生精确编辑时效率更高,表明其在基因治疗和其他应用中的准确性和安全性。
这是订阅内容的预览,通过你的机构获取
访问选项
订阅Nature+
立即在线访问《自然》和其他55种《自然》杂志
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
99.00美元
每期仅需8.25美元
所有价格均为净价格。
增值税将在稍后的结帐中添加。
税金计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问权限。
32.00美元
所有价格均为净价格。


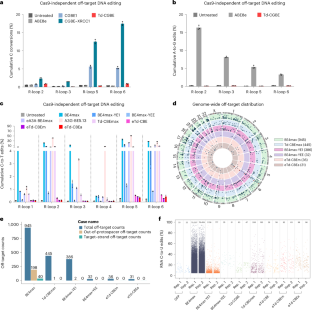

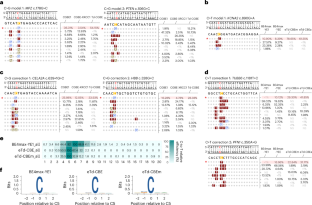
数据可用性
HTS数据已存放在NCBI序列读取档案数据库下的登录代码PRJNA822038,PRJNA871961,PRJNA855334,PRJNA835691, PRJNA835701和PRJNA882574(参考文献。47,48,49,50,51,52).RNA-seq数据已存放在NCBI序列读取档案数据库中,登录代码为PRJNA871962和PRJNA830998(参考文献。53,54).对数据可用性没有限制。源数据都提供了这张纸。
代码的可用性
Detect-seq数据的相关分析代码存放在GitHub (https://github.com/menghaowei/Detect-seq)55.
参考文献
碱基编辑:活细胞基因组和转录组的精确化学。Nat. Rev. Genet。19, 770-788(2018)。
科莫a.c.,金玉波,帕克,M. S, Zuris, J. a .和刘德仁。基因组DNA中无双链DNA切割的目标碱基的可编程编辑。自然533, 420-424(2016)。
高德利,n.m.等人。基因组DNA中A·t到g·C的可编程碱基编辑自然551, 464-471(2017)。
王磊等。通过共同表达游离尿嘧啶DNA糖基化酶抑制剂增强碱基编辑。细胞Res。27, 1289-1292(2017)。
库尔特,i.c等人。CRISPR C-to-G碱基编辑器用于诱导人类细胞中的靶向DNA转位。生物科技Nat。》。39, 41-46(2021)。
赵,D.等。糖基化酶碱基编辑器可以实现C-to-A和C-to-G碱基的改变。生物科技Nat。》。39, 35-40(2021)。
柯布兰,L. W.等。高效的C·G-to-G·C基编辑器使用CRISPRi屏幕、目标库分析和机器学习开发。生物科技Nat。》。39, 1414-1425(2021)。
陈,L.等。可编程C:G到G:C基因组编辑与crispr - cas9导向的碱基切除修复蛋白。Commun Nat。12, 1384(2021)。
左,e,等。胞嘧啶碱基编辑器在小鼠胚胎中产生大量脱靶单核苷酸变异。科学364, 289-292(2019)。
Jin, S.等。胞嘧啶,而不是腺嘌呤,碱基编辑在水稻中诱导全基因组脱靶突变。科学364, 292-295(2019)。
周,C.等。DNA碱基编辑诱导的脱靶RNA突变及其突变消除。自然571, 275-278(2019)。
Grunewald, J.等。由crispr引导的DNA碱基编辑器诱导的转录组范围的脱靶RNA编辑。自然569, 433-437(2019)。
Gehrke, J. M.等。一个APOBEC3A-Cas9碱基编辑器,最小化旁观者和脱靶活动。生物科技Nat。》。36, 977-982(2018)。
Kim, Y. B.等。用工程cas9 -胞苷脱氨酶融合增加基因组靶向范围和碱基编辑的精度。生物科技Nat。》。35, 371-376(2017)。
金H. S,郑Y. K, Hur, J. K, Kim J. S,裴s腺嘌呤碱基编辑器催化细胞内胞嘧啶转化。生物科技Nat。》。37, 1145-1148(2019)。
Grunewald, J.等。减少RNA脱靶和自编辑活性的CRISPR DNA碱基编辑器。生物科技Nat。》。37, 1041-1048(2019)。
陈,L.等。工程精确的腺嘌呤碱基编辑器与无限小的旁观者突变率和脱靶编辑。Nat,化学。医学杂志。https://doi.org/10.1038/s41589-022-01163-8(2022)。
Lapinaite, A.等。通过crispr - cas9引导的腺嘌呤碱基编辑器捕获DNA。科学369, 566-571(2020)。
郑玉奎等。腺嘌呤碱基编辑工程减少了旁观者胞嘧啶的编辑。生物科技Nat。》。39, 1426-1433(2021)。
谭建军,张芳,Karcher, D.和Bock, R.用于位点特异性单核苷酸替换的高精度碱基编辑器工程。Commun Nat。10439(2019)。
Thuronyi, B. W.等。不断发展的基础编辑器与扩大的目标兼容性和改进的活动。生物科技Nat。》。37, 1070-1079(2019)。
西马苏,H.等。扩展靶向空间的CRISPR-Cas9核酸酶工程。科学361, 1259-1262(2018)。
张旭,等。通过融合单链dna结合蛋白结构域,提高胞苷碱基编辑器的效率和靶向范围。Nat细胞生物学。22, 740-750(2020)。
李,S.等。使用工程化的APOBEC3G-nCas9碱基编辑器进行单次c -t替换,使基因组和转录组范围的脱靶效应最小。科学。睡觉。6, eaba1773(2020)。
Bae, S., Park, J.和Kim, J. S. Cas-OFFinder:一种快速和多功能的算法,用于搜索Cas9 rna引导内切酶的潜在脱靶位点。生物信息学30., 1473-1475(2014)。
Doman J. L., Raguram, A., Newby, G. A.和Liu D. R.胞嘧啶碱基编辑器对cas9独立脱靶DNA编辑的评估和最小化。生物科技Nat。》。38, 620-628(2020)。
王磊等。消除碱基编辑器诱导的全基因组和转录组脱靶突变。Nat细胞生物学。23, 552-563(2021)。
Lei, z,等。Detect-seq揭示了胞嘧啶碱基编辑器的原间隔外编辑和目标链编辑。Nat方法。18, 643-651(2021)。
Lei, z,等。线粒体碱基编辑器诱导大量核脱靶突变。自然606, 804-811(2022)。
袁,T.等。机器学习方法可预测序列上下文偏好的C-to-G碱基编辑器优化。Commun Nat。12, 4902(2021)。
Auer-Grumbach, M., Strasser-Fuchs, S., Robl, T., Windpassinger, C. & Wagner, K.由两种新的突变引起的迟发性夏-玛丽-图斯2综合征合成基因。神经学61, 1435-1437(2003)。
罗德里格斯-埃斯库德罗等。综合功能分析PTEN突变:在肿瘤和自闭症相关综合征中的意义。嗡嗡声。摩尔,麝猫。20., 4132-4142(2011)。
sybe, S.等人。功能的新生丧失或获得突变KCNA2引起癫痫性脑病。Nat,麝猫。47, 393-399(2015)。
埃斯特加玛特等人。CELA2A突变易导致早发性动脉粥样硬化和代谢综合征,并影响血浆胰岛素和血小板激活。Nat,麝猫。51, 1233-1243(2019)。
Ropero, P.等人。Hb Johnstown [β 109 (G11) Val - >Leu]:第2例描述并首次与β相关0-两个西班牙家庭的地中海贫血。点。j .内科杂志。65, 298-301(2000)。
法泽利,w。一个TUBB6突变与常染色体显性非进展性先天性面瘫、双侧上睑下垂和腭咽功能障碍有关。嗡嗡声。摩尔,麝猫。26, 4055-4066(2017)。
吴昌华等。profilin 1基因突变导致家族性肌萎缩性侧索硬化症。自然488地球物理学报,1999 - 503(2012)。
阿巴布等。基于目标库分析和机器学习的碱基编辑结果的决定因素。细胞182, 463-480(2020)。
Walton, R. T, Christie, K. A, Whittaker, M. N. & kleinstver, B. P.无约束基因组靶向近无pam工程CRISPR-Cas9变体。科学368, 290-296(2020)。
李,J.等。最小化RNA脱靶活性的腺嘌呤碱基编辑器的结构引导工程。Commun Nat。12, 2287(2021)。
Shi, K.等。APOBEC3A和APOBEC3B靶向DNA胞嘧啶脱氨基和诱变的结构基础。Nat。结构。摩尔。杂志。24, 131-139(2017)。
张旭,等。双碱编辑器催化细胞内胞嘧啶和腺嘌呤碱的转化。生物科技Nat。》。38, 856-860(2020)。
Dobin, A.等。STAR:超快通用RNA-seq对准器。生物信息学29, 15-21(2013)。
克鲁格,S.等。感觉运动性多神经病变和系统性淀粉样变性是乳腺类癌样分化良好癌的副肿瘤症状。Dtsch。地中海,Wochenschr。123, 179-184(1998)。
李,D.等。利用CRISPR-Cas系统在小鼠和大鼠中进行可遗传基因靶向。生物科技Nat。》。31, 681-683(2013)。
黄,G. h等。基于web的CRISPR碱基编辑设计和分析工具。BMC生物信息学19, 542(2018)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA822038https://www.ncbi.nlm.nih.gov/bioproject/PRJNA822038(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA871961https://www.ncbi.nlm.nih.gov/bioproject/PRJNA871961(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA855334https://www.ncbi.nlm.nih.gov/bioproject/PRJNA855334(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA835691https://www.ncbi.nlm.nih.gov/bioproject/PRJNA835691(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA835701https://www.ncbi.nlm.nih.gov/bioproject/PRJNA835701(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA882574https://www.ncbi.nlm.nih.gov/bioproject/PRJNA882574(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA871962https://www.ncbi.nlm.nih.gov/bioproject/PRJNA871962(2022)。
陈,L.等。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。NCBI SRA,生物项目PRJNA830998https://www.ncbi.nlm.nih.gov/bioproject/PRJNA830998(2022)。
陈,L.等。Td-BE-Detect-seq分析。GitHubhttps://github.com/menghaowei/Detect-seq(2022)。
确认
感谢华东师范大学公共创新平台(011)。感谢华东师范大学生命科学学院流式细胞检测核心设备的张颖和上海交通大学生命科学与生物技术学院SLSB核心设备与技术服务中心的姜浩。感谢L. Ji (MedSci)设计的原理图。基金资助:国家重点研发计划项目(2019YFA0802800: m.l., 2019yfa010802: D.L., 2019YFA0802200: C.Y., 2019YFA0802200: C.Y.),国家自然科学基金(32025023:D.L., 32230064: D.L., 31971366: L.W, 82230002: m.l., 21825701: C.Y, 91953201: C.Y., 92153303: C.Y.),上海市科学技术委员会(21CJ1402200: D.L., 20140900200: D.L.),上海市教委创新计划项目(2019-01-07-00-05-E00054,资助D.L.)、中央高校基本科研业务费专项资金(NK2022010207,资助D.L.)、药物研究国家重点实验室(SIMM2205KF-01,资助C.Y.)、华东师范大学优秀博士生学术创新能力提升项目(YBNLTS2021-026,资助L.C.)。
作者信息
作者和隶属关系
贡献
L.C.和D.L.设计了实验。L.C B.Z。广义相对论,h.m. Y.Y, M.H,部件,S.Z, H.G。mit获得,部件,r和N.X.进行了实验。L.C B.Z。广义相对论,h.m. Y.Y, M.H, D.Z,部件,布什,部件,Z.L, Y.C, Y.G,砂岩,陈守惠、地勤人员,L.W,陈守惠、马丁和D.L.分析数据。d.l.、l.c.、B.Z、g.r.、h.m.、C.Y.和G.S.在听取了所有作者的意见后撰写了手稿。D.L.监督了这项研究。
相应的作者
道德声明
相互竞争的利益
作者已根据本研究报告的结果(l.c., d.l., g.r., c.l., h.g., B.Z, J.Y, s.b., R.D.和M.L.)提交了专利申请。其余作者声明无竞争利益。
同行评审
同行评审信息
自然生物技术感谢Francisco Sanchez-Rivera和其他匿名审稿人对这项工作的同行评议做出的贡献。
额外的信息
出版商的注意施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
权利与权限
自然或其许可方(例如,社会或其他合作伙伴)根据与作者或其他权利所有人签订的出版协议,对本文享有专有权;作者对本文已接受的手稿版本的自我存档完全受此类出版协议条款和适用法律的约束。
关于本文
引用本文
陈亮,朱斌,茹,朱斌。et al。重组腺嘌呤脱氨酶TadA-8e,用于高效和特异性的基于crispr的胞嘧啶碱基编辑。生物科技Nat》(2022)。https://doi.org/10.1038/s41587-022-01532-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41587-022-01532-7
