肠道、植物根系、海洋沉积物:微生物工程师前往探索和微生物复杂系统模型。
微生物是熙熙攘攘,时空上发展,复杂的微生物群落,变量异构1。承担这种复杂性,哈里斯王说,欧文哥伦比亚大学医学中心的研究员,他的实验室的哲学是:“拥抱一些自然环境的混乱”,因为它将有助于工程技术适应这样的变化。微生物的核心作用的发现在健康和疾病已经医学,不因格贝尔说,主任在哈佛大学生物工程研究所,然而几乎所有我们知道它的功能是基于基因组和宏基因组相关性。”
最了解微生物基因的功能来自于孤立地研究微生物,但在自然界中,微生物很少独行者说特伦特北方,劳伦斯伯克利国家实验室研究员指导几个项目在美国能源部的基因组联合研究所。也许这集中导致了缺乏功能了解很多微生物的基因,这使得它紧急,他说,研究基因工程微生物在社区环境中看到,代谢物更生态相关的方法和途径。微生物工程可以帮助微生物实验室测试和完善他们的理解。工程微生物可以帮助解决条件如肠易激疾病,提供环境生物修复或提供更好的观点在生态系统微生物的作用。实验室还没有微生物设计工作室,但推进方式模型和调查微生物群落向这一目标。
从土壤到肠道,然后回来
凯利说:“她的实验室是“ecosystem-agnostic Wrighton科罗拉多州立大学。团队研究微生物生态系统土壤、破碎页岩、肠道。“我实验室试图追踪新陈代谢整个生态系统,”她说,尤其是厌氧代谢,探索约束,过程和变化由于物理空间或微生物的相互作用。这些天,实验室可以得到一个好的genome-resolved strain-resolved代谢的理解潜在的微生物群落,Wrighton说。接下来,他们探索微生物在生态系统的功能贡献找到守恒的特性和功能”不仅仅是肠道,但在这个星球上的生态系统,更全面的了解。“使用合成社区,她的小组放开的微生物生态系统复杂性,跟踪和监控,并获得时间分辨数据,她说。“理想情况下,我们可以利用这些信息,回到现场。“例如,她研究破碎页岩井地表以下2500米。释放天然气、水和化学物质注入高压下的地面。岩石破裂之前,持有小生命和水;他们是“paleopasteurized,”她说。 Fracturing introduces microbes that can form biofilms, and there can be corrosive ‘souring’ in these wells, too. She explores these effects to understand microbiome metabolism also with a view to potential remedies. The lab’s models and metabolic profiling indicate that a metabolic network sustains the microbiome at these depths2。辅酶的氨基酸。例如,甜菜碱,一种氨基酸衍生物,是丰富的页岩井骨折。一些细菌类群丰度-Halanaerobium,Geotoga和Methanohalophilus-预测这些页岩中碳和氮的可用性。科学家们重建这种页岩微生物实验室的“缩影”,约占75%的碳和氮循环的自然系统。”,对我来说,是第一次,我欣赏这些领域和实验室之间的联系,”Wrighton说。研究人员也一直在分析甲胺循环页岩,在土壤和人类的肠道。页岩的团队已经学会所教他们很多关于肠道肠道。“这不仅仅是理解代谢网络在一个系统中。“她的团队看着酶生物,微生物相互作用和竞争在不同的生态系统。
有时lab-field链接失败。在实验室团队建模soil-based微生物甲烷生产与“美”的结果,她说。他们发现甲烷生产“重量级”和寻找这些genome-resolved菌株。“他们根本不存在;他们不活跃,”她说。实验室模型帮助解决,研究因果关系和范围可能会操纵。”字段是一个不同的野兽;它给了我们真正的上下文信息,”她说。Wrighton项目涉及的合作。她的团队处理微生物学、基因组学和计算任务。 Colleagues, including the Northen lab, generate mass spectrometry, nuclear magnetic resonance spectroscopy or analytical chemical data. She uses the services of the Department of Energy’s Environmental Molecular Sciences Laboratory, a facility at the Pacific Northwest National Laboratory.
包含了微生物
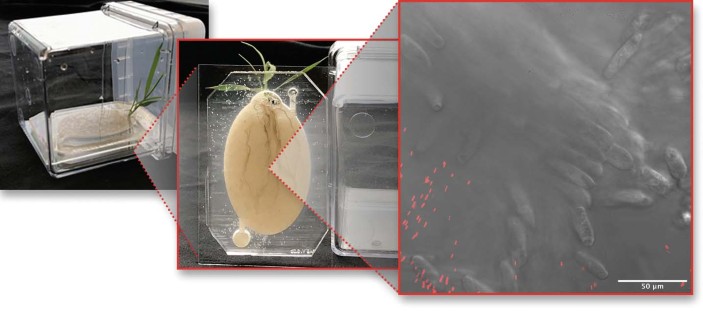
北方和他的团队构建EcoFABs,透明的微生物生态系统包含的模型。一种类型是3 d打印模具的幼苗生长水产微生物可以被添加。他们运用质谱等方法获取根微生物代谢通量和交换。系统开始作为无菌环境,确保集团是研究微生物,北方说。然后他们可以预测合成微生物代谢物交换和保持设计和测试。“这是早期,”他说,但EcoFABs design-build-test-learn迭代周期的一部分。大规模exometabolite分析微生物会给团队发展可概括的数据模型。团队计划在单细胞EcoFABs图像分辨率,使用多色荧光成像技术,构建记者结构,并应用荧光环境敏感探测定义的空间分析和描述微生物在其微环境。在开放这样的系统可以帮助解决问题,说北方,是:“做植物调节根渗出液选择有益的微生物,这种变化与环境条件吗?如果是,哪些服务选择的微生物提供植物吗? How interdependent are rhizosphere microbes?” When designing synthetic microbiomes, Northen advises carefully selecting constituent microbes. If researchers choose by phylogeny, they need to design how closely related the microbes should be. They might also choose on the basis of functional assessment of microbes. “I just don’t think we know how to think about soil microbial communities yet,” he says. The communities are so diverse, yet labs are limited in how they can study microbial activities and interactions, especially at scale. To amplify collective efforts, Northen hopes the research community can converge on a few complementary laboratory ecosystems so it becomes easier to compare systems across labs3。这是类似于生物模型达成一致”,但我们希望这个过程发生得更快,”他说。这样的努力可以通过EcoFAB指导委员会组织。
采猎者
“我们进入世界上奇怪的栖息地,那里很酷发生化学和微生物负责,”米歇尔·奥马利说加州大学圣芭芭拉分校。她主要研究厌氧生物,包括肠道微生物群,微生物群落在垃圾填埋场或海洋沉积物微生物群落在缺氧。这样一个“古怪”的栖息地是瘤胃。山羊,例如,或多或少吃东西,由于其他厌氧消化器。教训这些微生物实验室指导如何构建最小系统能够茁壮成长在这样的食物来源的多样性。他们描述微生物和微生物的相互作用,从基因组所有社区成员的重建。除了基因组组装,研究人员评估丰度和代谢活动。重建基因组指示哪些微生物吃糖,降解纤维,使短链脂肪酸或产生甲烷。有一个缺口,奥马利说,当涉及到后续问题:测试微生物如何相互作用,如何和哪种类型的信息是最好的碳循环模型。“在我看来,大多数人们与肠道微生物组现在只是猜测,”她说。 It’s tempting to see microbes as specialists rather than communities with built-in redundancy. “There’s some rhyme and reason to that, even though I as an engineer struggle with that,” she says. “But that’s not what nature does,” she says. Functional redundancy helps microbiomes recover from perturbation.

她的实验室应用基因编辑工具来控制不同的微生物群落的成员。但CRISPR-Cas同样无法部署在所有微生物,她发现。工程师的团队也开发方法的微生物环境”“造型微生物,如通过限制只有特定成员的营养或促进经济增长。奥马利的工具箱范围从1950年代微生物富集研究,媒体是用来支持增长的一些微生物在别人RNA-sequencing等新工具,基因重建、新创总成,代谢分析和建模。许多微生物实验室只关注细菌,但不是她的。她还思想偏见的危险:DNA提取可以偏见捕获基因签名,因为一些细菌方法效果更好;其他有利于真菌或古生菌。“我们会做很多优化的样本收集和准备和稳定的样品,这样我们可以看到谁在那里,得到一个真正的照片,”她说。这是一个视图,帮助团队发现,由于厌氧真菌,食草动物的最大biomass-degrading酶测序的生物4。“那之际,给我们一个惊喜,”她说。在瘤胃真菌通常被看作是病原体,或忽略了由于取样偏差。研究人员通常样本食草动物瘤胃液体,充斥着细菌。真菌液体低丰度,主要是与食物颗粒和通常在样品准备丢弃的,她说。fiber-eating灵长类动物正在进行的工作,包括黑猩猩和大猩猩,表明他们在消化道中有足够的真菌,它揭示了人类肠道。
Gut-on-a-chip
因格贝尔和他的团队建立和验证一个intestine-on-a-chip设备配备肠道上皮细胞,细胞和周围的流体流动的能力模拟肠道的蠕动运动在芯片上。研究人员最近扩大5模型实验室可以研究复杂的人类肠道微生物生活在直接接触人类肠道细胞及其上覆粘液,因格贝尔说。在人类肠道中,含氧血液流经毛细血管和梯度的降低氧气穿过tissue-tissue接口与肠上皮和腔内移。含氧介质的设备模型这一个频道,没有氧气。gut-on-a-chip可以维护一个复杂的微生物与超过200个不同类型的细菌、厌氧菌、需氧菌,对应于人类肠道微生物。在其他工作中,因格贝尔和他的团队模拟感染肠出血性敏感性的差异大肠杆菌在人和小鼠:调节在很大程度上由肠道微生物产生的代谢物。瀑样可用于研究细胞分化和细胞生物学的基本问题。因格贝尔说,他们往往是组织,而他的模型更像organs-on-chips,进入肠道和毛细管腔。瀑样球体。“出于这个原因,你没有在其中心的控制条件,和细菌生长过度,杀死人类细胞在大约一天,”他说。年前,他开始着手建立一个肠芯片为探索人类host-microbiome受控条件下的相互作用。实验具有挑战性,正常的人类肠道微生物包括许多专性厌氧菌及需氧菌:需氧菌不喜欢非常低氧,反之亦然,和非常低的氧杀死人类细胞。“所在搓…”他说。
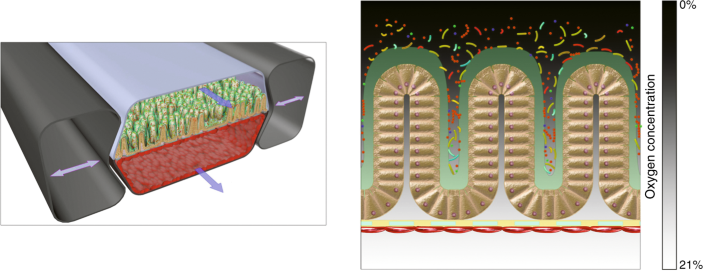
评论系统,哥伦比亚的王说,这是一种优雅的方式来研究微生物。“但是你仍然缺少一些组件,如免疫系统,”他说。,说,“我发现广泛的对这些类型的设备,但pudding-broad验收的证明将会遵循科学有效。“推进这些工作需要广泛的专业知识,如材料科学家和人民关注生态系统制造微流体。王说,与合成社区一个普遍的问题是,它们并不总是在复杂环境中保持稳定。“这些定义的系统是非常重要的在理解什么是微生物交互背后的管理规则的关键因素是什么,”他说。实验室可以使用它们来关注少数微生物相互作用;实验和定量处理。例如,系统帮助跟踪代谢物中定义的设置。“如何翻译这些类型的知识库很混乱的环境是一个开放的挑战,”他说,正如很难推断研究捕食关系一个丛林的生态系统。
进行了重新设计大肠杆菌
因格贝尔由新合成细菌感兴趣大肠杆菌紧张,说:“我们的模型可以用来推进工作。“这些都是变异的大肠杆菌叫Syn61 DNA完全在实验室合成的杰森的下巴在英国医学研究委员会分子生物学实验室6。团队重做四百万- base -对生物体的基因组通过应用他们的方法叫雷克斯(复制子切除增强基因组工程通过编程重组)。DNA是化学合成、组装成块并运送到细菌。团队的数量减少大肠杆菌遗传密码子的氨基酸从64年到61年。两个丝氨酸密码子和一个终止密码子被换出18000的基因位点。这是一个方法,可以帮助构建生物和合成生物学家是不易基因编辑来完成。当研究人员本杰明钝和汤姆·埃利斯在新闻和观点陪同下巴的纸,这合成和减少的大肠杆菌的遗传密码代表“新纪录”,具有类似项目。“基因组最小化和减少密码子是第一个使用这种新技术,这可能有一天给我们功能重组基因组和基因组定制设计直接细胞执行特殊任务,”他们说。下巴解释说,除了这种方式的工程单基因组,实验室可以使用这种方法来综合发展设计师微生物。“这可能组装微生物组成的有机体,使用不同的压缩基因编码一个路线限制微生物有机体之间的水平基因转移,”他说。
思维的进化
在微生物研究方面,麻省理工学院研究员塔米利伯曼应用进化推理。她通过预测评估细菌的个性化的自然历史积累的微生物突变成过去。获得适应性突变基因和通路给”的细菌生存最紧迫的挑战是什么,”她说,并指导如何可能支持或削弱这些挑战。鉴于低频变异难以获取与短内容测序和组装,实验室文化的特定菌株单独有氧和厌氧细菌。文化避免微生物竞争但反映体内的多样性。团队的基因组序列很多独立的殖民地在高吞吐量。细菌可能从一个肿瘤,从粪便样本或皮肤拭子。例如,利伯曼实验室看的进化过程金黄色葡萄球菌在患有湿疹。细菌可能不是驾驶条件,但降低葡萄球菌含量可能会帮助病人。微生物种群的个体倾向于保持稳定,除了有一个大扰动时,如使用抗生素,她说。尽管大量暴露于外部压力,居民的似乎击败他们。但是其他的课程都是可能的。实验室的血统追踪脆弱拟杆菌健康人的内脏了18个月。“我们看到的是,实际上有什么看起来像一个功能多样化两株共存的物种,”她说。

评论更普遍的是,奥马利说,微生物工程师应该包括进化方面的考虑在他们的工作。在微生物群落中,成员可以突然偏离非常相似。她和她的团队工作的触发事件,这可能包括水平基因转移,可以借微生物竞争,适应的优势。“如果你能找出方法来利用工程,这将是非常酷的,”她说。沿着这些线路发生在王实验室工作。
Mobilome移动
实验室可以配置文件中的微生物复杂的方式但缺乏工具来控制微生物,王说。他和他的团队构建可归纳的方法在不同的微生物,希望让他们“可概括的人口规模的工程技术。“一个平台利用移动元素:宏基因组改变肠道微生物原位接合(魔术)7。一个大肠杆菌供体菌株改造与设计基因有效载荷是通过移动元素。魔术是一个工具箱的开始混乱的体内环境,他说。“我们使用肠道,但它可能是皮肤,可以是任何类型的微生物,”卡洛塔说朗达,在王的实验室博士后;她采访了与他联合。魔法可以用来deconvolve系统的组件和理解复杂的系统。团队利用细菌与彼此分享资料的习惯,如交换通过接合质粒或遗传元素。王说,这很好理解,但不是它的范围。基因组分析揭示了基因从何而来,这使得分析一种“基因组考古学。“共享元素保留只有当他们传达优势,王说。例如,从海洋细菌基因转移到人的微生物群解释了为什么有些人在日本,否则会消化消化紫菜多糖。基因转移可以罕见,仍然重要,特别是当它涉及到抗菌素耐药性基因。 By tracking networks of transfer events, the team hopes to find ways to functionally activate desirable elements in “friendly recipients,” he says.
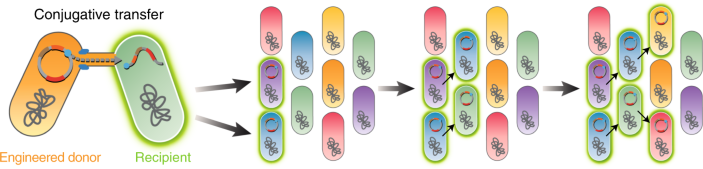
实验室继续优化魔法,朗达说。向量需要导致稳定、可重复编程的功能。在这种方法中,实验室需要仔细选择质粒,考虑到甲基化和监管元素可能会影响水平基因转移,这样他们就可以优化结构的特定目的,她说。“在某种程度上这些移动遗传元素是自私的元素,”王说。他们寻求尽可能多的生物繁殖。合成应用,“如果你可以劫持,这是一个方法植入新功能不依赖只是一个工具。”,更有可能的元素将被保留在整个人口。小王和他的实验室建立了人工生态系统中重要metabolites-amino酸是“交易”。可以定量模型与交易相关的能源成本,他说,通过研究与bio-economic集成模型和所谓的微生物贸易理论。但它是具有挑战性的规模大,复杂的社区空间异质性,颞可变性和环境波动。工程微生物方法设计的系统都是健壮的和稳定的,他说。 When they’re not, he and his team head back to the drawing board. Labs are only just tapping into nature’s metabolic capacity, says Ronda, which might lead to new approaches to fermentation and sustainable production processes.
另外,王实验室已经描述的速度水平基因转移在自然环境中8。他们通过CRISPR-Cas垫片收购过程中,充当DNA记录器,王说。记录的应变时暴露于微生物样本,碎片入侵元素捕获和集成的“逆电流器”CRISPR数组。这些间隔器保护:转录时,细菌的免疫系统的一部分。逆电流器可以测序确定转移元素。这允许一个工程师的情况,王说,应变的录音功能是放置在一个复杂的环境中,它将记录数组的DNA狂轰乱炸。这个记录的微生物实验室积极分享在这个复杂的环境中可以帮助,例如,找到新的类型的向量。记录压力可能是一个专用的基因转移系统能够跟踪在不同生物的基因去哪里。
比较、交流、合作
它可以是一个难题:复杂的微生物系统很难模型,但没有一个模型,理解是难以实现的。在北方的观点,“我们猜测在社区”包括遍历的周期,测试,识别缺陷,思考改进,建立一个新的社区,然后重复循环。”研究的多组相同的系统和专业知识的多样性越大,越快我们的理解会进步,”他说。模型建造者们需要明确他们的目标和他们的系统可以和不能做什么。“我们正在建造“模型”的生态系统,而不是自然生态系统,“北方说。模型必须提供控制和再现性因此,科学家们可以有效地确定因果机制,他们可以推出概念和计算模型。然后模型的有效性和普遍性必须建立于自然生态系统。
“天然”系统可以基准相比工程微生物实验室之间。如何最好地踢轮胎是一个模型?“容易,谁更好的模拟人体生理学和临床结果获胜,“因格贝尔说。有很多向自然学习,奥马利说。“工程师们倾向于过度简化任何系统,这可能是微生物工程对我们工作,”她说。“我们需要思考很多比我们做的更复杂。“承担复杂微生物,微生物人员接受合作。“实验室研究不同微生物组使用不同方法,朗达说,但有“共同点。“微生物工程方法需要可概括的,这样他们就可以被应用在土壤微生物是否,植物的根或肠道。“有很多不同领域之间的交流”她说。 In her collaborations, Wrighton finds shared patterns, shared questions, shared resources. “The field,” she says, “is really open to new perspectives right now.”
引用
贝母,m . et al。科学353年,1147 - 1151 (2016)。
现在,m . a . et al。Proc。国家的。学会科学。美国115年E6585-E6594 (2018)。
Zengler, k . et al。Nat方法。https://doi.org/10.1038/s41592 - 019 - 0465 - 0(2019)。
所罗门,k v . et al。科学351年,1192 - 1195 (2016)。
Jalili-Firoozinezhad, s . et al。Nat,生物医学。Eng。https://doi.org/10.1038/s41551 - 019 - 0397 - 0(2019)。
Fredens, j . et al。自然569年,514 - 518 (2019)。
朗达,c . et al。Nat方法。2,167 - 170 (2019)。
Munck, c . et al .预印本https://www.biorxiv.org/content/10.1101/492751v1(2019)。
作者信息
作者和联系
相应的作者
权利和权限
关于这篇文章
引用这篇文章
马克思,诉工程师接受微生物混乱。Nat方法16,581 - 584 (2019)。https://doi.org/10.1038/s41592 - 019 - 0460 - 5
发表:
发行日期:
DOI:https://doi.org/10.1038/s41592 - 019 - 0460 - 5
